论文地址:ProteinCrow:A Language Model Agent That Can Design Proteins
论文实现:<无>
ProteinCrow:Agent+BindCraft设计蛋白
Abstract
ProteinCrow 是一个基于大型语言模型(LLM)的代理(agentic)蛋白质设计助手 。近期,深度学习领域的突破(如AlphaFold, RFdiffusion, BindCraft和ProteinMPNN等工具)彻底改变了蛋白质结构和序列建模,使得设计具有新功能的蛋白质成为可能 。成功的计算机蛋白质工程流程通常需要整合多个专用模型,并结合生物化学知识进行迭代优化 。
ProteinCrow正是在此背景下被提出的。它能够整合多模态信息,包括结构数据、深度学习模型、科学文献以及用自然语言表达的生物化学数据 。通过使用36个专家策划的工具,ProteinCrow可以自动化地执行蛋白质设计任务 。
为了评估其性能,研究人员在以下三个任务中对ProteinCrow进行了测试:
- 设计针对特定二级结构基序(secondary-structure motifs)的结合蛋白库(binder libraries) 。
- 重新设计蛋白质骨架以提高其稳定性 。
- 优化结合蛋白以消除预测的主要组织相容性复合体I类(MHC Class I)抗原表位 。
1 Introduction

计算蛋白质设计已成为加速创造具有新功能蛋白质的重要工具,其应用范围从工程化高亲和力的治疗性结合蛋白到优化酶的活性和稳定性 。传统方法依赖于基于物理学的模型,如Rosetta,来评估蛋白质结构 。
近年来,深度学习方法——例如AlphaFold2 (AF2)、RFDiffusion和ProteinMPNN——通过实现精确的结构预测和序列优化,已经彻底改变了这一领域 。然而,没有任何单一方法能完全捕捉序列、结构和功能之间的复杂相互作用 。
本文的假设是:大型语言模型(LLM)能够将深度学习模型的输出、生物化学数据、科学文献以及基于物理学的方法统一为自然语言,从而可以作为一个框架来整合关于蛋白质的多模态信息,并促进有效的蛋白质设计 。
在这项工作中,作者们提出了ProteinCrow,这是一个LLM代理(agent),它通过整合多种工具,能够根据自然语言提示(prompts)生成满足特定约束条件的蛋白质序列库 。ProteinCrow还通过PaperQA(一个为检索和总结科学文献而优化的代理)来整合基于文献的见解,这模仿了专家查阅特定蛋白质家族或任务相关知识的方式 。ProteinCrow可以执行目前由人类完成的子任务,例如将大蛋白质裁剪成小区域、识别结合的“热点”残基,以及使用计算机模拟指标分析设计出的序列 。通过为该代理提供人类计算蛋白质工程师所使用的深度学习方法、数据库查询工具和基于物理学的方法(如Rosetta),ProteinCrow能够产出具有优化稳定性的设计,并生成用于实验验证的蛋白质序列库 。
尽管近期有一些将LLM融入蛋白质设计流程的尝试,但它们在自主性(autonomy)和范围上有所不同 。例如,310.ai提供了一个自然语言驱动的聊天界面,但它并非自主代理 。相比之下,ProteinForceGPT是一个自主LLM代理,但其应用范围更特定,用于从预训练结构中预测力-分离曲线 。
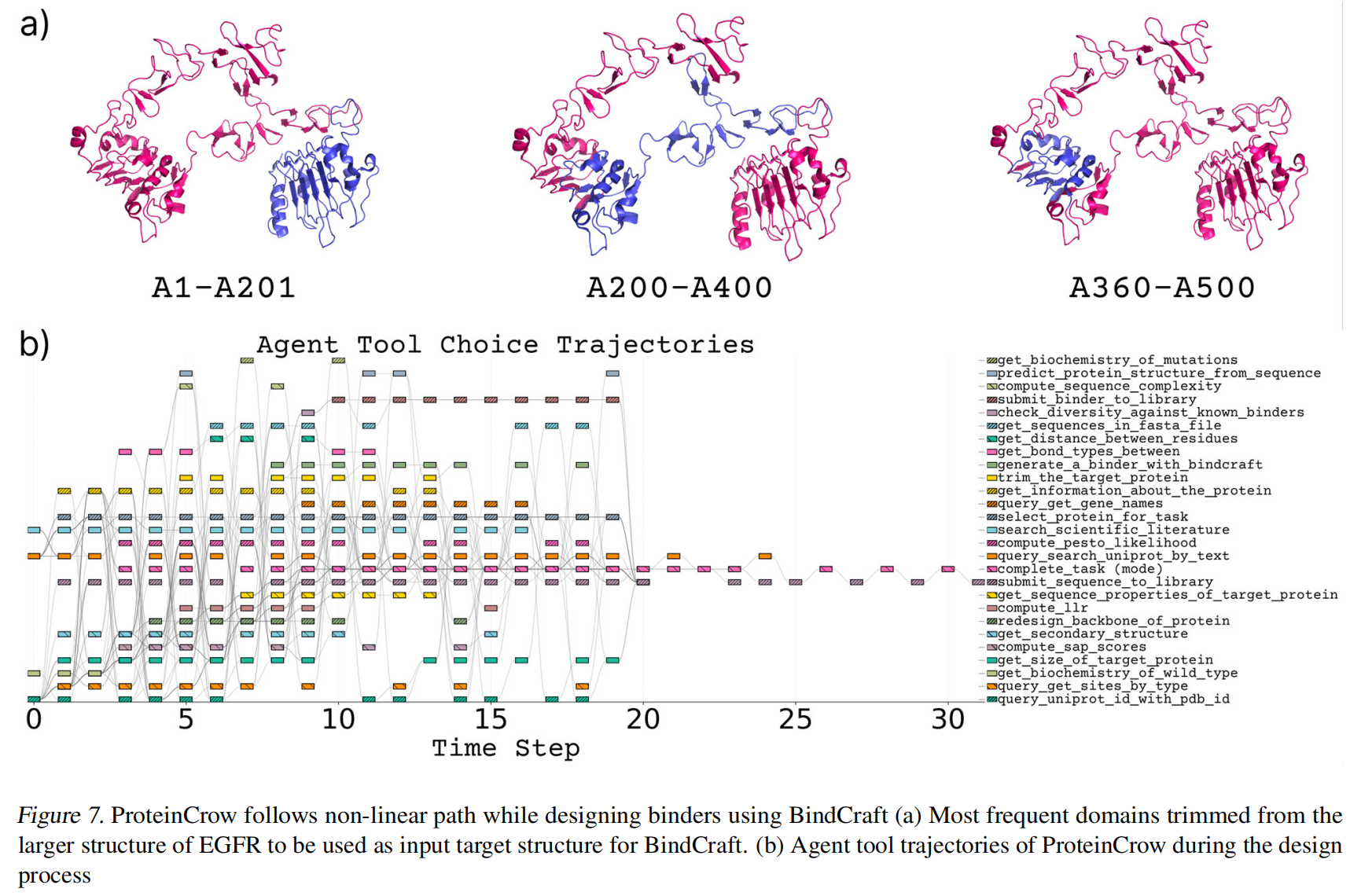
尽管大多数蛋白质设计代理是专用或范围有限的,但在化学合成、材料研究和实验设计等其他领域,已经探索了更通用的LLM代理 。ProteinCrow是使用Aviary框架构建的LLM代理,旨在促进通用的蛋白质设计 。Aviary是一个用于在复杂任务上构建和训练LLM代理的框架 。一个LLM代理是一个决策实体,能够观察环境、进行推理并执行一个动作 。对于ProteinCrow而言,“动作”就是一次工具调用(tool call),用于蛋白质结构/序列分析、生成式设计或库设计 。该代理在一个循环中持续运行,根据新的观察或工具输出来决定下一步行动,直到达到预期目标 。
作者们在以下三个任务上对ProteinCrow进行了评估 :
- 任务1:目标导向的蛋白质设计 - 在结构和功能约束下提高目标序列的稳定性 。
- 任务2:自动化的结合蛋白设计流程 - 生成在库组成、结合蛋白结构和序列属性上具有约束的结合蛋白库 。
- 任务3:优化结合蛋白 - 通过重新设计一个先前设计好的结合蛋白来生成一个序列库,以消除预测的MHC I类抗原表位 。
2 Methods
2.1. LLM Framework - Aviary
ProteinCrow 在 Aviary 框架内运行,该框架是一个可扩展的语言代理gymnasium 。Aviary 将代理的序贯决策过程构建为一个基于语言的、部分可观察的马尔可夫决策过程(POMDP),这使得代理能够迭代地优化蛋白质序列 。为 ProteinCrow 构建的工具也可以转移到 Aviary 之外的其他代理框架中 。
ProteinCrow 通过调用工具并接收观察结果(observations)来为下一步行动提供信息,从而优化蛋白质稳定性、设计符合特定约束的结合蛋白库,以及优化已知的结合蛋白 。所有实验均使用 Claude-3.5-Sonnet-20241022 模型,采样温度设置为0.1 。
2.2 Tools
ProteinCrow 可以使用多种工具,大致分为以下几类 :
- 生化描述符 (Biochemical Descriptors): 用于生化分析的工具,包括界面表征、键类型分析、序列复杂性、残基疏水性和二级结构注释 。
- 深度学习模型 (Deep Learning Models): 用于各种蛋白质设计任务的深度学习模型,包括结合蛋白设计、反向设计(inverse design)、骨架生成和蛋白质语言模型 。
- Rosetta协议 (Rosetta Protocols): 执行Rosetta协议的工具,广泛用于蛋白质结构建模和蛋白质-蛋白质界面分析 。
- 任务管理 (Task Management): 用于管理与提示相关的各种任务,例如向库中提交序列和跟踪任务完成情况 。
- 序列信息学 (Sequence Informatics): 用于基于序列的分析,包括残基属性、多序列比对和识别保守位点 。
- 知识检索 (Knowledge Retrieval): 用于从数据库和文献中查询和检索相关信息 。
为了解决各种深度学习模型之间频繁出现的版本冲突和兼容性问题,ProteinCrow 中的大多数工具都通过 Modal 在API端点上运行,这使得 AlphaFold、ProteinMPNN、ESM2 和 BindCraft 等多个模型能够在沙盒环境中执行 。
2.3 Task 1 - Goal Directed Protein Design
2.3.1 Redesign Protein Backbones for Improved Stability with Structural Constrains
目标导向的蛋白质工程涉及对目标蛋白进行突变以实现预期目标,例如提高稳定性 。在此任务中,ProteinCrow 被要求在重新设计蛋白质骨架时,不仅要提高其稳定性,还要增加盐桥的数量 。ProteinCrow 可以通过调整 ProteinMPNN 中氨基酸类型的偏置权重(bias weights)来偏向于特定的残基 。盐桥是通过计算突变后所有酸性(Asp, Glu)和碱性(Arg, Lys, His)原子对之间距离小于4.0 Å的数量来识别的 。
为了评估性能,研究人员从一个大规模稳定性数据集中随机选择了30个蛋白质(长度为40-72个氨基酸) 。突变引起的自由能变化(ΔΔG)使用 Rosetta cartddG 协议计算 。此外,还计算了以下指标进行综合评估:
- Rosetta ΔΔG 小于0的突变百分比
- 氨基酸使用频率
- 序列多样性
- 使用ESM2计算的伪对数似然值(pseudo log-likelihood)
- 来自AlphaFold2的pLDDT置信度分数
2.3.2 Redesign Enzyme Backbones While Preserving Functional Residues
为了测试 ProteinCrow 在增强蛋白质稳定性同时不损害其功能的能力,研究人员使用了先前研究中的两种蛋白质:肌红蛋白(Myoglobin)和TEV蛋白酶(TEV protease) 。研究人员将 ProteinCrow 选择的突变位点与原始研究中人类设计师为保留功能而选择保留的位点进行比较,以评估 ProteinCrow 是否能避免在这些功能性位点上提出突变 。
评估指标中增加了一项“功能位点保留”(Functional site preservation),即计算 ProteinCrow 选择的突变位点与先前研究中功能位点的重合次数 。
3. Task 2 - Automated Binder Design Pipeline
文中首先介绍了BindCraft,这是一个用于de novo(从头)蛋白质结合蛋白设计的高效深度学习模型,其报道的实验成功率在10%到100%之间 。BindCraft的设计流程包含多个需要根据不同蛋白质靶点进行优化的设置,例如迭代次数、设计权重和筛选标准,这些通常由人类专家来选择 。
3.1 Designing Binders for Epidermal Growth Factor Receptor (EGFR)
为了在一个具有挑战性的真实世界靶点上评估ProteinCrow,研究人员选择了表皮生长因子受体EGFR蛋白(PDB ID: 6ARU) 。该靶点曾在一个由AdaptyvBio主办的全社区范围的结合蛋白设计竞赛中使用过 。ProteinCrow的设计质量采用了与AdaptyvBio竞赛相同的评估指标,以便与那些经过实验验证的顶级参赛作品进行比较 。
ProteinCrow在多种约束条件下接受了评估,具体实验包括:
- 自动化的端到端结合蛋白生成:要求ProteinCrow为EGFR生成一个结合蛋白,并进行10次重复实验 。
- 靶向结构基序设计:竞赛中表现最好的结合蛋白具有显著的β-折叠(β-sheet)元件 。为了测试ProteinCrow是否能优化BindCraft的设计参数以生成特定结构基序,研究人员要求它设计一个包含β-折叠元件的结合蛋白 。
- 约束感知的结合蛋白库设计:要求ProteinCrow生成一个包含5个结合蛋白的库,并满足三个约束条件 :
- 每个新的结合蛋白必须与已知的结合蛋白至少有10个点突变的差异 。
- 生成的库在二级结构组成上应表现出多样性 。
- 针对蛋白质上不同的“热点”残基进行设计 。
3.2 Generalizing ProteinCrow to Distinct Protein Targets
该部分首先总结道,结果表明ProteinCrow能够通过为BindCraft选择合适的输入,在没有人工干预的情况下,为多样的蛋白质靶点和库约束条件自动化并执行BindCraft设计流程 。
每个结合蛋白库中序列的性能通过以下广泛使用的计算机模拟(in-silico)指标进行评估:
- AlphaFold分数:使用AlphaFold-Multimer模型产生的pLDDT、iPAE和iPTM分数,来评估设计出的结合蛋白序列的结构质量、对齐准确性和预测的转换矩阵 。
- ESM伪对数似然分数:使用蛋白质语言模型ESM2来评估生成序列的似然度(likelihood) 。
- 二级结构组成:分析结合蛋白的二级结构注释,以确保它们包含设计约束在提示中要求的结构元件 。
4. Task 3 - Optimizing a Binder to Reduce MHC Class I Epitopes
在治疗性结合蛋白的设计中,一个关键步骤是优化其特性,例如稳定性、聚集倾向和免疫原性(immunogenicity),同时还要保持对靶点的亲和力 。
为了评估ProteinCrow执行此类优化任务的能力,研究人员从一个经过实验验证的EGFR结合蛋白(来自Adaptivbio社区结合蛋白设计竞赛)开始 。他们要求ProteinCrow生成一个包含10个变体序列的库,并进行5次重复实验,目标是消除预测出的MHC I类抗原表位 。
- 预测工具:研究中使用了 netMHCpan 4.1 来预测所有8-14个氨基酸长度的肽段与
HLA-A*02:01等位基因的结合亲和力 。 - 评估指标:最后,研究人员通过两个指标对设计出的序列进行排序和比较:
- ESM2伪对数似然分数 。
- 预测出的MHC I类结合蛋白的总数(包括强结合和弱结合的) 。
5 Results
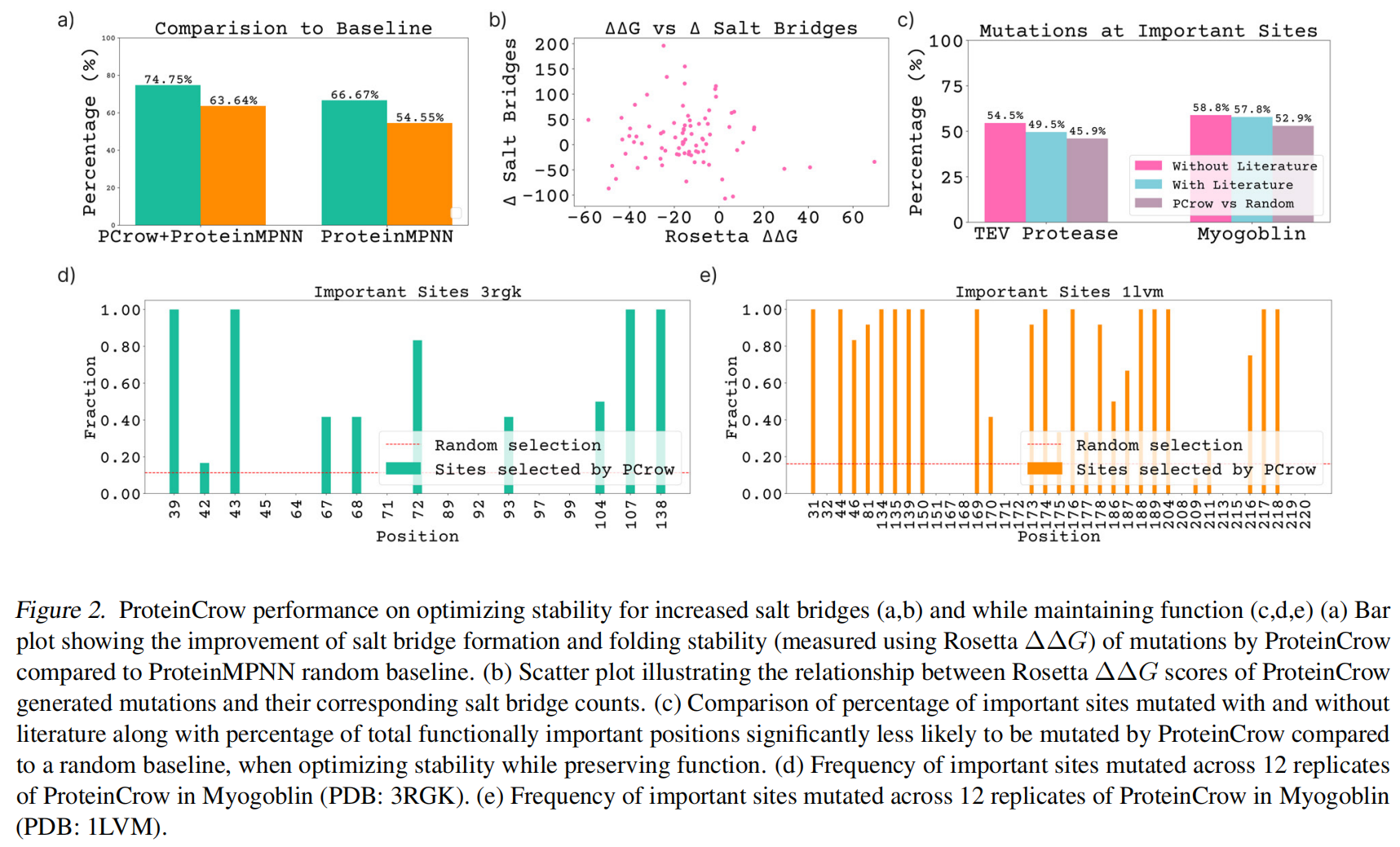
5.1 Optimizing Protein Stability while improving number of salt bridges
这项任务旨在评估ProteinCrow执行目标导向蛋白质设计的能力 。研究人员要求它提出既能提高折叠稳定性(通过Rosetta ΔΔG<0 衡量)又能引入更多盐桥的突变 。ProteinCrow综合使用了基于结构、序列、文献、深度学习和Rosetta的工具来分析每个目标蛋白并生成设计方案 。
- 结果:在30个基准蛋白质上,ProteinCrow在许多情况下成功地找到了同时满足这两个标准的突变 。
- 与基线比较:相比之下,基线模型ProteinMPNN虽然能有效生成低$\Delta\Delta G$的变体,但本身并不会增加盐桥的数量 。
- 结论:这些结果表明,ProteinCrow有潜力处理那些通常需要专家指导和迭代才能解决的、更细致的设计目标 。
5.2. Designing stable mutations while preserving function
这项任务评估了ProteinCrow识别功能性残基并避免对其进行突变的能力 。
- 结果:与随机基线相比,对于肌红蛋白和TEV蛋白酶,ProteinCrow在超过45%的重要位点上提出的突变显著减少 。一项消融研究(ablation study)显示,在重新设计骨架时,引入文献检索可以进一步减少ProteinCrow选择功能性重要位点的数量 。
- 讨论:作者认为,尽管该代理成功地捕捉了一些专家知识,但它可能仍会忽略其他关键残基,这可能是由于其在解释功能和结构信息方面存在局限性 。不过,其模块化框架为通过训练或整合更多工具来进行改进提供了可能 。
5.3. Designing Binders for Epidermal Growth Factor Receptor (EGFR)
- 自动化的端到端结合蛋白生成:在10次重复实验中,ProteinCrow每次都成功地为EGFR生成了结合蛋白 。它会自动检索蛋白质结构,进行结构分析,识别关键结合热点,执行BindCraft工作流程,并构建一个潜在的结合蛋白序列库 。

- 靶向结构基序设计:当被要求设计包含β-折叠元件(一种在竞赛顶级结合蛋白中出现的结构)的结合蛋白时,ProteinCrow通过调整BindCraft流程中的设计权重,成功设计并添加了一个具有显著β-折叠区域的结合蛋白到库中 。
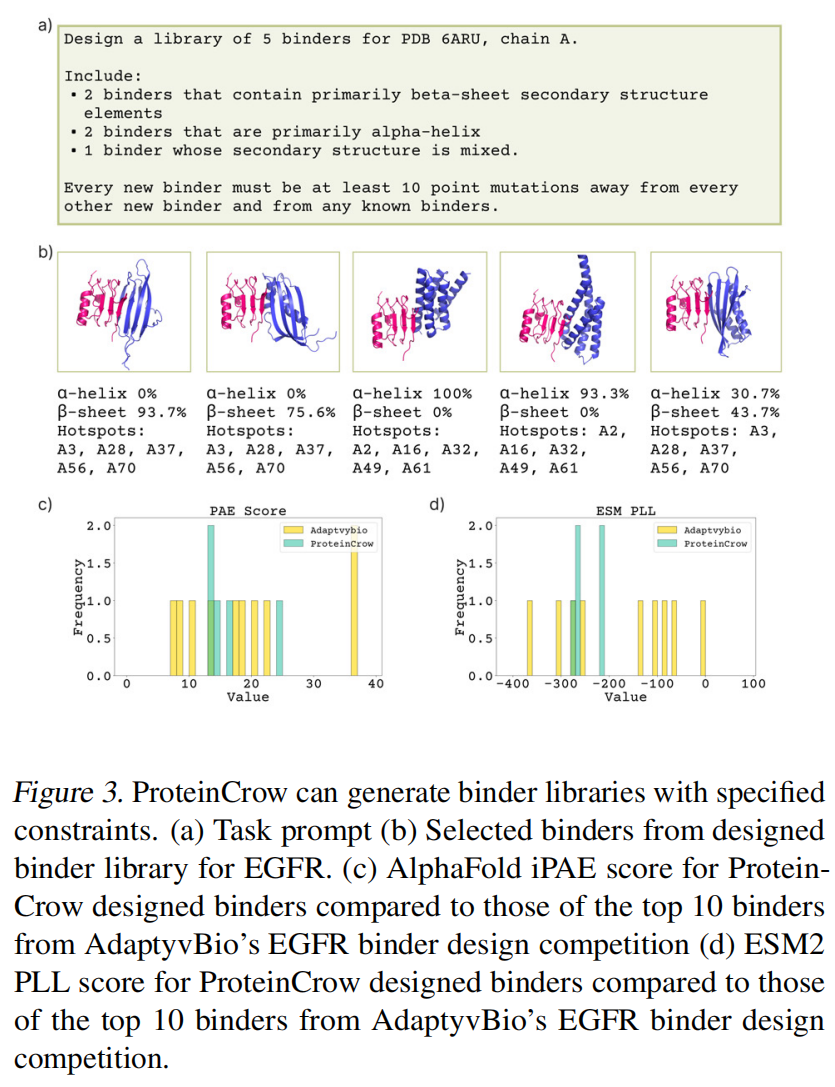
- 约束感知的结合蛋白库设计:ProteinCrow成功地生成了一个包含5个结合蛋白的库,同时满足了三个复杂的约束条件:1)每个结合蛋白与已知序列有足够差异;2)库的二级结构组成多样化;3)利用了不同的靶点热点残基 。
5.4. Generalizing ProteinCrow to Distinct Protein Targets
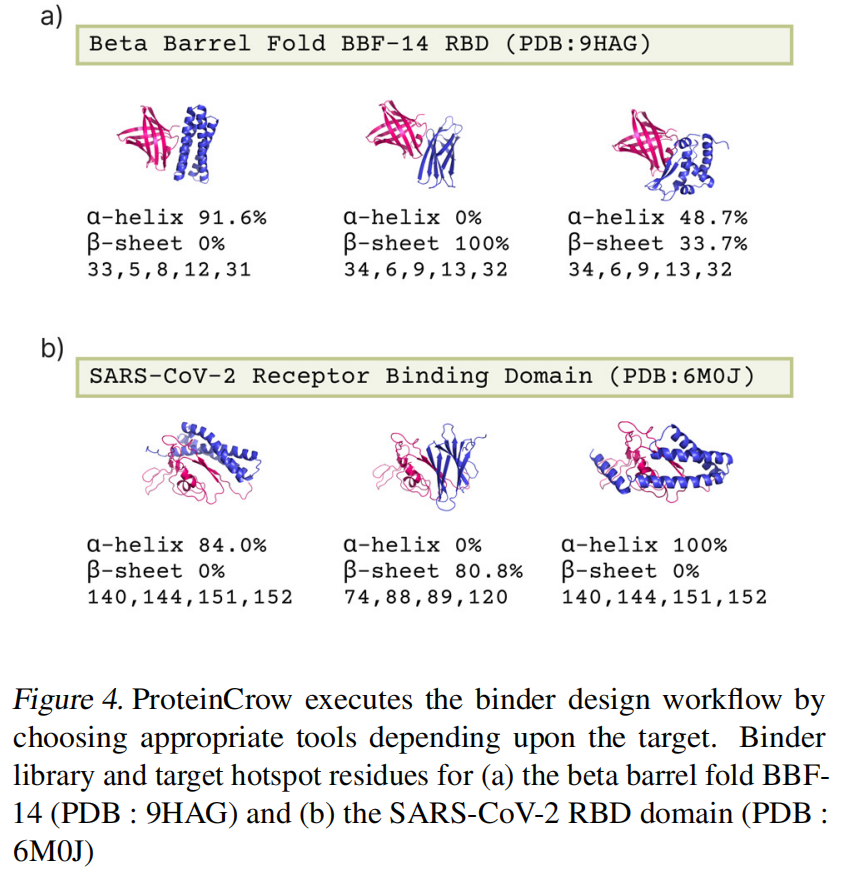
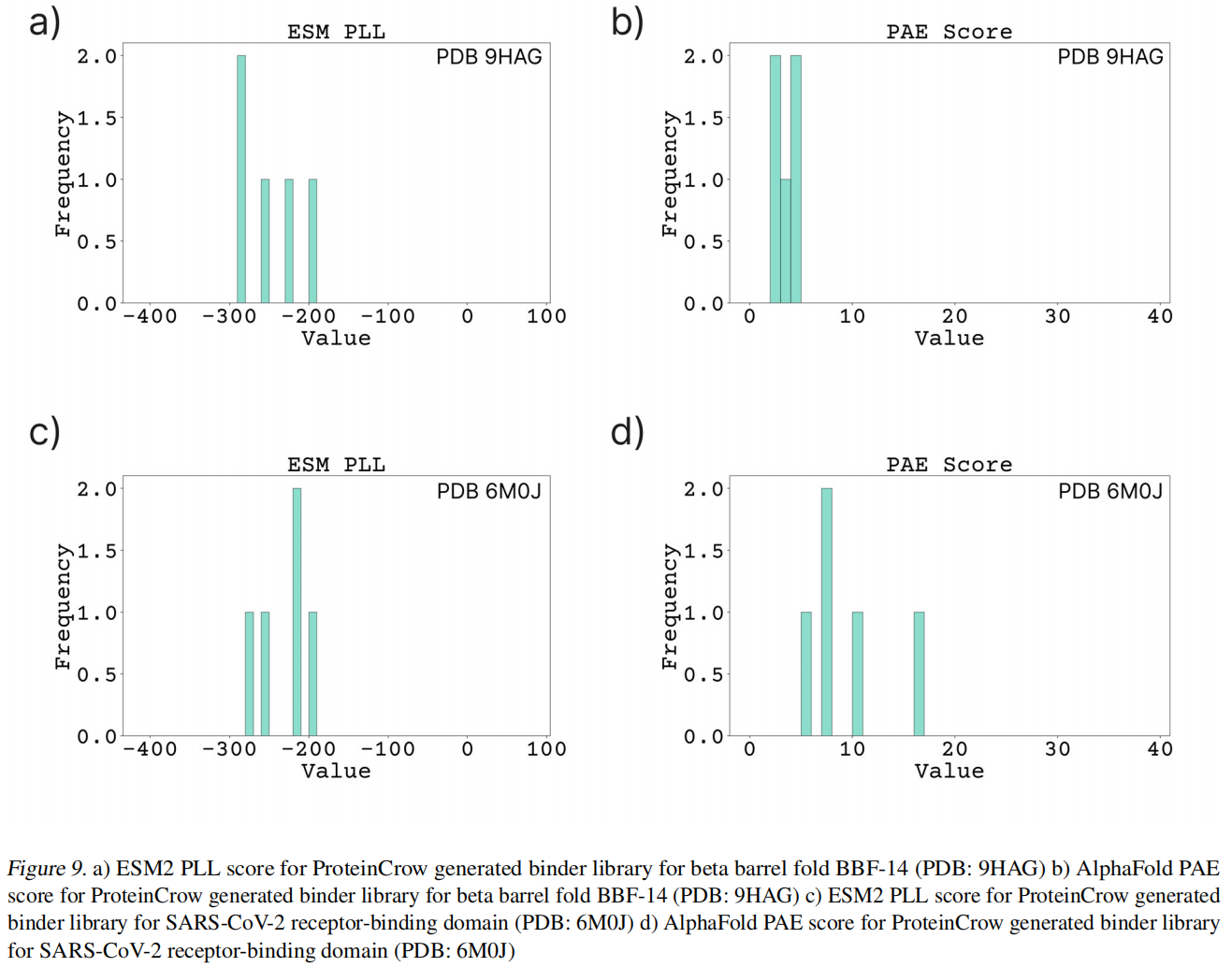
ProteinCrow成功地为SARS-CoV-2受体结合域(RBD)和β-桶折叠BBF-14这两个截然不同的靶点生成了包含5个结合蛋白序列的库,并且遵循了所有必需的库约束条件 。这些结果表明,ProteinCrow的设计流程可以泛化到多种多样的蛋白质靶点上 。
5.5. Reducing MHC Class I Epitopes in a Binder
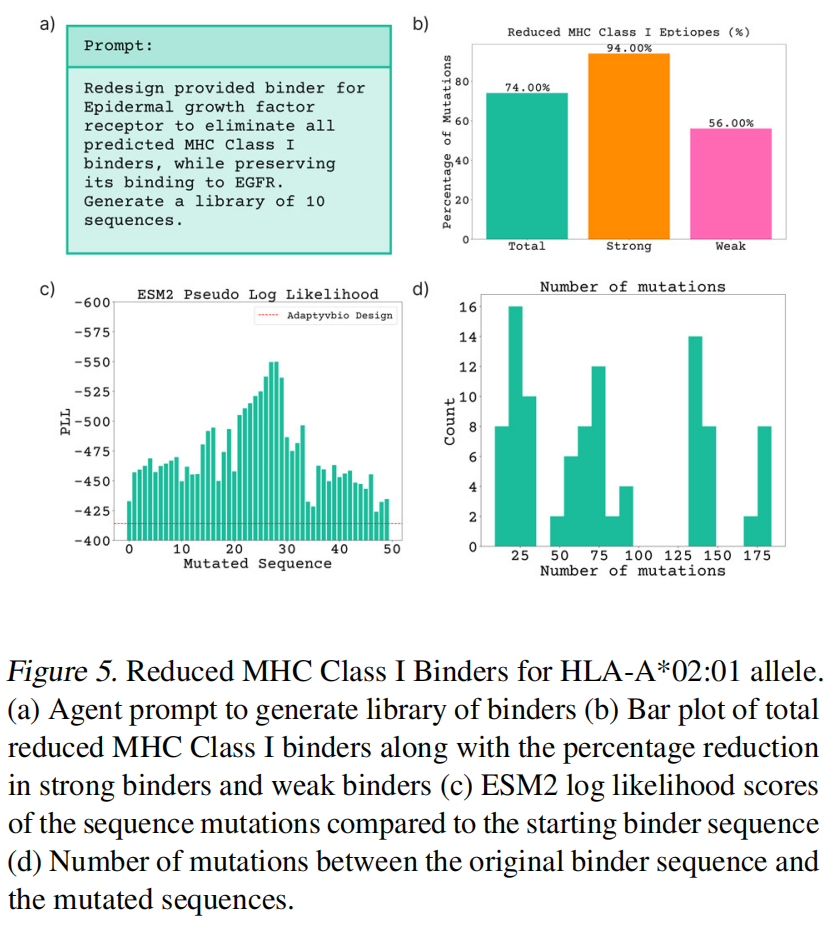
在这个任务中,ProteinCrow被赋予了使用netMHCpan工具的能力,用于预测一个已知EGFR结合蛋白中的MHC I类结合表位 。
- 结果:在总共50个设计中,74%的提议突变降低了预测的MHC I类结合表位的数量 。
- 额外发现:此外,所有50个经过优化的序列的ESM2伪对数似然分数都比原始结合蛋白更高 。
6. Discussion
用于结构预测、反向设计和蛋白质语言模型的深度学习方法已经改变了蛋白质工程领域,使得新型蛋白质的快速生成成为可能 。人类蛋白质工程师通常会利用专家知识,将这些关于蛋白质结构和序列的基础模型结合起来,以构建有效的设计流程 。
ProteinCrow整合了蛋白质设计界常用于构建这些流程的工具 。它是一个用于蛋白质设计的自主LLM代理(Agent),在这项工作中被应用于三个常见的蛋白质工程任务:蛋白质稳定性优化、结合蛋白设计和结合蛋白优化 。
研究表明,ProteinCrow能够遵循复杂的、非线性的工作流程 。这一点对于构建一个通用代理至关重要,因为蛋白质设计是高度依赖于特定靶点和任务的,并且可以通过多种多样的工具来实现预期结果 。此外,ProteinCrow环境的模块化特性使得新工具的集成变得十分便捷 。
作者指出,本研究仅限于使用计算机模拟(in-silico)的评估标准来展示LLM代理完成多样化蛋白质工程任务的能力 。在未来的工作中,他们期望在实验室中评估ProteinCrow的成功率,将计算机模拟的预测转化为实验验证,并探索如何整合反馈来训练和增强该代理 。
