论文地址:Evaluating Protein Transfer Learning with TAPE
TAPE:评估蛋白质embedding的任务
Abstract
包含了五个生物学相关的半监督学习任务来评价蛋白质嵌入Tasks Assessing Protein Embeddings (TAPE)。该任务包括蛋白质结构,进化和蛋白质工程的相关预测,同时表示自监督学习模型在生物领域还有更大的潜力
Background
- 蛋白质表示:将长度L的蛋白质x用25个字母组成的氨基酸序列表示:(x1, x2, ……, xL)。其中25个字母为20个标准氨基酸,2个非标准氨基酸,2个模糊氨基酸和1个未知氨基酸
- 蛋白质3D结构:初级结构(氨基酸序列)二级结构(局部特征)三级结构(整体特征)
- 同系物:两种具有相同祖先的蛋白质,但由于在很远的过去就开始分支,所以可能在序列上有很大的不同
- 在本文中,作者主要应用sequence identity(氨基酸序列比对)来定量进化关系
Dataset
预训练语料库:大量未标记序列数据集,Pfam (31M 蛋白质结构域数据库)
监督数据集:每个任务都不同,在 8000 到 50000 个训练示例之间变化
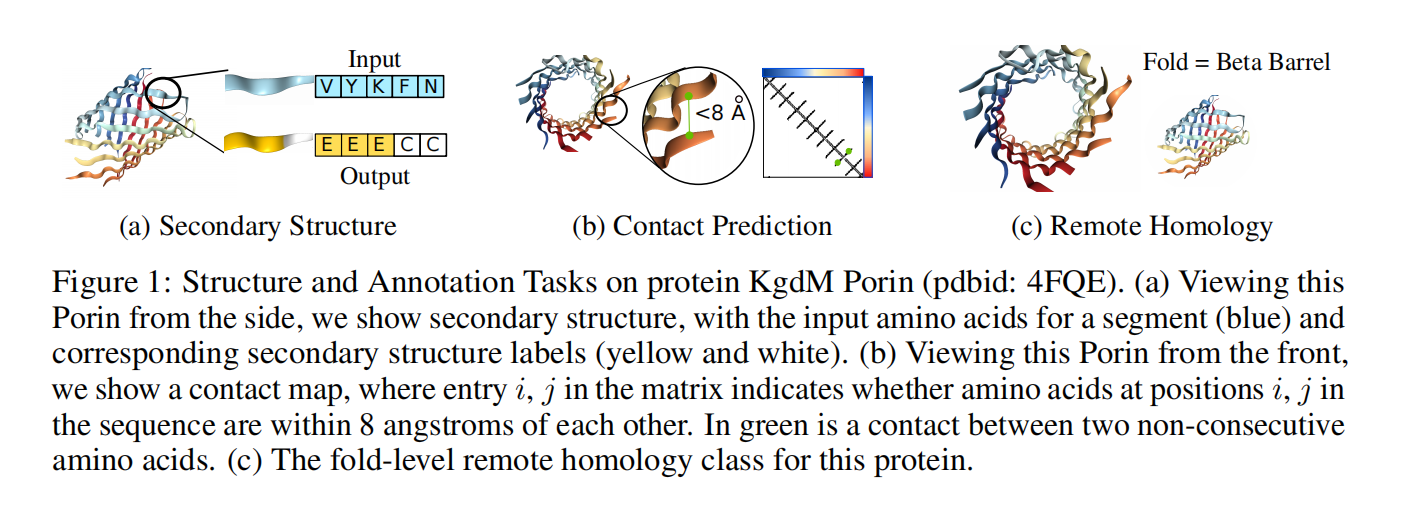
Task 1: Secondary Structure Prediction
图1a
定义:Seq2Seq任务,每个输入的氨基酸xi对映一个标签 $y_i∈{Helix(H), Strand(E), Other©}$
影响:二级结构是了解蛋白质结构的重要特征;二级结构预测工具也常用来为高级模型提供更丰富的输入特征。
泛化:检测模型学习局部结构的水平
Task 2: Contract Prediction
图1b
定义:成对氨基酸的预测任务,来自蛋白质的氨基酸 $x_i$ 和 $x_j$ 被映射为标签 $y_{ij}∈ {0, 1}$,代表他们是否“接触”
影响:提供了强大的全局信息,完整3D蛋白质结构的鲁棒建模
泛化:考察模型对整体蛋白质环境的理解
Task 3: Remote Homology Detection
图1c
定义:序列分类任务,每个序列x对应一个标签 $y∈ {1,…, 1195}$,代表蛋白质可能的不同折叠
影响:可以应用于微生物学和医学,例如检测新出现的抗生素抗性基因
泛化:检测模型在远距离相关输入上识别结构相似性的能力
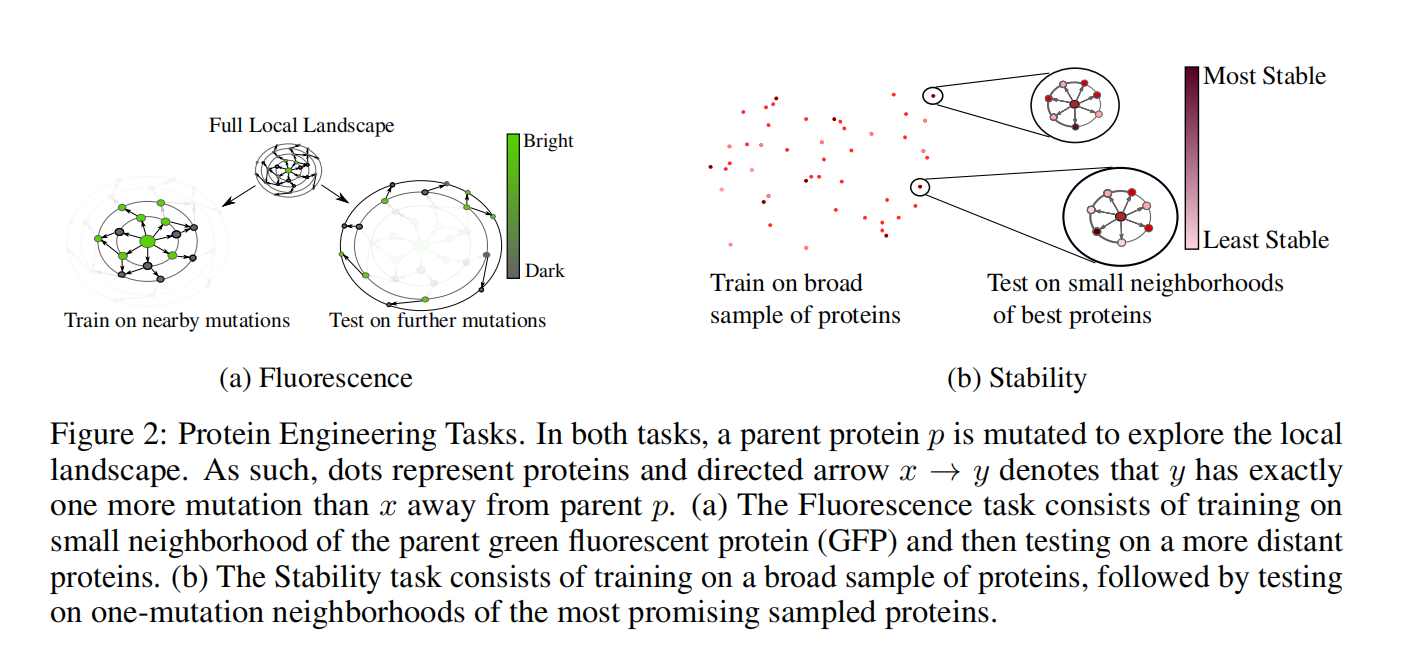
Task 4: Fluorescence Landscape Prediction (Protein Engineering Task)
图2a
定义:回归任务,每个输入蛋白质x对应一个标签 $y∈ R$,反应x的对数-荧光强度
影响:更有效地探索landscape
泛化:测试模型判断极为相似输入,以及概括未知突变组合的能力
Task 5: Stability Landscape Prediction
图2b
定义:回归任务,每个输入蛋白质x对应一个标签 $y∈ R$,代表折叠的稳定性
影响:寻找对昂贵的蛋白质工程实验的最佳候选者更好的改进
泛化:测试模型从广泛的序列样本中进行概括,以及在几个序列的邻域中定位信息的能力
Model and Experimental Setup
模型:
作者评估了三个(受NLP启发)模型:Transformer, Residual network和LSTM
Baselines:CNN/LSTM (Bepler, Tristan, and Bonnie Berger), LSTM(Alley, Ethan C., et al.), one-hot baseline 以及alignment-based baseline
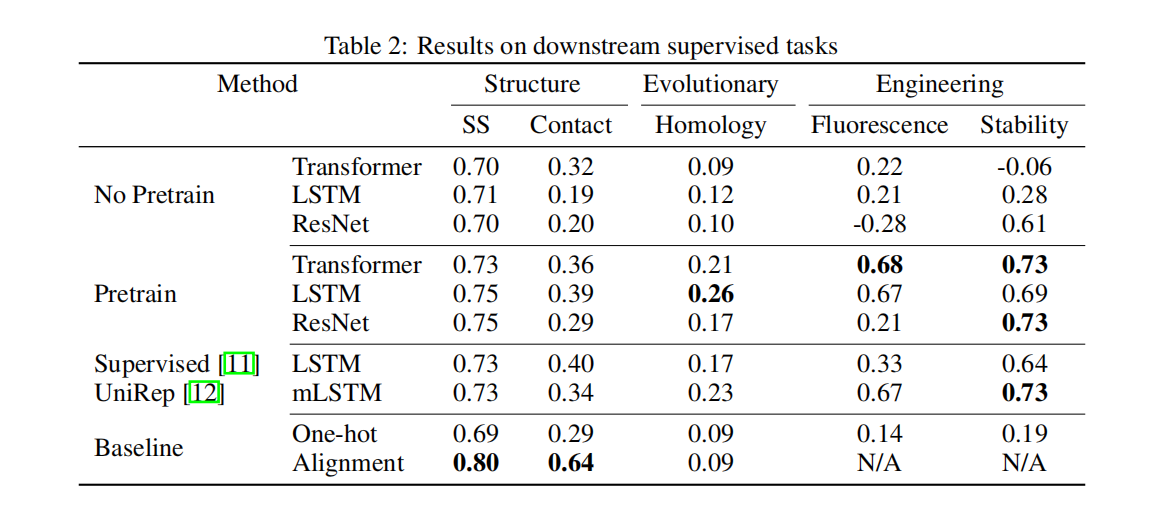
结果:
- 自监督预训练几乎总是可以提升性能;
- 对于结构任务,基于NLP的模型效果不如alignment-based baselines。