论文实现:https://github.com/facebookresearch/esm
facebook蛋白质esm系列工作,主线是利用蛋白质语言模型实现从蛋白序列预测蛋白质结构和功能,介绍了该团队基于Transformer训练的顶尖水准(state-of-the-art)蛋白质语言模型ESM-1b,能够直接通过蛋白的氨基酸序列预测该蛋白的结构、功能等性质
ESM-1b:探究Transformer能否从蛋白序列中提取结构信息
Abstract
AI领域大规模数据和无监督模型的组合能够在表征学习和统计生成方面有很大的进步,作者使用无监督学习训练了一个深度语言模型,针对跨越进化多样性的2.5亿个蛋白质序列中的860亿个氨基酸。学习到的表示空间具有多尺度的组织结构,反映了从氨基酸的生化特性水平到蛋白质的远端同源性的结构,同时,二级和三级结构信息也被编码在了表征中,这些信息能够通过线性投影(linear projection)显化
Introduction
重点是将一个单一的模型适合于跨进化的许多不同的序列。因此,研究高容量的神经网络,研究从大规模建模进化数据中了解蛋白质生物学
蛋白质序列生成和自然语言是很大不同的,不能确定自然语言的目标函数和模型能否迁移到不同领域,作者通过在进化数据上训练高容量的transformer模型来探究这个问题
Scaling language models to 250 million diverse protein sequences
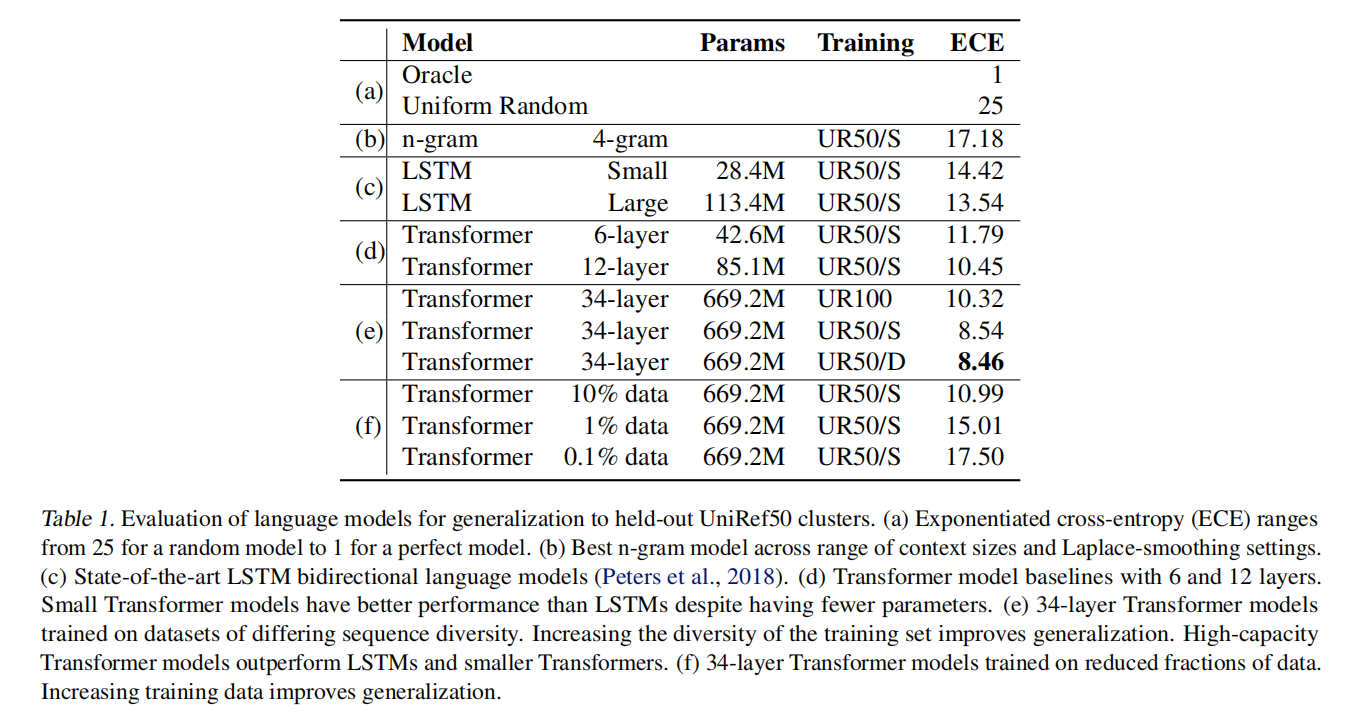
训练数据:UR50/S,训练参数:650M,33层
发现仅仅6层的Transformer模型即可达到比LSTM模型更优的效果;进一步探究发现Transformer,层数越大交叉熵越小,训练数据丰富度越大交叉熵越小。在数据集UR50/D上,34层Transformer交叉熵可降低至8.46,达到了SOTA
本模型的最终目标是探究Transformer能否从蛋白序列中提取结构信息,但该目标不容易直接通过训练实现,因此模型训练使用的是代理任务MLM,基于序列中其他未被遮盖的残基预测被遮盖部分真实的残基是什么
这种代理训练任务能够将蛋白结构特性嵌入特征表示的原因猜想:
- 要根据序列中其他处七年级预测masked token的实际值,就要学习序列中残基之间的联系,而残基联系是蛋白结构在序列中的反映,因此学习了残基联系也就学习了结构功能
Multi-scale organization in sequence representations
我们从生化到进化同源性的多个尺度上研究了网络表示空间,以寻找生物组织的特征。为了理解学习的过程是如何塑造表征的,有必要在表征被训练前后进行比较
Learning encodes biochemical properties

模型的输出嵌入可以看作n维向量,而n维向量又可以看作n维空间中的散点。将这些点通过t-SNE方法降维到二维,发现具有相似生化特征的氨基酸残基在二维平面坐标中被聚成一类(如疏水的、极性的、芳香的、正电性的、负电性的等)
Biological variations are encoded in representation space
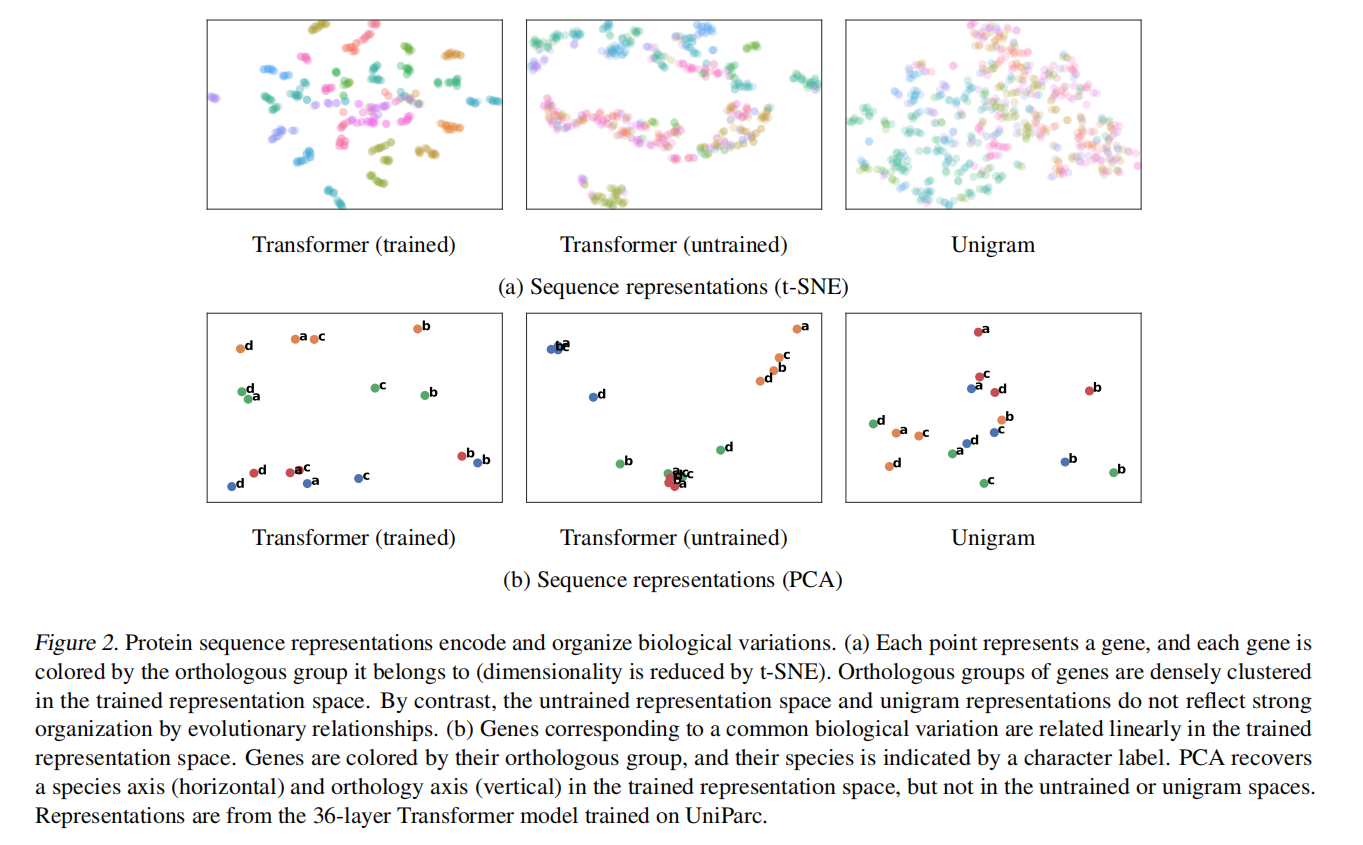
同样将这些点通过t-SNE方法降维到二维,发现代表直系同源基因的蛋白被聚成一类。在二维空间中,沿横轴蛋白被聚成不同物种,沿纵轴蛋白被聚成不同直系同源基因。而未训练的特征表示中蛋白散乱分布
Learning encodes remote homology & Learning encodes alignment within a protein family
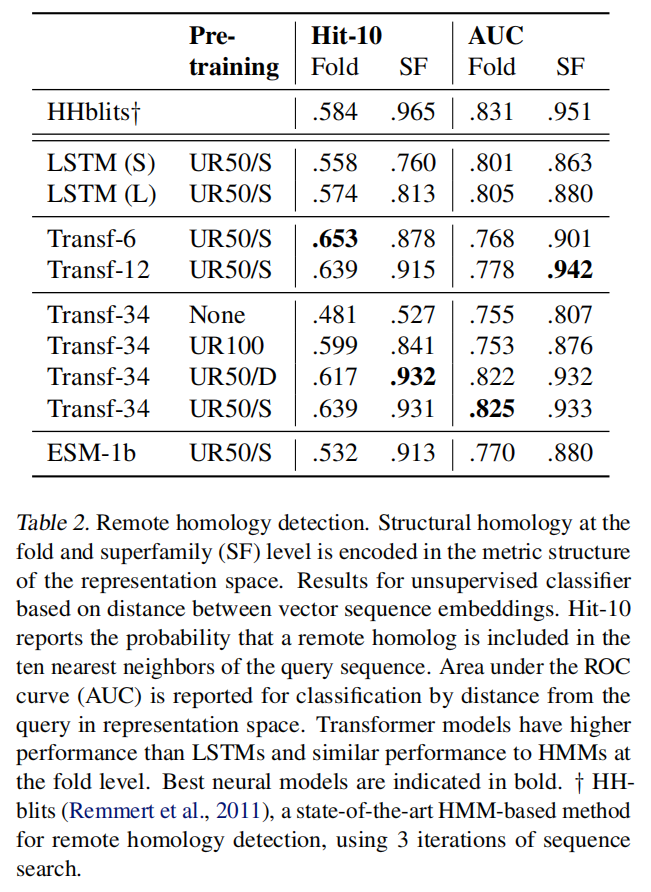
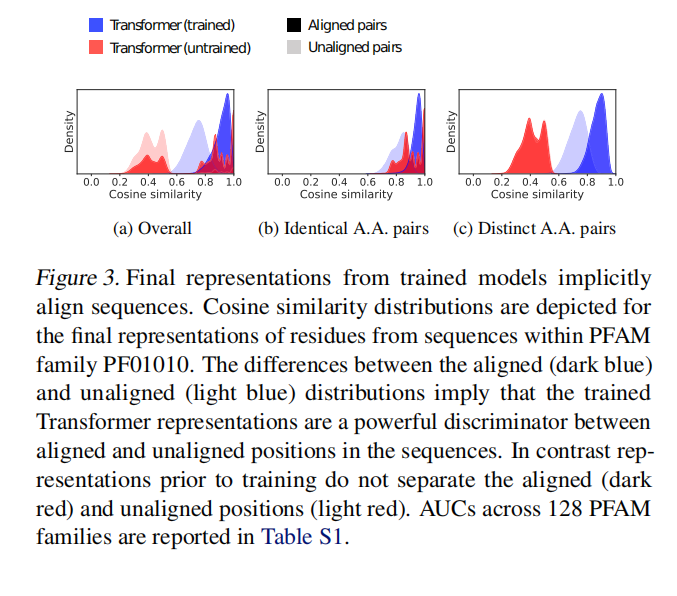
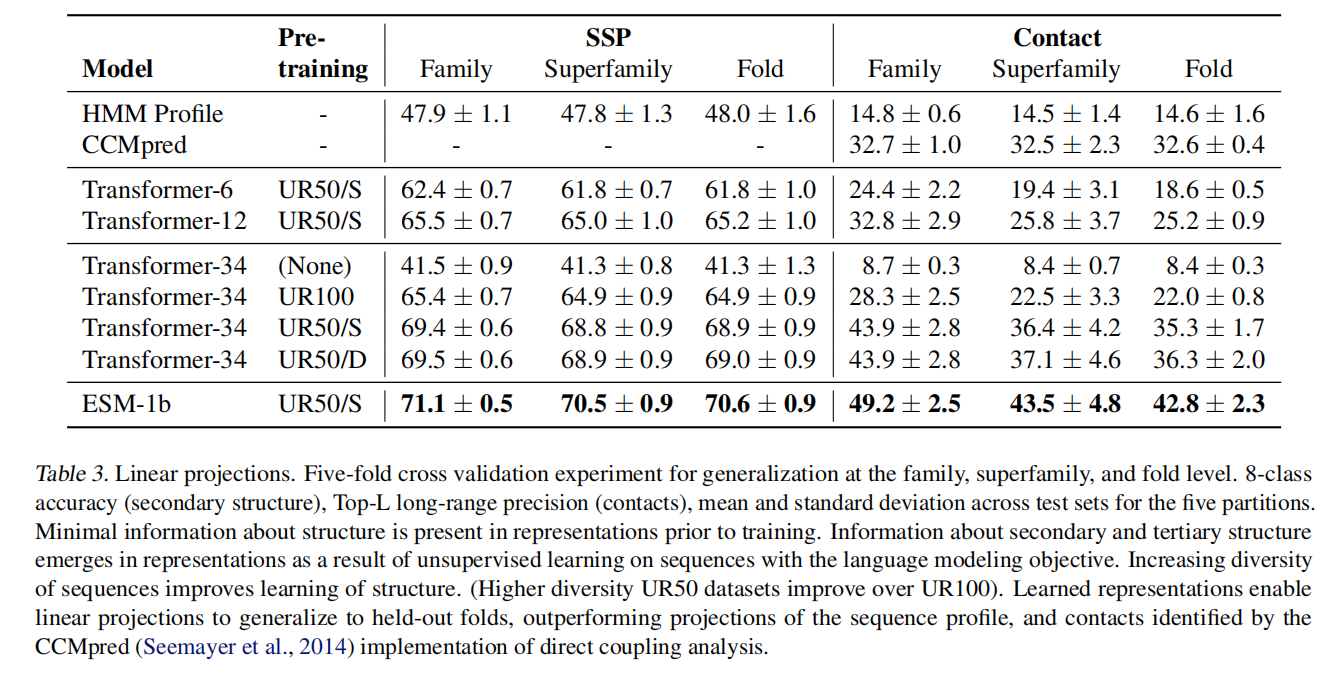
基于训练后的特征表示,用SCOPe方法预测蛋白是否属于同一superfamily或fold
Prediction of secondary structure and tertiary contacts
-
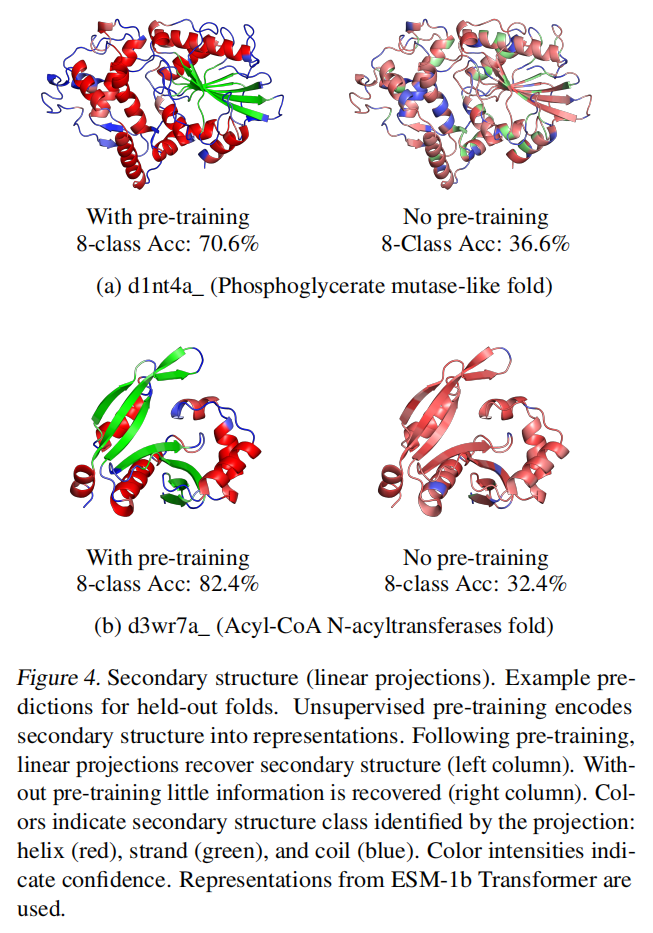
二级结构
- 基于ESM-1b的特征表示,以蛋白的8种二级结构作为标签,拟合logistic回归模型来预测蛋白序列的二级结构
-
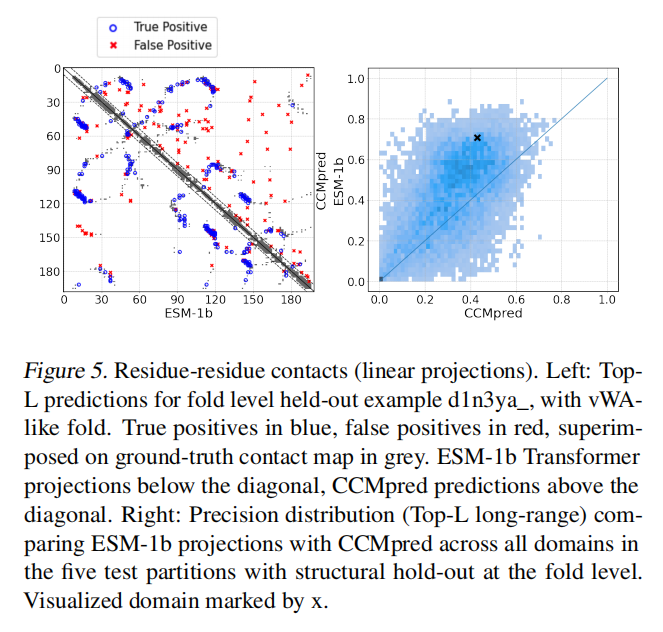
三级结构
- 为序列两个位点分别做线性投影,二者做点积,得到一个二元变量,1表示两位点在蛋白质三级结构中有联系,0表示二者在三级结构中无联系


