论文地址:Modeling Protein Using Large-scale Pretrain Language Model
ProteinLM:悟道·文溯的30亿参数蛋白预训练模型
Abstract
使用30亿参数的语言模型对进化规模的蛋白质序列进行预训练建模,将蛋白质生物学信息进行编码表示。模型在5个标识符级和序列级的下游任务中都获得了明显的改进,表明本文的大规模模型能够准确地从进化规模的单个序列预训练中捕捉到进化信息
Introduction
传统蛋白质实验分为两部分:实验和分析,实验包括纯化,晶体化和X光晶体学等;分析包括序列比对,分子动力学模拟;难以处理大规模的蛋白质数据
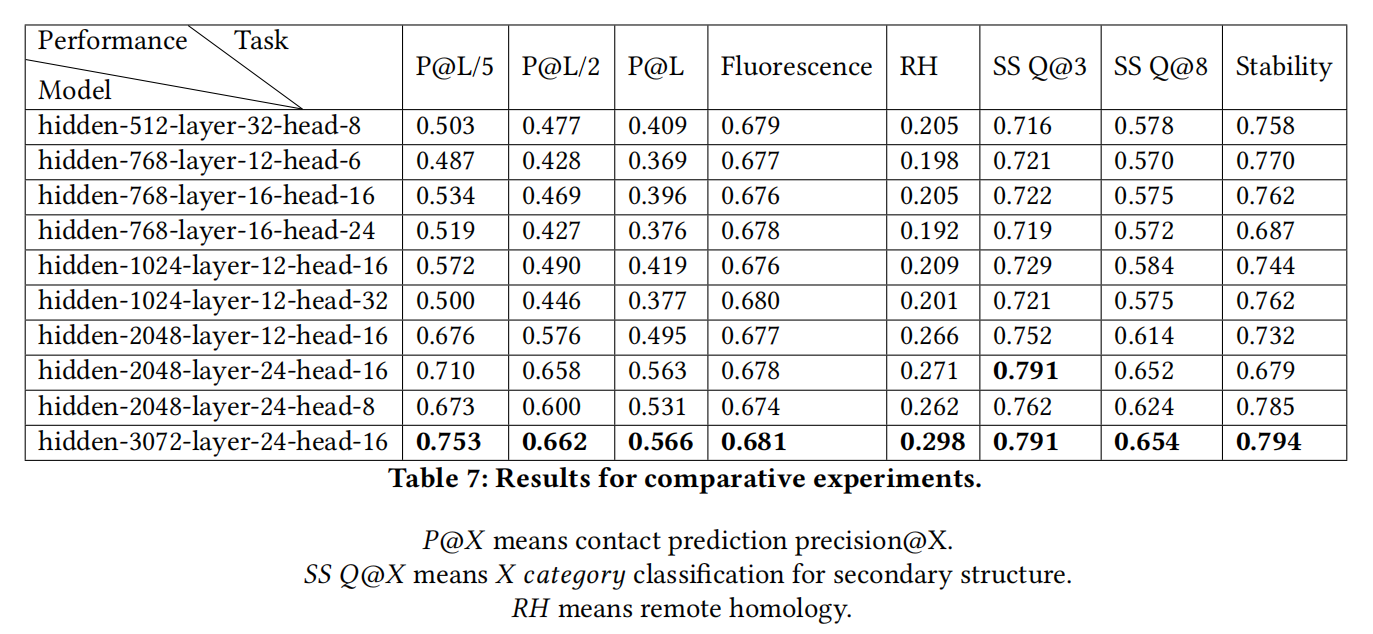
在PFAM数据集上训练,有30亿参数,超过了TAPE的性能
Methodology
Training Objective
用的BERT的MLM和NSP
Downstream Classification Task
三个分类任务,对应词元,序列和词元对分类
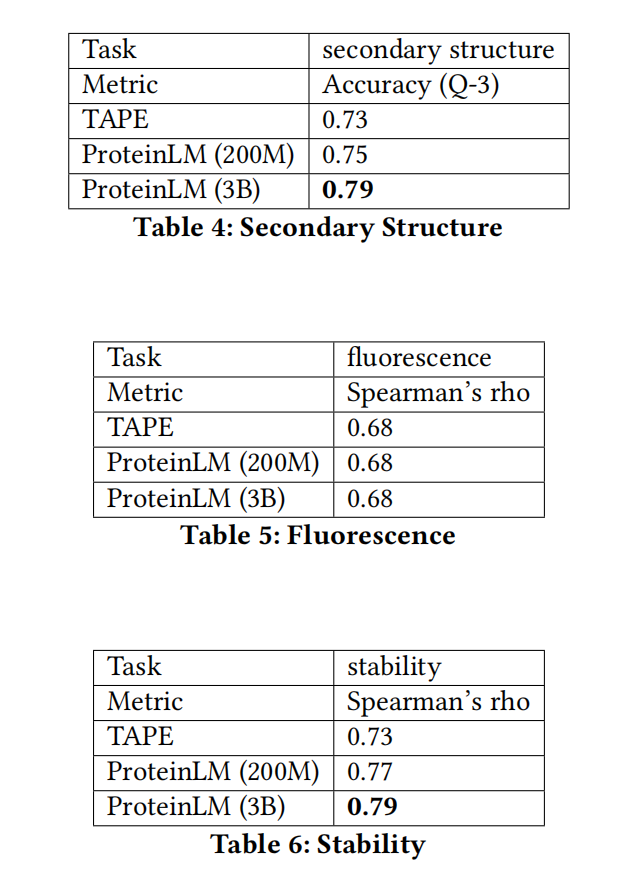
secondary structure
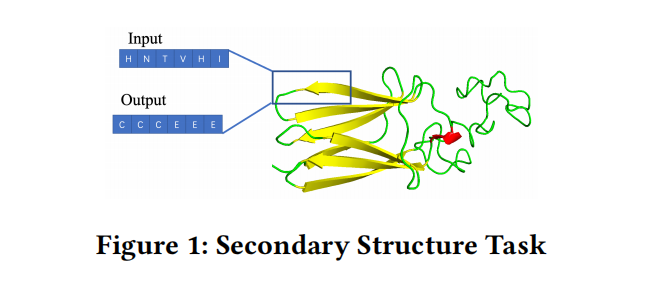
这是一个token-level任务,输入蛋白质序列,输出等长的Label序列,表示相应氨基酸的二级结构位置
数据集是CB513

Remote Homology
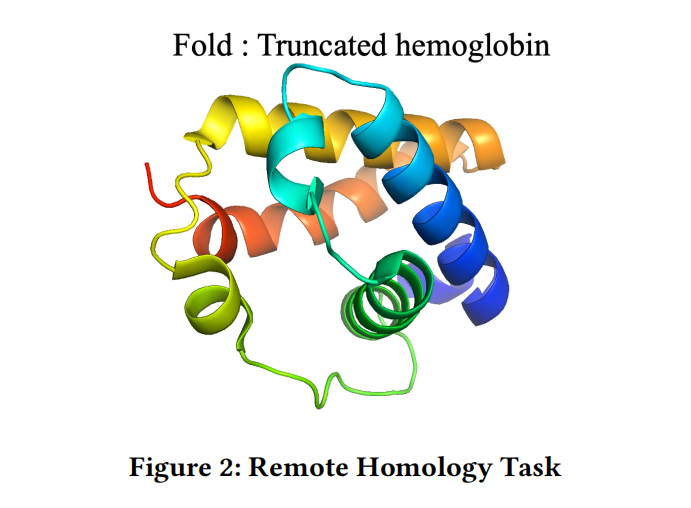
是一个sequence-level任务,检测相关输入的结构相似性,总共有1195个类别
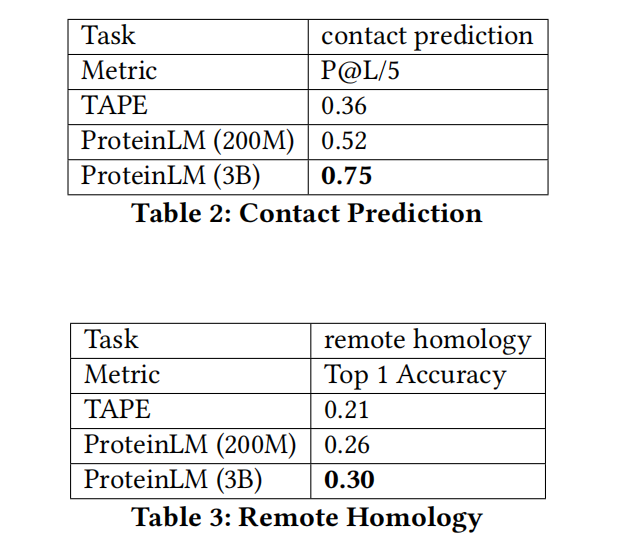
Contact Prediction
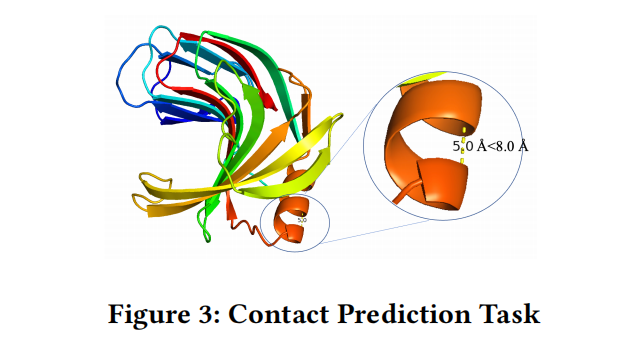
是指预测折叠结构中的氨基酸对是否处于“接触”状态,in contact是指折叠结构的距离在8angstroms内
数据集来源于ProteinNet
Downstream Regression Task
Fluorescence
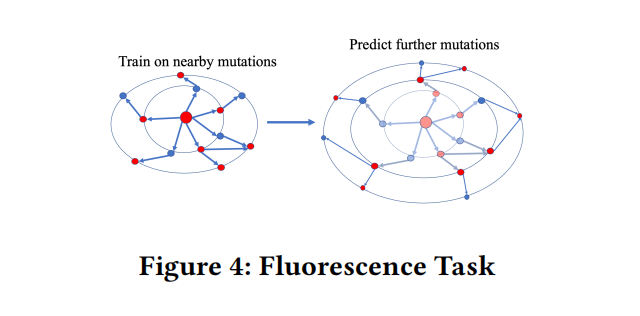
区分具有不同突变的蛋白质序列可能是困难的,因为计算成本随着突变的数量𝑚呈指数增长,荧光预测任务可以评估模型区分非常相似的蛋白质序列的能力,以及它推广到看不见的突变组合的能力
Stability
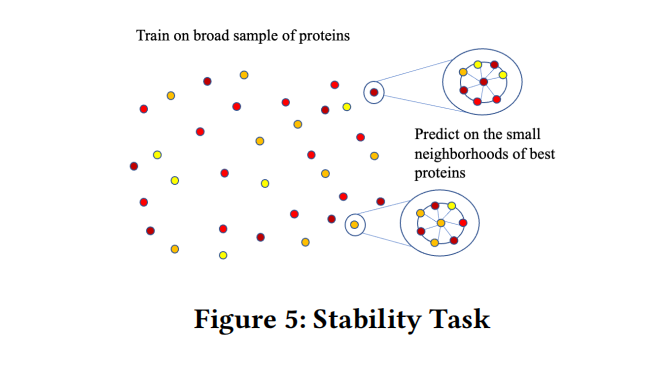
蛋白质可以保持其原始折叠结构的浓度上限,输入是氨基酸序列,而输出是一个连续的值,预测蛋白质在多大程度上可以保持其折叠结构
Result
Training
480块v100的服务集群训练两种模型3个星期,用的megatron-lm
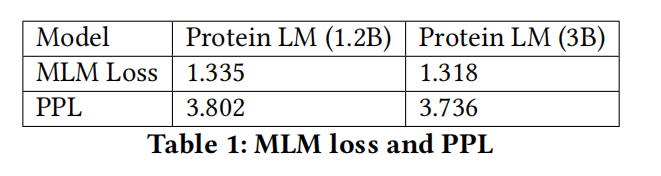
Evalution