论文地址:OntoProtein:Protein Pretraining With Gene Ontology Embedding
OntoProtein:知识图谱+对比学习+预训练微调
Abstract
目前预训练数百万序列的蛋白质语言模型可以将参数规模从百万级提高到十亿级,但先前的工作很少考虑知识图谱(KG),作者认为增加知识图谱可以增强蛋白质表征。本文提出OntoProtein,第一个使用GO(Gene Ontology)结构到蛋白质预训练模型中。构建了一个新的大规模的包含GO和相关蛋白质的知识图谱,提出了一种新的知识感知负抽样的对比学习,在训练前联合优化知识图和蛋白质嵌入
Introduction
目前的蛋白质预训练模型不能充分捕捉生物学上的factual knowledge,所以作者采用了来自基因本体论的知识图。由于蛋白质的形状决定了其功能,因此模型更容易利用具有相似形状的蛋白质功能的先验知识来识别蛋白质的功能
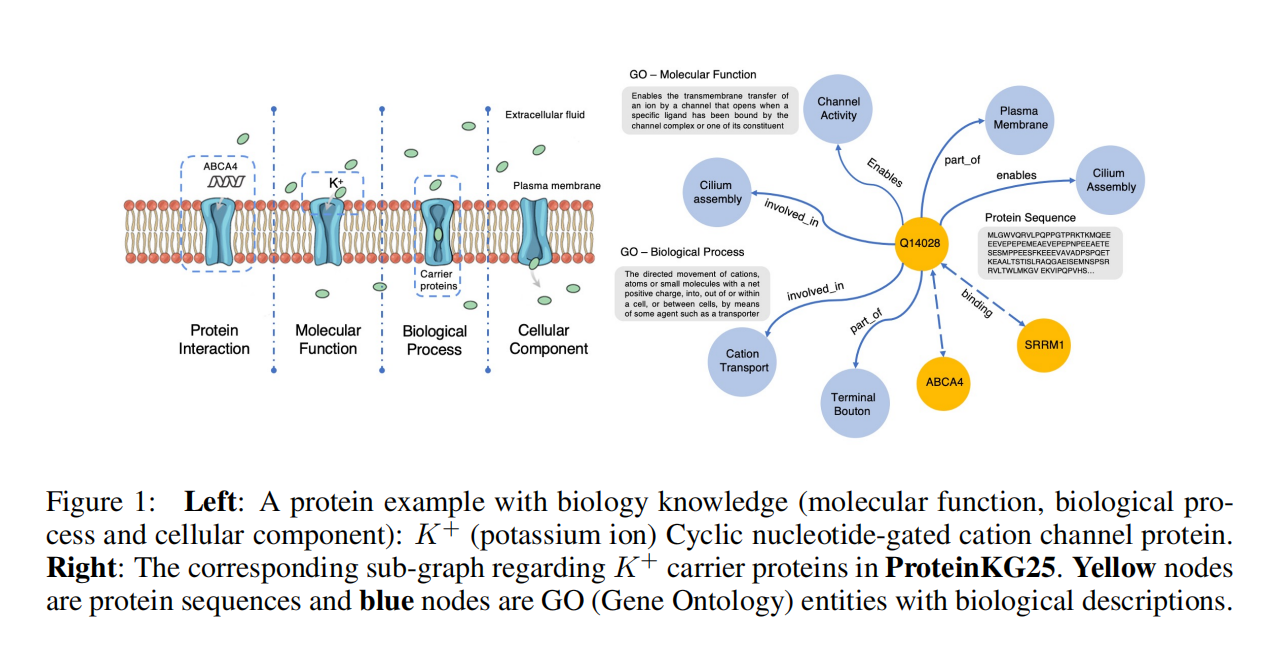
但是蛋白质序列和基因本体论数据类型不同,序列是氨基酸,但基因本体论是文本描述的知识图谱,因此,结构化知识编码和异构信息融合的严重问题仍然存在
通过访问公开的基因本体知识图谱“Gene Ontology(Go)”,并将其和来自Swiss-Prot数据库的蛋白质序列对齐,来构建用于预训练的知识图谱ProteinKG25,该知识图谱包含4,990,097个三元组, 其中4,879,951个蛋白质-Go的三元组,110,146 个Go-Go三元组
主要贡献:
- 提出了第一个知识增强的蛋白质模型OntoProtein
- 通过对比学习与知识感知采样,共同优化知识和蛋白质嵌入
- 构建了ProteinKG25数据集
Methodologies
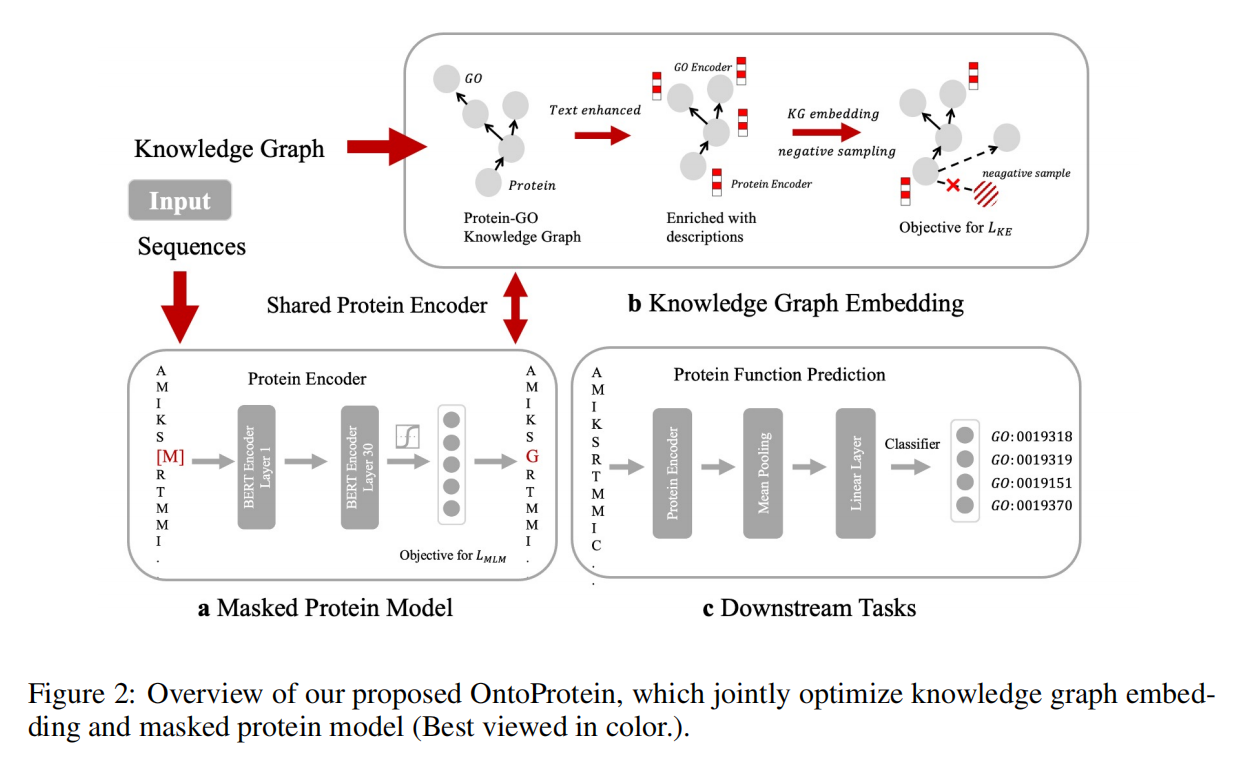
Hybrid Encoder
对蛋白质序列采用已有的蛋白质预训练模型ProtBert进行编码
对文本序列(GO encoder)采用BERT进行编码,也是用的预训练好的模型
Knowledge Embedding
使用Knowledge Embedding objective来获取知识图谱的表征
知识图谱由一堆元组组成,可以定义为(h, r, t),h 和 t 是 head 和 tail 实体,r 是关系类型
这里有两种不同的节点, $e_{GO}$ 和 $e_{protein}$ ,前者是图里的注释文本,后者是蛋白质序列,整个知识图谱可以定义为两组 $triple_{GO2GO}$ 和 $triple_{Protein2GO}$
Contrastive Learning With Knowledge-Aware Negative Sampling
KE是学习实体和关系的低维表征,对比估计是一种可扩展的、推理连接模式的有效方法
对比学习是把分布损坏来得到负样本,迫使模型去学习更有区别性的特征和关键特点,但先前的方法都太简单了,本文提出知识感应的负样本采样方法,KE目标如下:
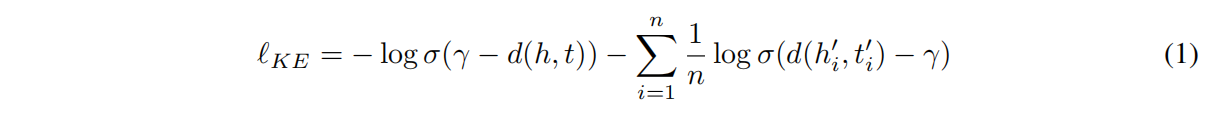
其中 $(h_t’,t_i’)$ 是负样本,head和tail实体是损坏的元组里随机采样的,n是负样本数,γ是范围值,d是打分函数,为了简单使用TransE
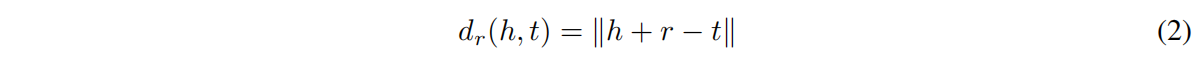
定义元组集和实体集分别为T和E,GO里的实体属于MFO(Molecular Function),CCO(Cellular Component)和BPO(Biological Process),对于 $T_{GO-GO}$ 元组,采样元组替换实体的时候用同一个方面的(MFO, CCO, BPO),最后定义负样本元组集 $T’$ ,采样如下:
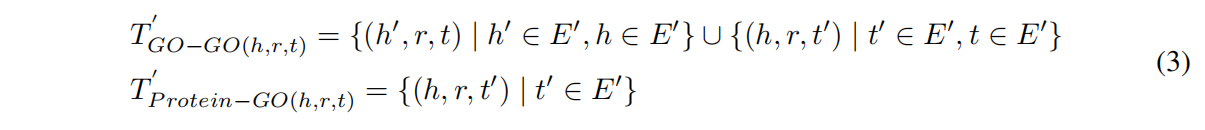
这里 $E’\in {E_{MFO},E_{CCO},E_{BPO}}$,对 $T_{Protein-GO}$ 只替换了tail实体
Pre-training Objective
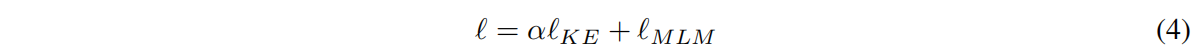
α是超参,整个方法可以嵌入到现有的微调场景中
Experiment
TAPE Benchmark
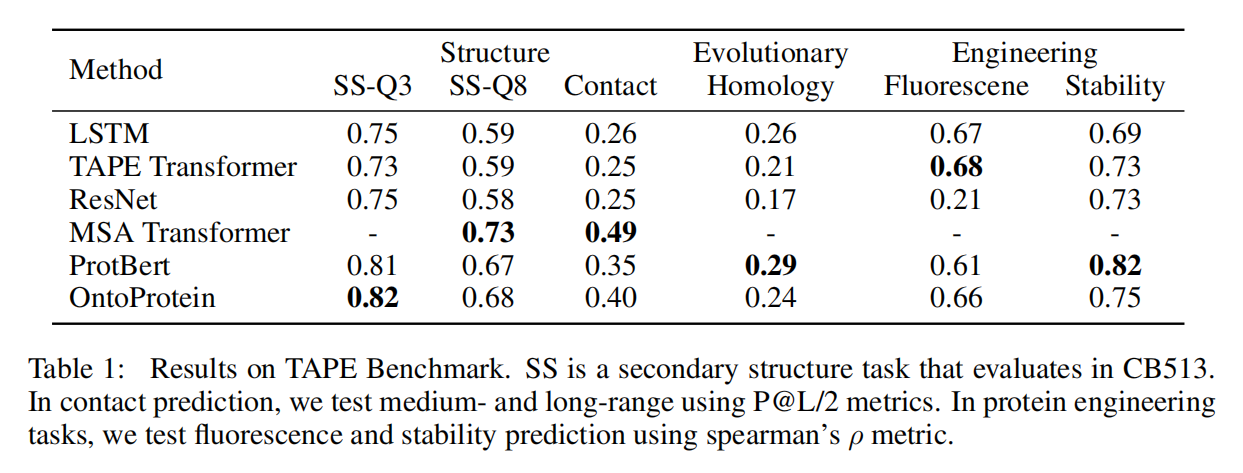
在token level上没有用MSA可以取得有竞争力的结果,但在sequence level上效果不好,怀疑是缺少了sequence level的目标函数,留待后续工作
Protein-Portein Interaction
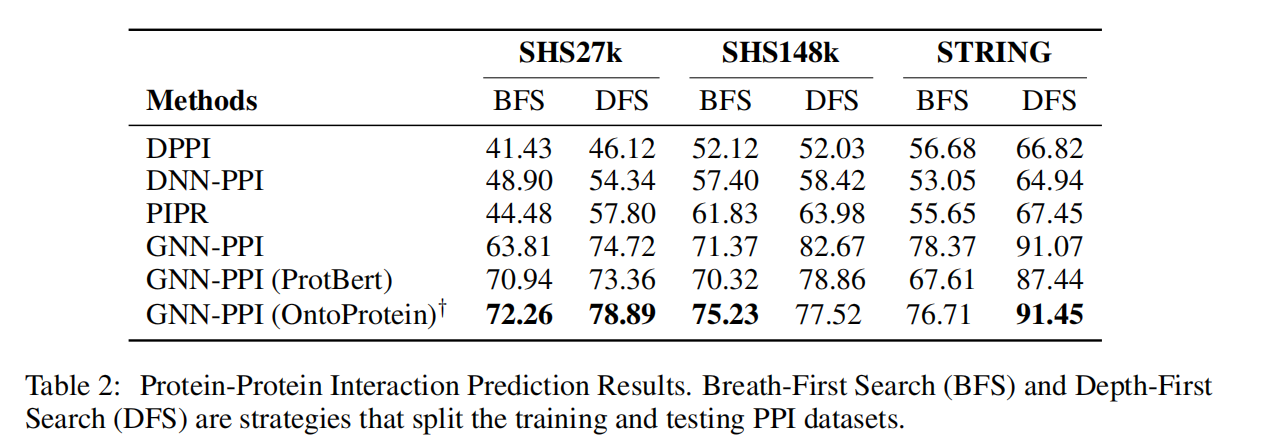
数据小效果好,数据大只能说有竞争力
Protein Function Prediction
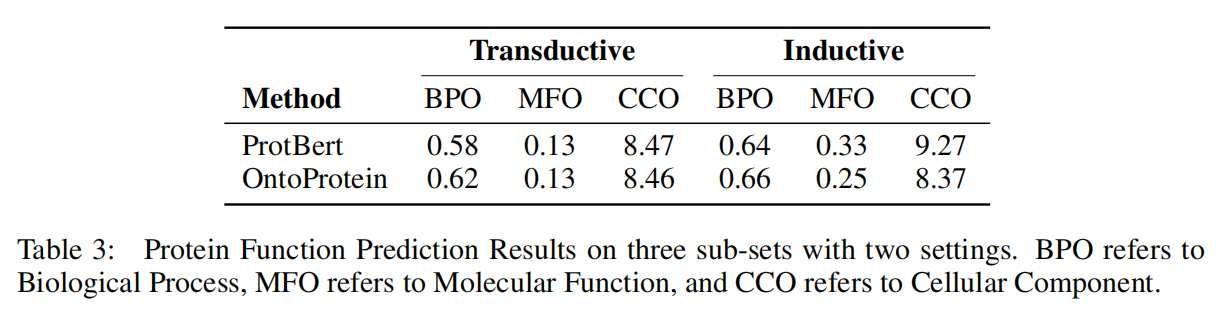
数据集中可能存在严重的长尾问题,知识注入可能会影响head的表示学习,但会削弱tail的表示,从而导致性能下降,留待后续工作
Analysis
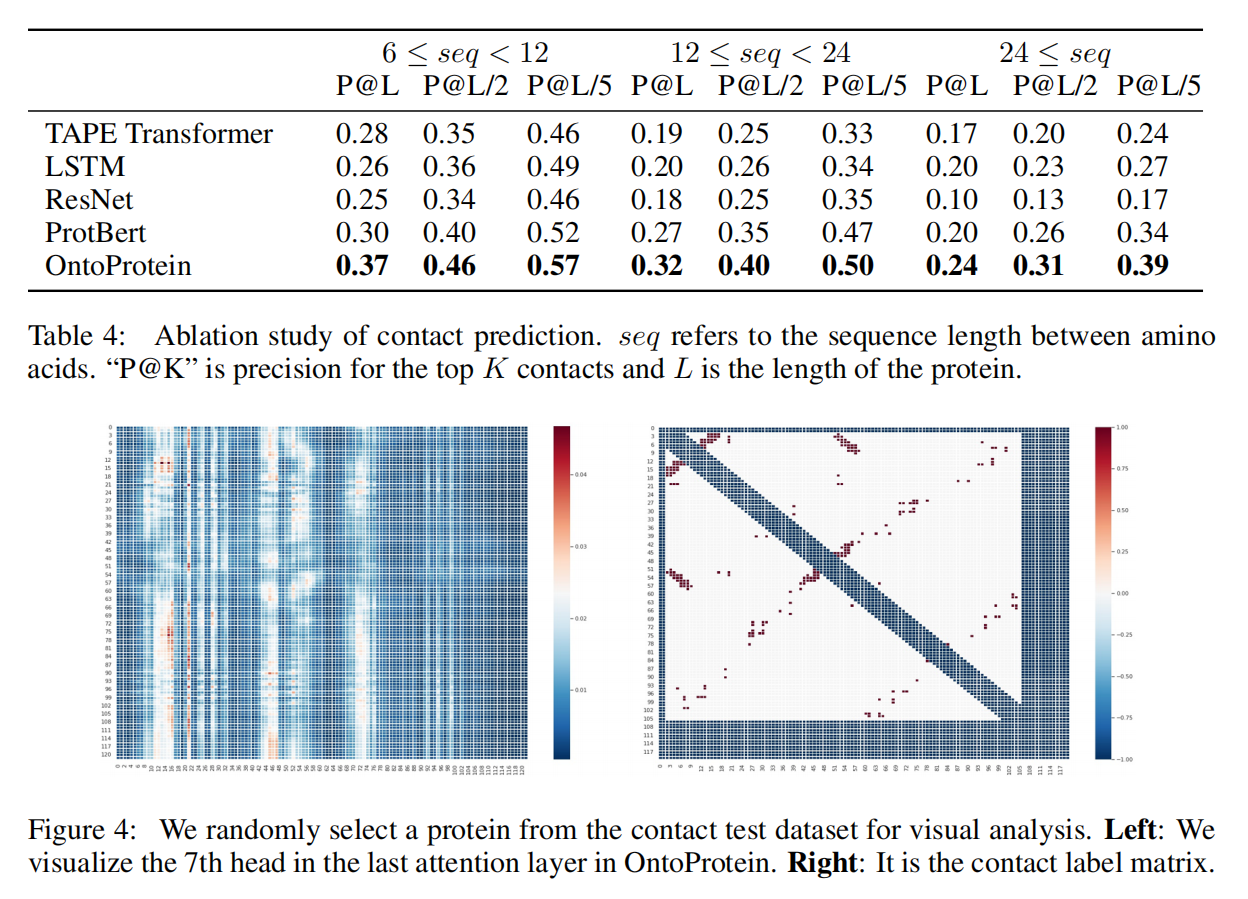
在contact prediction的不同设置下都有不小的提升


