Masked sequence-structure:掩码序列和结构做热稳定性单突变
Abstract
提出了一个更加数据高效的方法,通过半监督的方法联合训练蛋白质序列和结构来编码蛋白质信息,实验表明这种方法可以编码两种信息来形成一个丰富的嵌入空间更好的利用到下游任务当中。通过预测单个突变的影响,将丰富的结构信息纳入所考虑的环境中,可以提高模型的性能。我们将准确性的提高归因于利用丰富表示中的邻近性,以识别将受到突变影响的顺序和空间上接近的残基,使用实验验证或预测的结构
Results
Masked Structure and Sequence Recovery
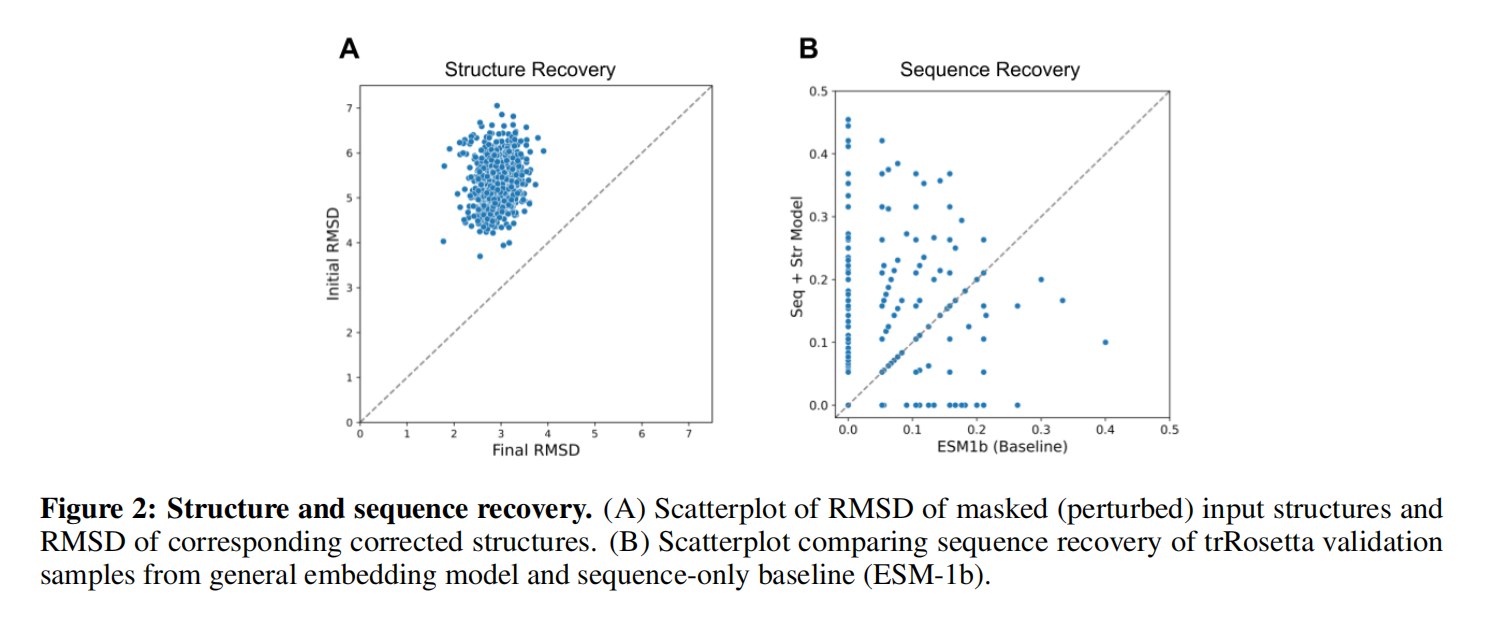
把连续的5个残基mask掉得到总共15%的masked residues,图2a是masked validation points,平均而言,初始验证样本是5angstroms扰动,校正后样品与各自天然蛋白质的RMSD约为2埃;图2b的esm-1b随机选了15%的token进行mask,增加了结构信息后准确率是0.13,纯粹的序列准确率是0.06
Predicting the effect of a single mutation on thermal stability
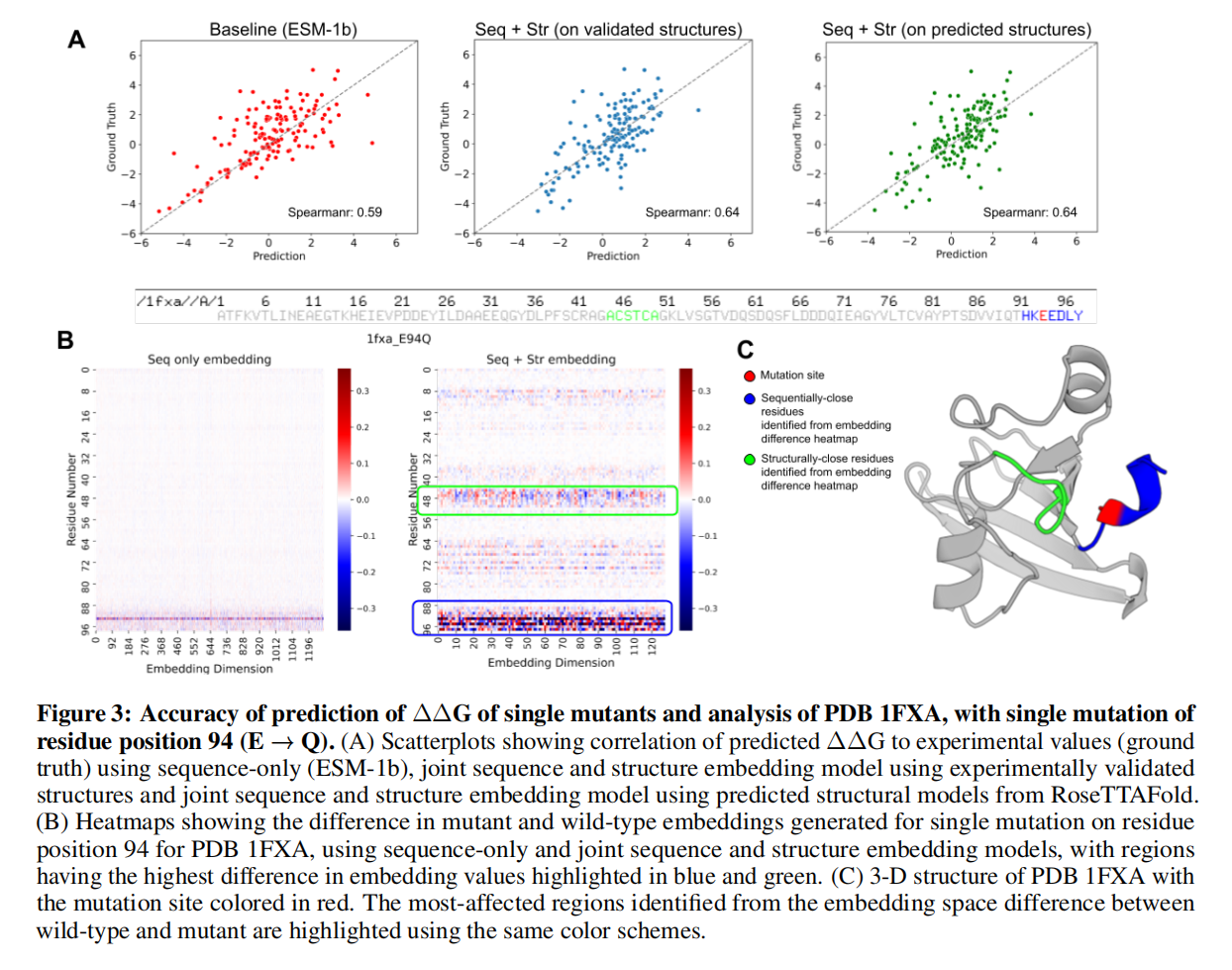
对sequence-only baseline和joint embedding model都微调了序列预测头(LMHead),结果如图3a
Evaluation of mutant vs wild-type embedding space
structure-aware embedding能比wild-type和mutant embedding spaces在PDB 1FXA的度数更高,结果如图3b
Single mutation effect prediction using predicted models
结果如图3c
Discussion
把结构和序列联合进嵌入模型学习可以更加的data-efficient,减少训练样本数量和计算量
用半监督的范式使用的模型是SE(3)-equivariant Transformer
本文的模型比sequence-only的模型在单点突变上更敏感,在热稳定性单点突变上效果更好
预测单突变效果用的RoseTTAFold模型
还有一些方向可以提升联合模型的性能:比如增加模型参数,预测原子的修复
Methods
Input sequence embedding and structure information
使用trRosetta2数据集,把masked sequence输入预训练好的esm-1b,使用最后一层sequence embedding和模型33层里每一层的attention map。对于结构,只考虑N,Ca,C和Cb原子,长度限制128
Training Objective and Details
最小化mask的氨基酸和预测的交叉熵,优化器是Adam($\beta1=0.9,\beta2=0.999$),学习率1e-3,batch_size=128
Model Architecture
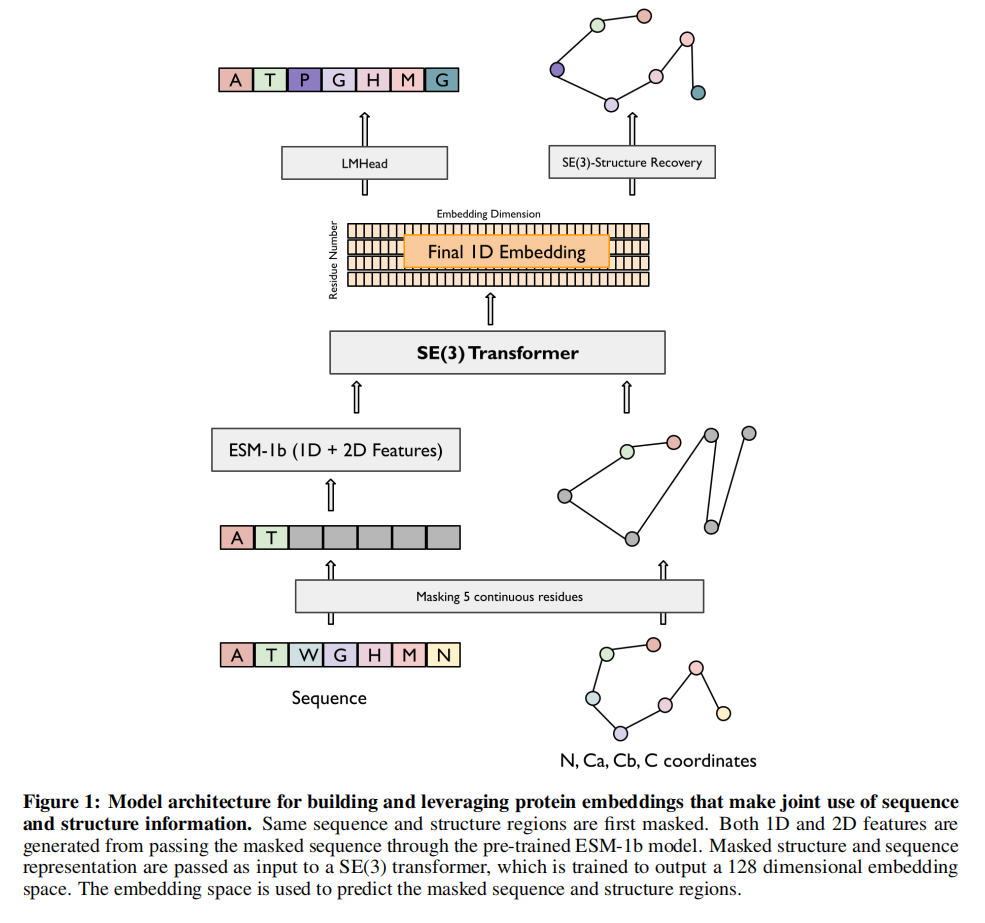
使用一个5层的图神经网络SE(3)-equivariant transformer,1D(sequence embedding) and 2D(attention map) esm-1b特征线性投影到32位来做noise reduction
构造图时考虑16个邻居节点,节点特征包括每个残基的ESM-1b序列嵌入和每个原子到Ca的位移向量(NCa、CCa、CbCa),边特征包括ESM-1b二维attention map和残基间的方向
LMHead是2层全连接+layer normalization,SE3-SR top model是3层SE(3)-transformer
Fine-tuning for single-mutant effect prediction
在预测单突变的热稳定性上微调,用的ProTherm子数据集,有126个wild-type蛋白质的1042个mutants,训练集测试集8:2


