论文地址:Learning from Protein Structure with Geometric Vector Perceptrons
GVP:集合向量感知机执行几何和关系推理
Abstract
我们提出了geometric vector perceptrons,扩展了标准的稠密层来操作欧几里得向量,配备了这种层的图神经网络能够对大分子的有效表示执行几何和关系推理。在model quality assessment和computaional protein design进行评估
Introduction
深度学习技术往往利用了领域的问题结构,比如CNN于CV,attention于NLP
一方面有氨基酸残基在空间上的排列和取向,它控制着分子的动力学和功能;另一方面,蛋白质在其氨基酸序列和介导上述蛋白质性质的残基-残基相互作用方面也具有关系结构;把这些分别称为geometric and relational aspect
最近使用结构信息的方法主要两方面:用GNN的关系推理或CNN直接操作空间结构
提出了GVP可以同时处理标量和几何特征,可以应用于输入域是单个大分子或分子相互结合的结构的任何问题;本次工作主要关注于两个蛋白质结构相关的问题:computional protein design and model quality assessment
Methods
提出的架构集合CNN和GNN的学习生物分子的结构的方法
前一节中描述的GNN通过按照旋转不变量标量编码向量特征(如节点方向和边方向),通常通过在每个节点定义局部坐标系,对蛋白质的3D几何结构进行编码。本文建议将这些特征直接表示为3D几何向量特征,这些特征在图传播的所有步骤中,在空间坐标的变化下进行适当的变换
有两个好处:
- 输入特征更加方便,用每个节点的绝对位置编码来替代该节点与其他所有邻居的相对位置编码
- 标准化了整个结构的全局坐标系,允许几何特征直接传播,无需局部坐标之间的转换
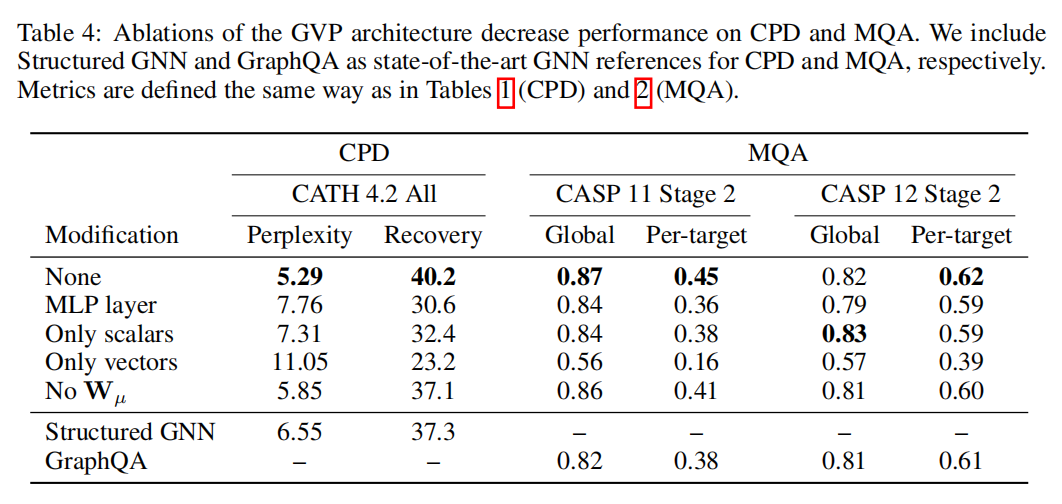
Geometric vector preceptrons
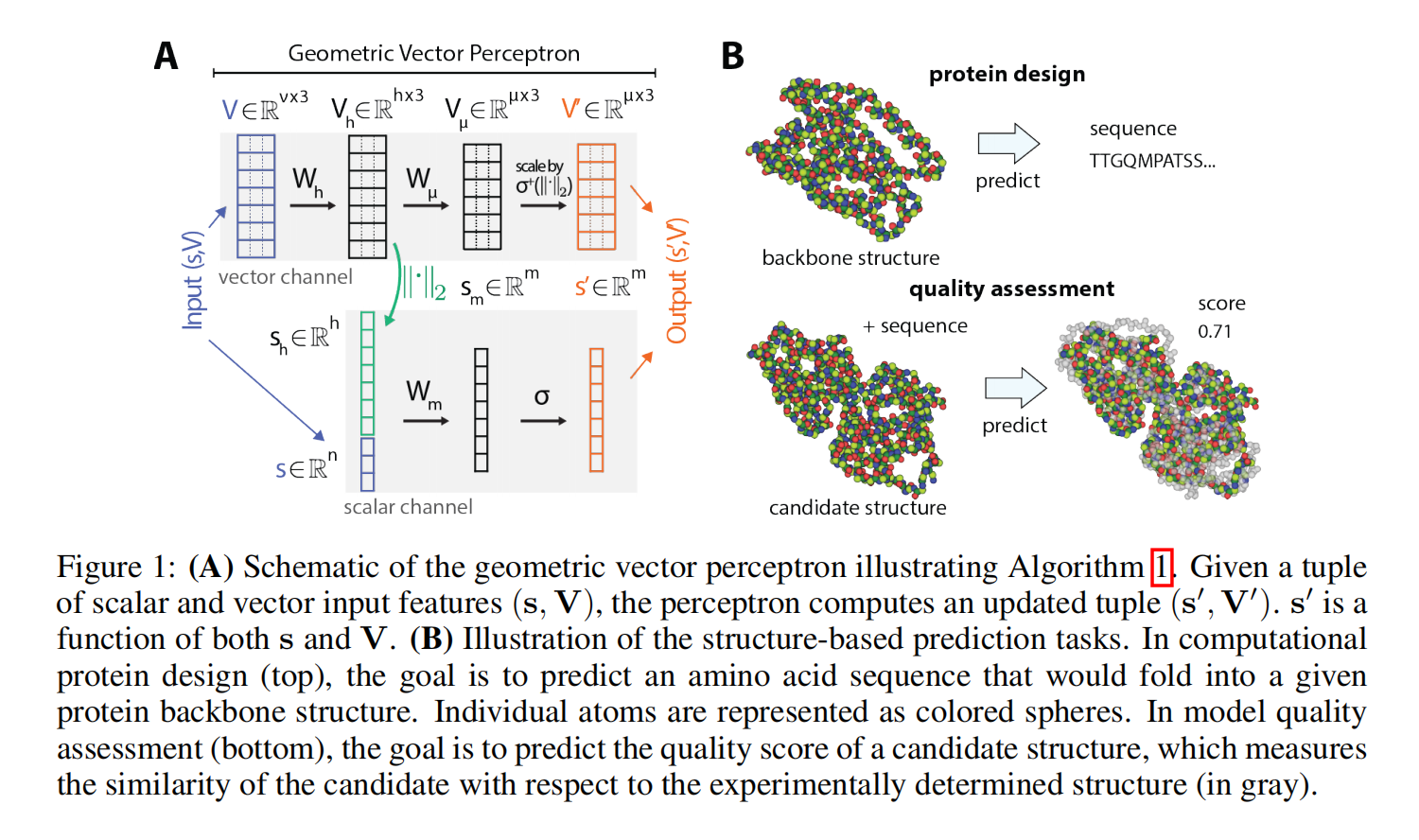

给定一个元组 $(s,V)$,其中标量特征 $s\in \mathbb{R}^n$,向量特征 $V\in \mathbb{R}^{v\times 3}$ ,计算得到新的特征 $(s’,V’)\in \mathbb{R}^m\times \mathbb{R}^{u\times 3}$
有两个分开的线性变换矩阵分别对应标量和向量,后面是非线性激活,L2 norm
这是因为对向量值输入的唯一操作是标量乘法、线性组合和L2范数。作者在附录A中提供了正式证明。下面的截图是作者在论证GVP体系结构可以逼近V \mathbf{V}V的任何连续旋转和反射不变标量值函数
Representations of Proteins
作者的体系结构的主要经验验证是它在两个实际任务上的性能:计算蛋白质设计(CPD)和模型质量评估(MQA)。这些任务,如图1b 所示,并在第4节详细描述,是互补的,其中一个(CPD)预测每个氨基酸的属性,而另一个(MQA)预测全局属性
将一个蛋白质结构输入表示为一个邻接图,带有最少数量的标量和向量特征,以指定分子的三维结构。蛋白质结构是一个氨基酸序列,其中每个氨基酸由四个主干原子和一组位于三维欧氏空间的侧链原子组成。只表示主干,因为侧链在CPD中是未知的,并且MQA基准只对应于主干结构的评估
蛋白质的主干部门表示成一个图 $G=(\nu, \varepsilon)$,每个点 $\nu_i$ 对应一个氨基酸,并且有一个embedding $ \mathbf{h}_\mathbf{\nu}^{(i)} $,该embedding具有如下特征:

边选择30个最近邻,每个边 $h_e^{(j\rightarrow i)}$ 特征如下:
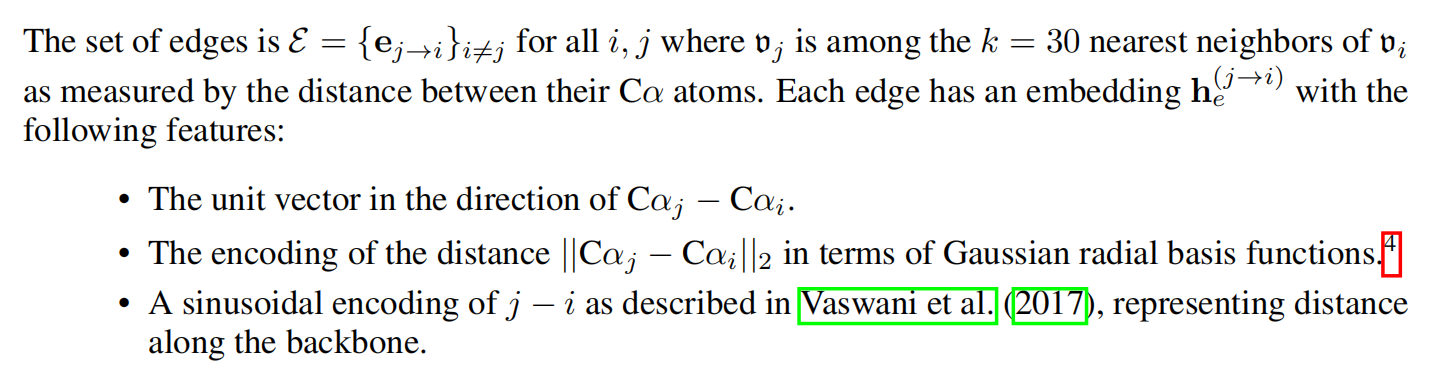
Network Architecture
图传播步骤如下:
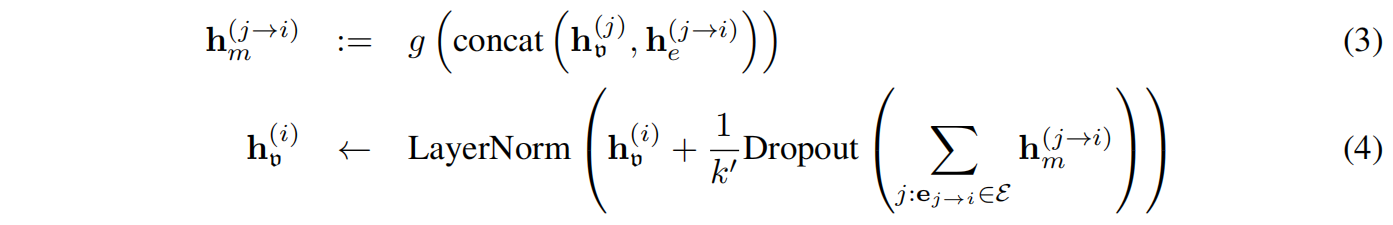
其中,g是三个GVPs的叠加函数
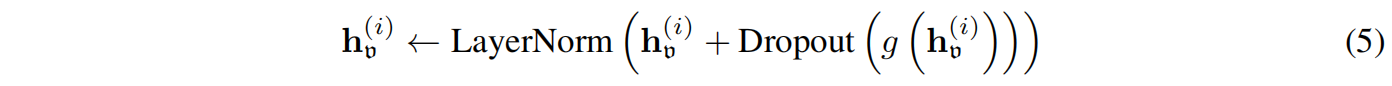
其中,g是两个GVPs的叠加函数。这些图传播和前馈步骤除了更新每个节点的标量特征之外,还更新了其向量特征
Evaluation metrics and datasets
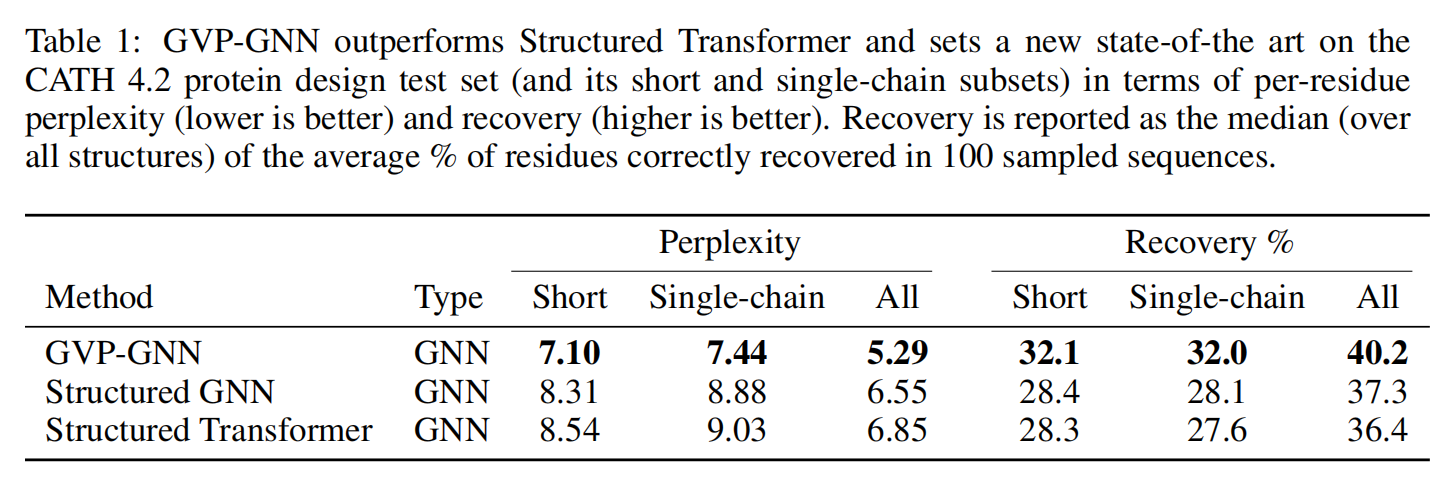
Protein design
计算蛋白质设计(CPD)是蛋白质结构预测的概念反转,旨在推断将折叠成给定结构的氨基酸序列
使用数据集是CATH4.2,以及评估了TS50,一个由50个原生结构组成的较旧测试集
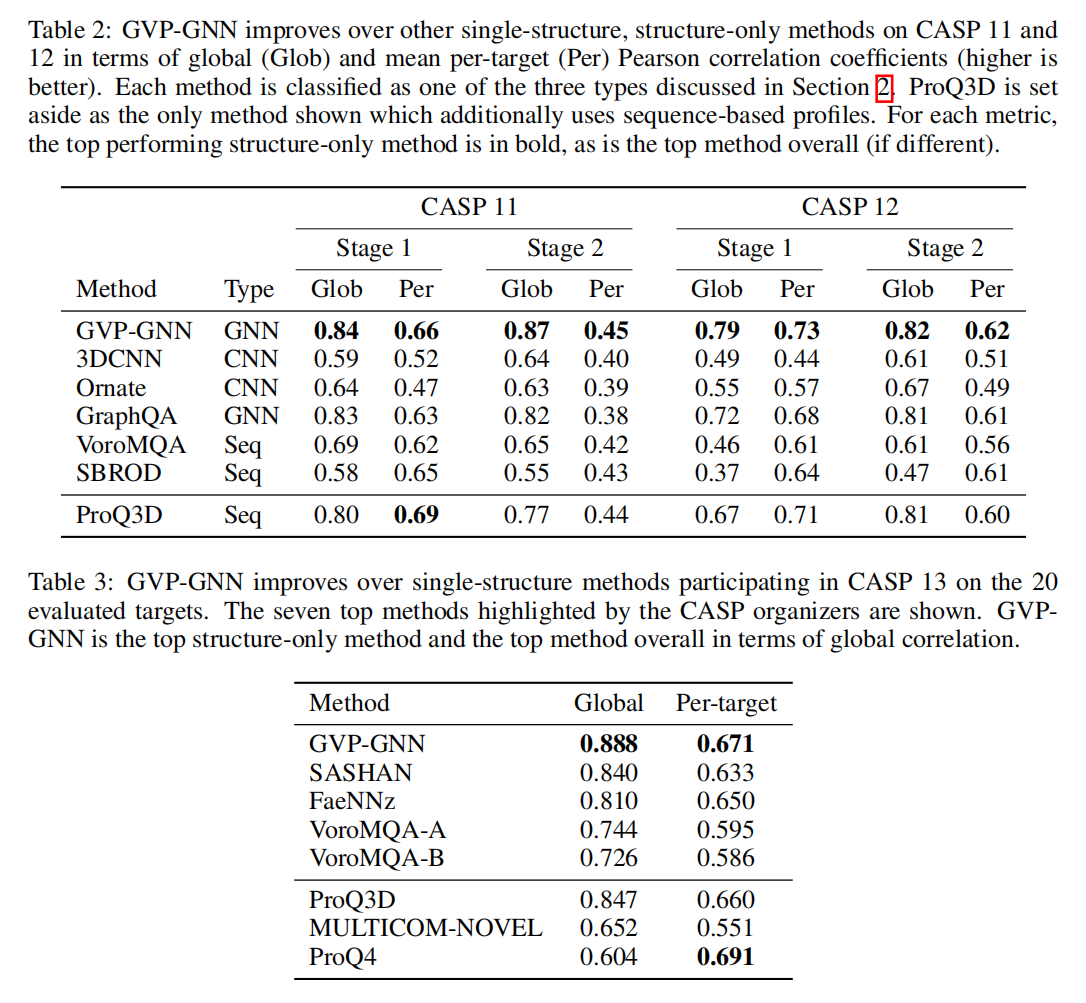
Model quality assessment
模型质量评估(MQA)旨在从大量候选结构中选择蛋白质的最佳结构模型
使用CASP数据集
Experiments