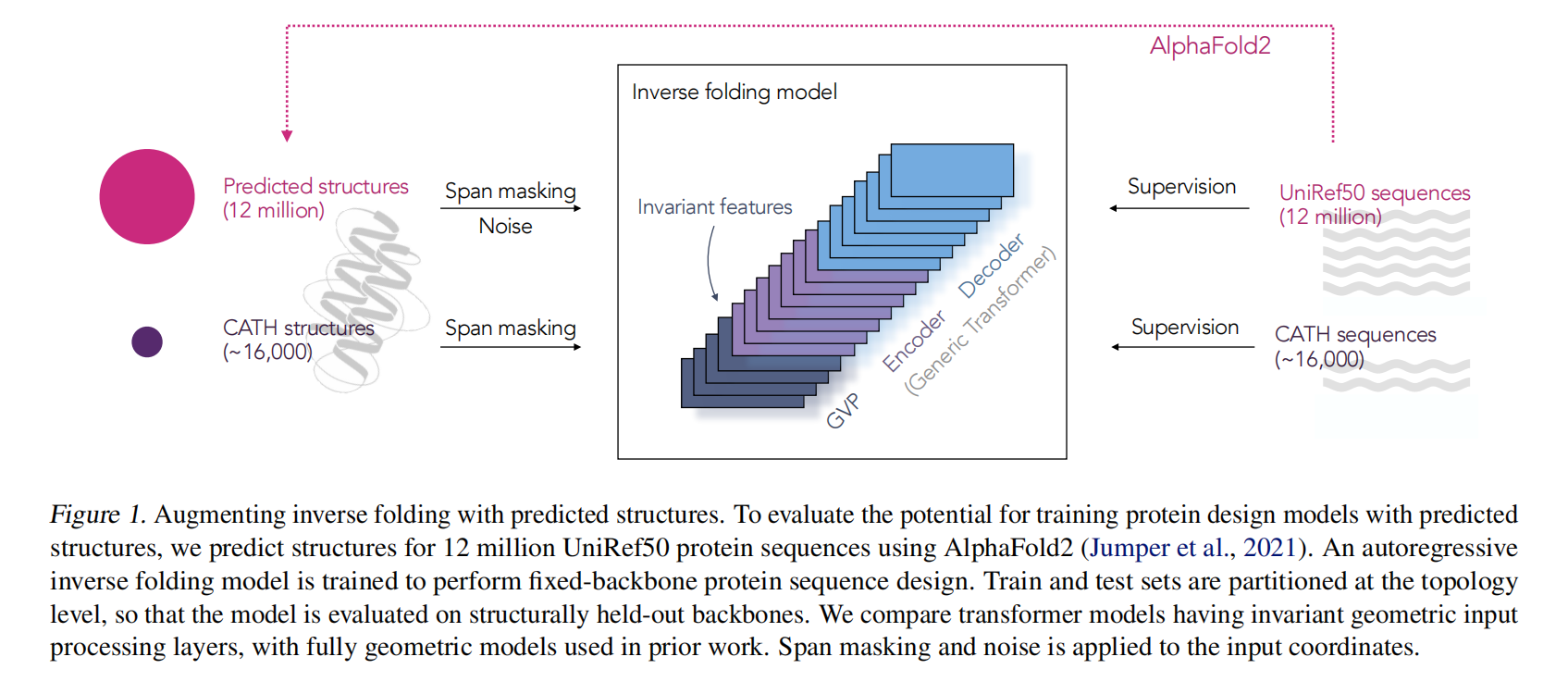
论文地址:Learning inverse folding from millions of predicted structures
ESM-IF:额外使用UniRef50的12M数据做inverse fold
Abstract
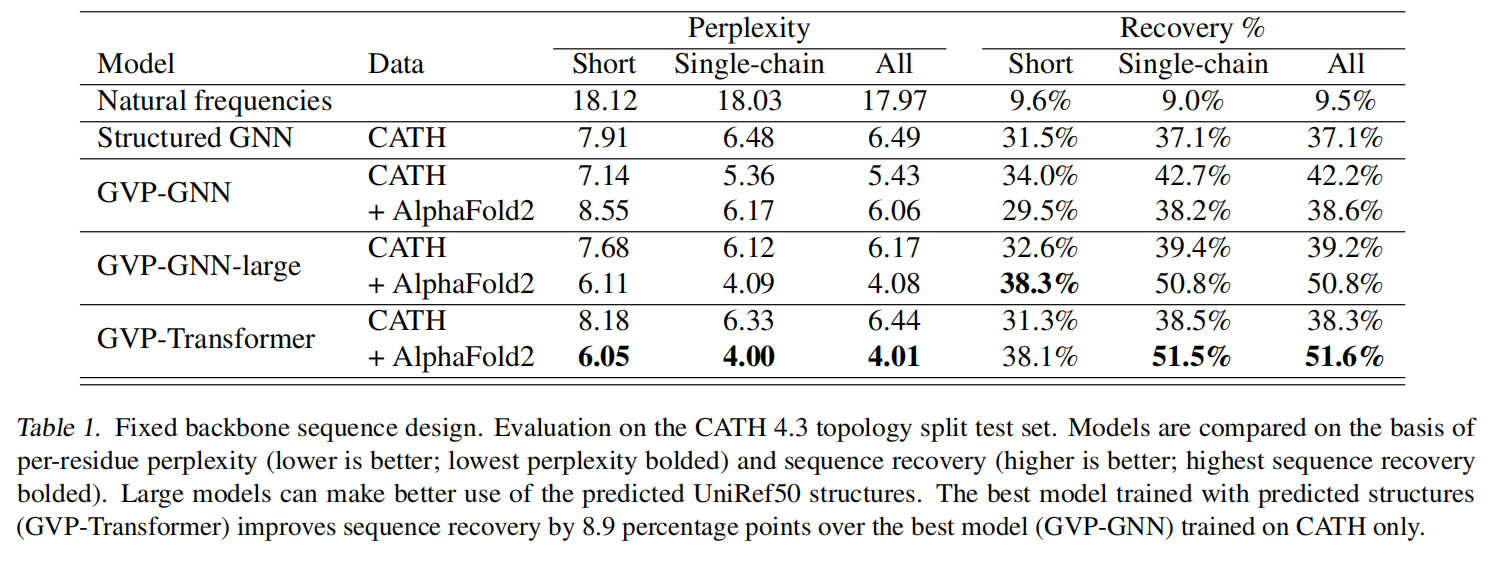
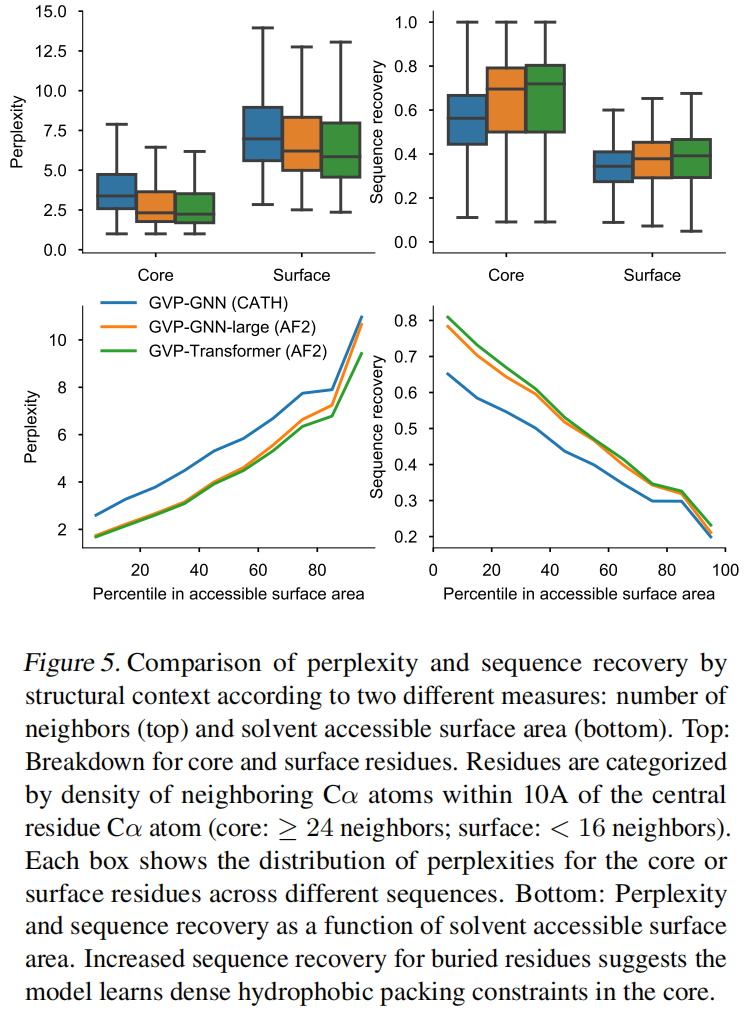
考虑从骨架原子坐标中预测蛋白质序列的任务,使用AlphaFold2预测了12M蛋白质序列得到的结构数据来作为数据增强,在带有invariant geometric输入层的transformer得到了51% native sequence恢复率,对buried residues恢复为72%
Introduction
设计编码具有所需特性的蛋白质的新型氨基酸序列,称为 de novo protein design
这里主要探索预测出来的结构能否克服实验数据量的局限性,所以使用AlphaFold2预测UniRef50里12m的序列,即使预测的输入(即结构)质量较低,反翻译也能有效地从额外的目标数据(即序列)中学习
Learning inverse folding from predicted structures
给定骨架原子N,Cα,C原子坐标 $(x_1,…,x_i,…,x_{3n})$,目的预测蛋白质序列序列 $(y_1,…,y_i,…,y_n)$,使用自回归编码架构进行建模
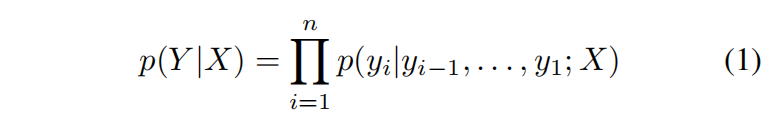
Data
Predicted structures
为了选择用来结构预测的序列,先用MSA Transformer对所有UniRef50序列的MSAs预测distograms,然后用distogram LDDT分数来代表预测质量进行排序,选取top 12m不超过500个氨基酸的输入AlphaFold2
Traning and evaluation data
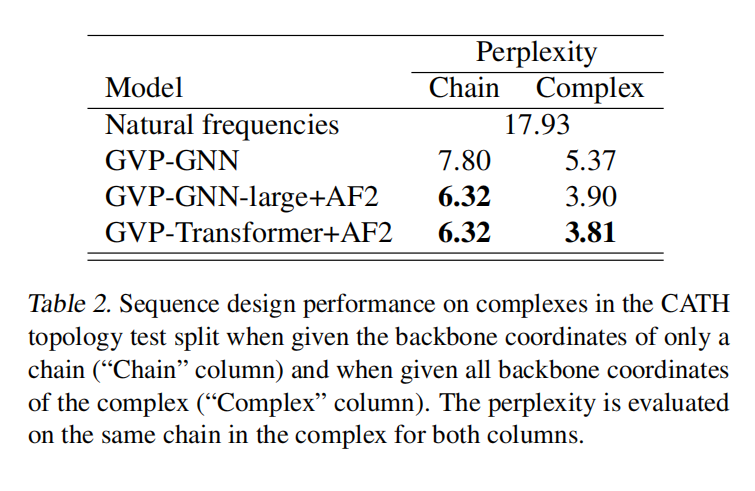
在CATH的一个结构保留子集上测试模型,在topology层划分80/10/10,16153训练,1457验证,1797测试。使用Gene3D topology classification过滤了使用在训练集作为有监督的序列,以及用于AlphaFlod2预测的MSA的输入
Model architectures
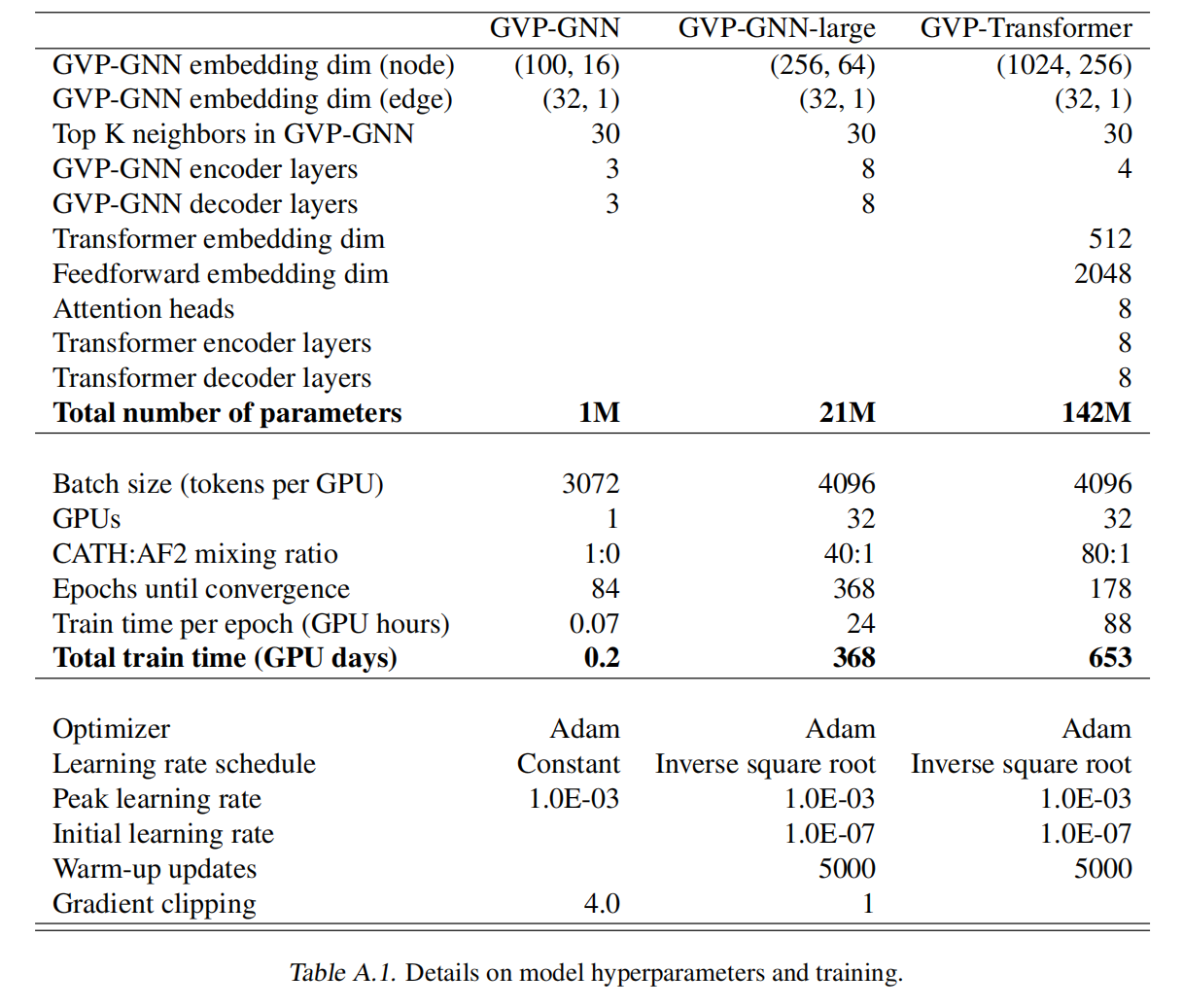
使用GVP学习向量特征的旋转等变变换和标量特征的旋转不变变换,展示了三种模型的效果:GVP-GNN,GVP-GNN-large以及GVP-Transformer
在反向折叠中,预测序列应该独立于结构坐标的参考框架。 对于输入坐标的任何旋转和平移 T,希望模型的输出在这些变换下不变,即 p(Y |X) = p(Y |T X)
GVP-GNN
3层encoder和3层decoder
GVP-GNN的蛋白质结构表示为图,节点特征是从二面角导出的标量节点特征和从骨架原子的相对位置导出的矢量节点特征的组合,而边缘特征捕获附近氨基酸的相对位置
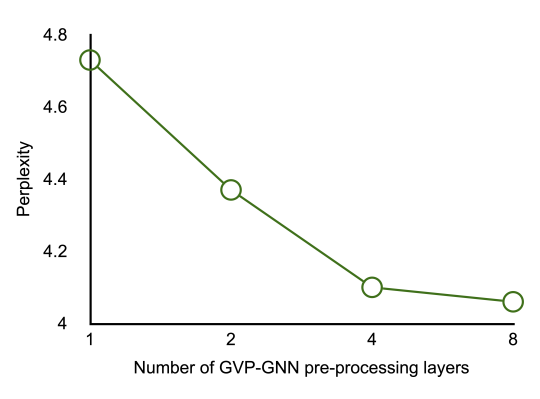
训练的时候发现更深更宽的8层GVP-GNN效果更好
GVP-Transformer
使用GVP-GNN encoder提取几何特征,然后跟上一个一般的自回归transformer模型
Training
Combining experimental and predicted data
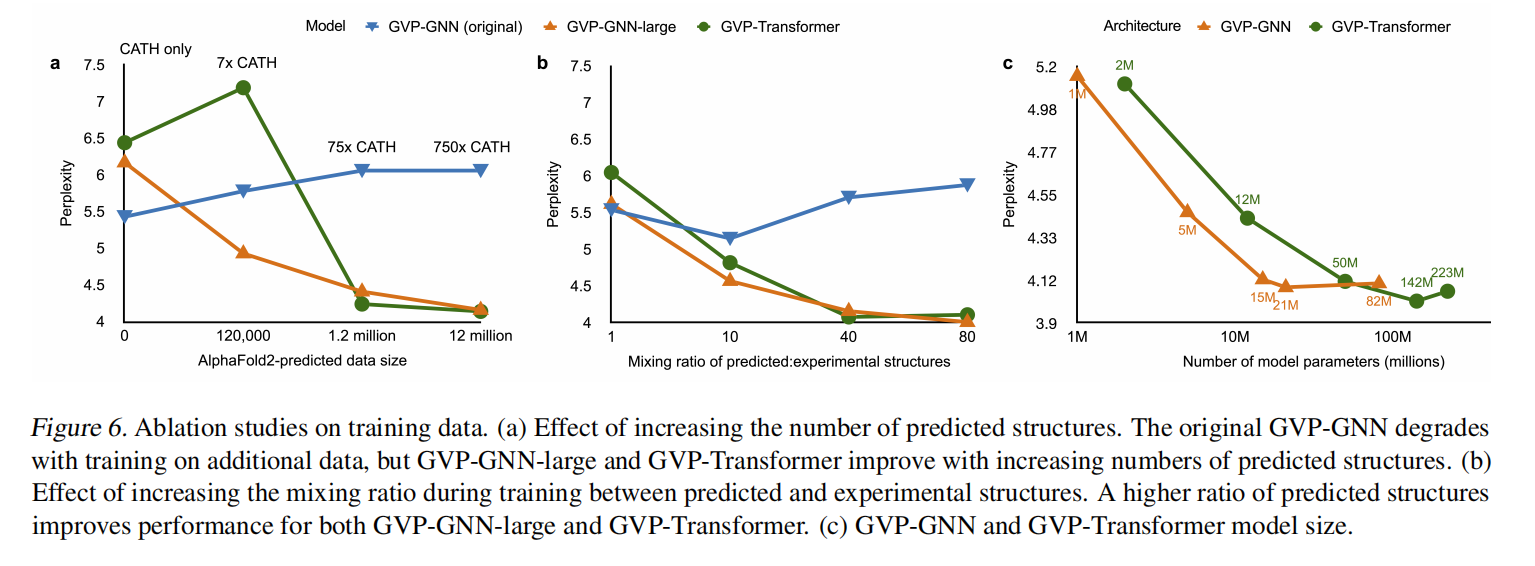
训练每个epoch的时候混合了实验得到的结构(16K),以及10%随机采样12M数据,大概是1:80 实验:预测。对于更大的模型更高的预测数据比例有利于防止过拟合如图6b,但只在预测的数据上训练效果会变差
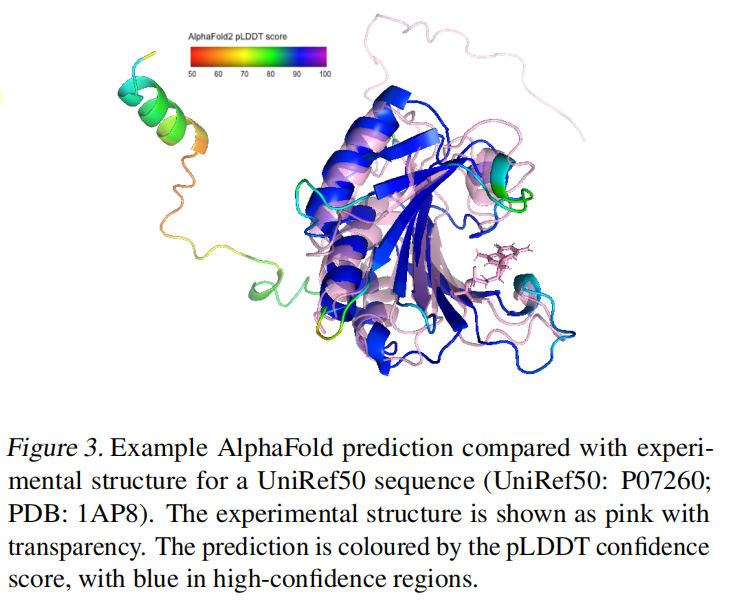
掩盖了使用AlphaFold2置信度评分(pLDDT)低于90的预测输入坐标,约为预测坐标的25%,如图3是可视化;以及在每个序列前定义了一个Token指示这个结构是实验还是预测;对于每个残差,提供了来自AlphaFold2的pLDDT置信度值,作为一个由高斯径向基函数编码的特征,效果略好如图C.1
Span masking
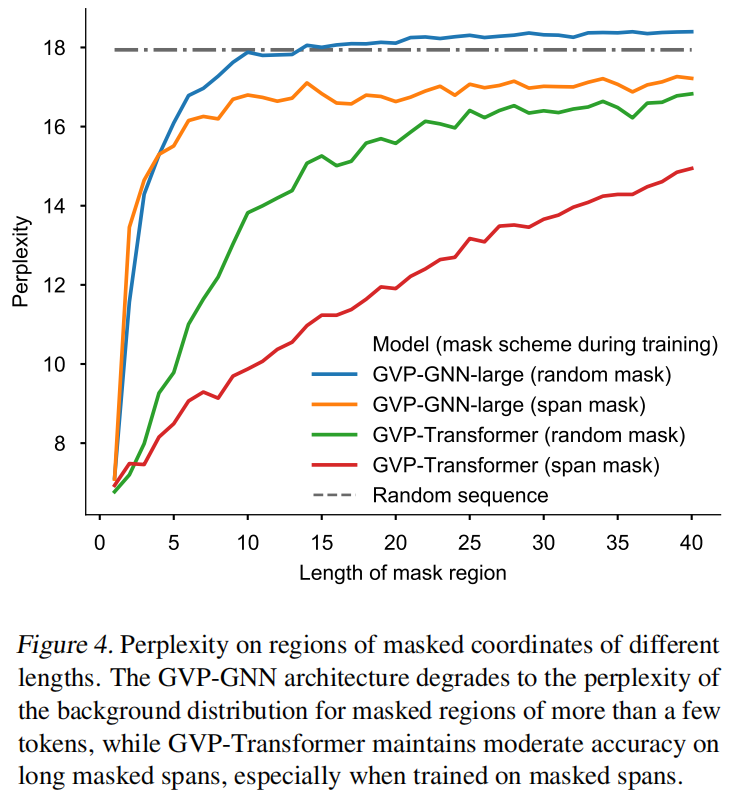
随机选择30个氨基酸知道是整个输入骨架的15%做mask
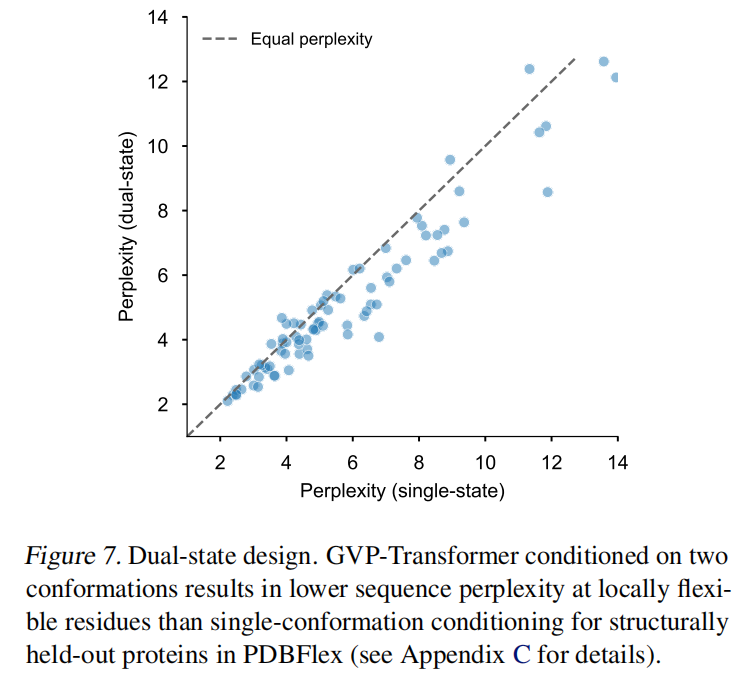
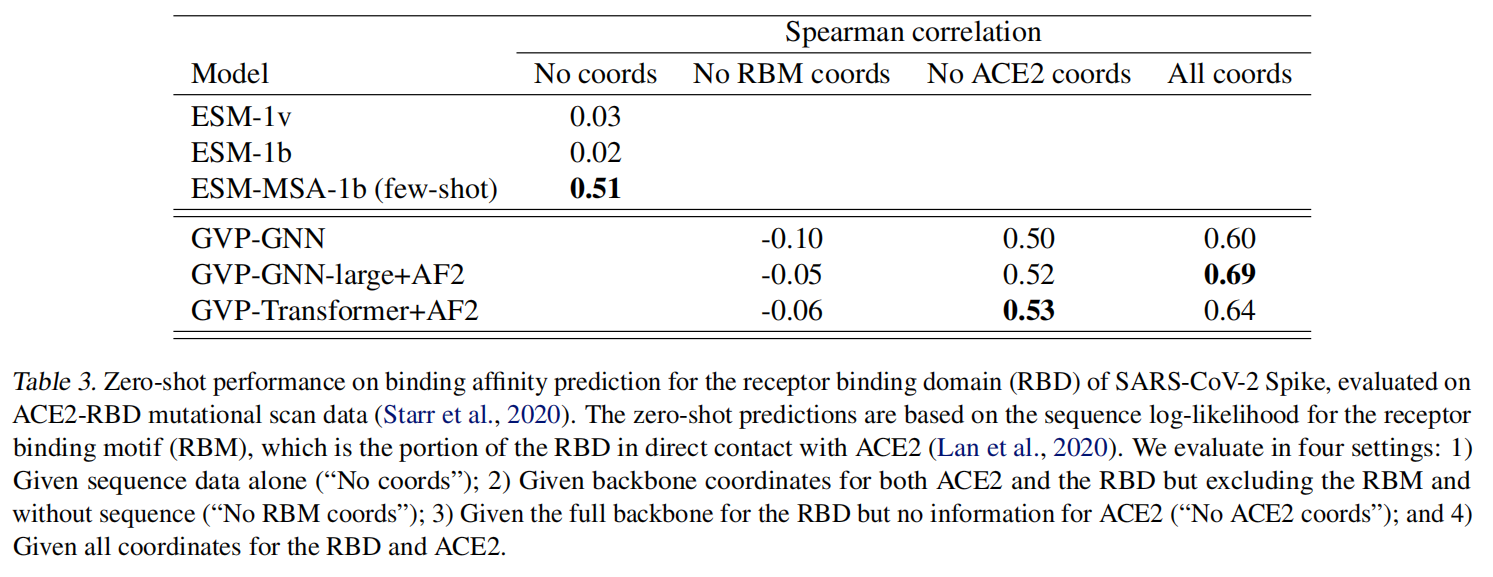
Results