论文地址:Language models enable zero-shot prediction of the effects of mutations on protein function
ESM-1v:zero-shot做蛋白质突变
Abstract
进化信息已经编码在了蛋白质序列里面,无监督模型可以从序列种学习到变体的影响。目前的方法都是来拟合一个家族的序列,这对每个预测任务都要做新模型,非常局限,所以本文提出了一个zero-shot模型
Introduction
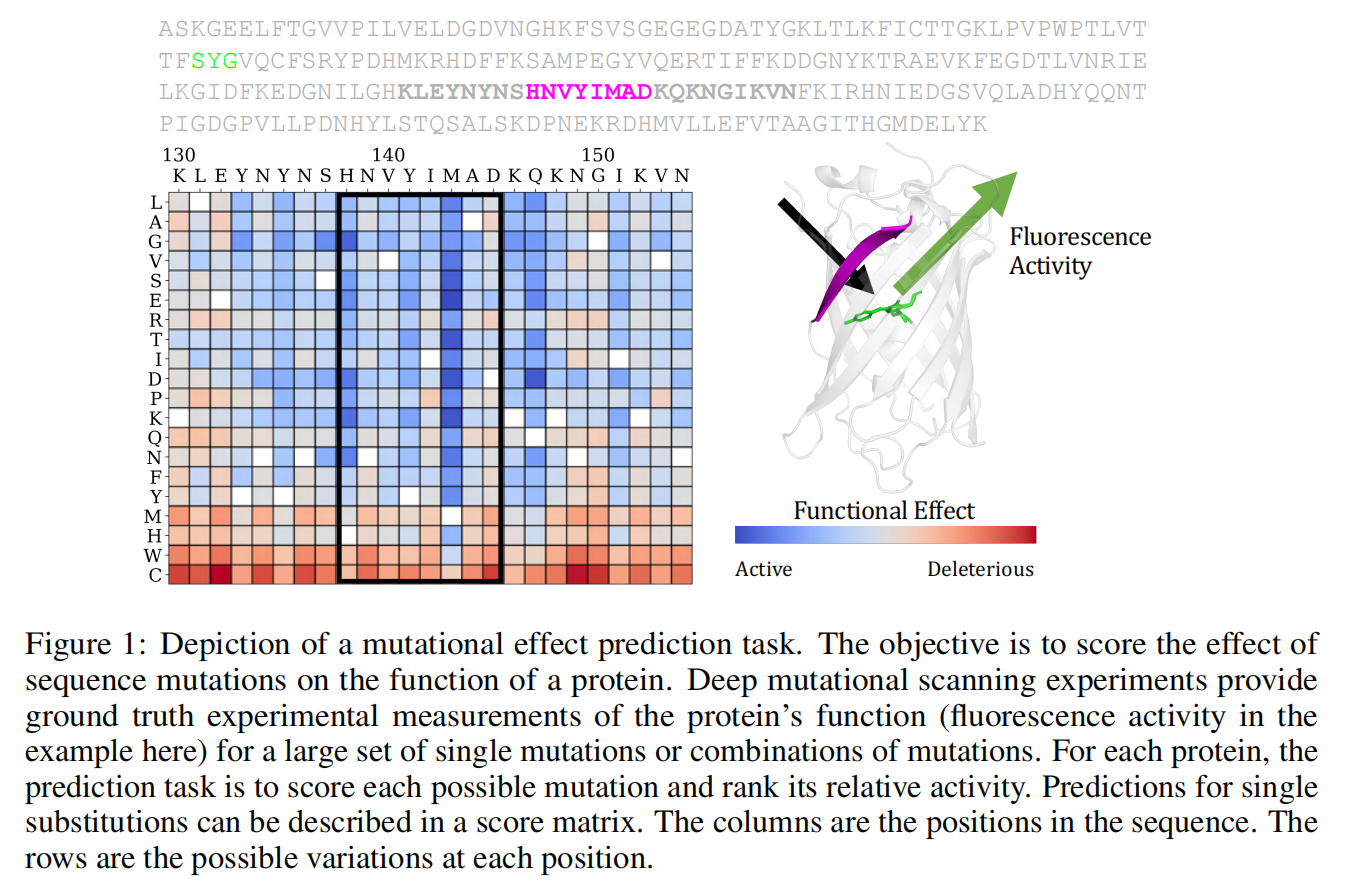
序列变异的功能效应可以通过深度突变扫描(deep mutational scanning)实验来测量
提出无监督模型esm1v
Zero-shot transfer
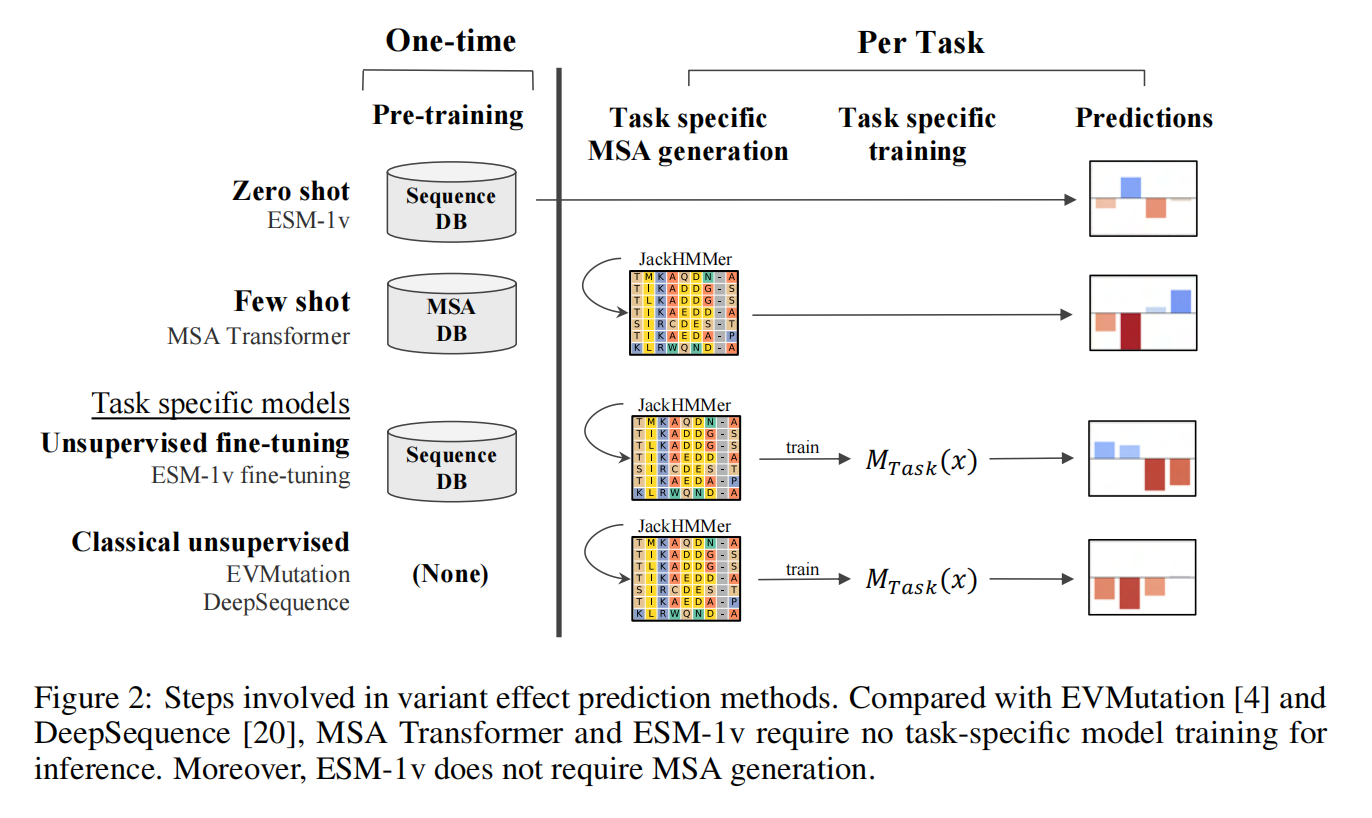
zero-shot要求模型在预训练阶段就学习到之后的具体任务所需的一切信息。本文中使蛋白质语言模型具有zero-shot预测能力的机制是,采用含有海量进化信息的蛋白数据库进行预训练。当所用数据库涵盖的序列足够多、足够多样(large and diverse),那么模型就有可能从数据库中学到横跨整个进化树的序列模式,那么该模型也就很可能会在预训练阶段学习到它将要应用的家族的序列模式,迁移应用时也就无需再额外训练
Method
用氨基酸的保守性衡量变异的影响力,若某位置与野生型相比其他氨基酸出现的概率很低,说明蛋白序列在该位点很保守,变异可能性很低,也就说明该位点的氨基酸可能对蛋白的结构和功能有重要影响
预测序列变异对蛋白功能的影响的方法:为每个变异对功能的影响打分来衡量其影响,比较某个被遮盖位置(masked)变异成某个氨基酸的概率和该位置变异成野生型蛋白的概率,求它们的log-odds,即求它们概率的比值再取对数
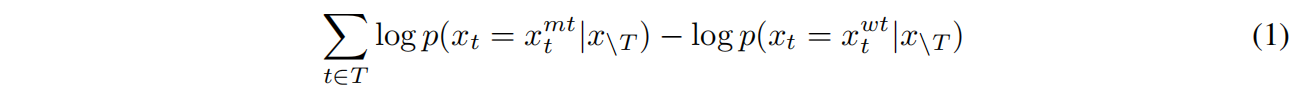
ESM-1v模型采用与ESM-1b相同的结构
-
训练方法:masked training
-
训练数据:UR90数据库,含有98million条多样的蛋白序列,数据量和数据多样性都远远大于ESM-1b和MSA Transformer训练所用的UR50数据库,因此训练出的模型迁移能力更强
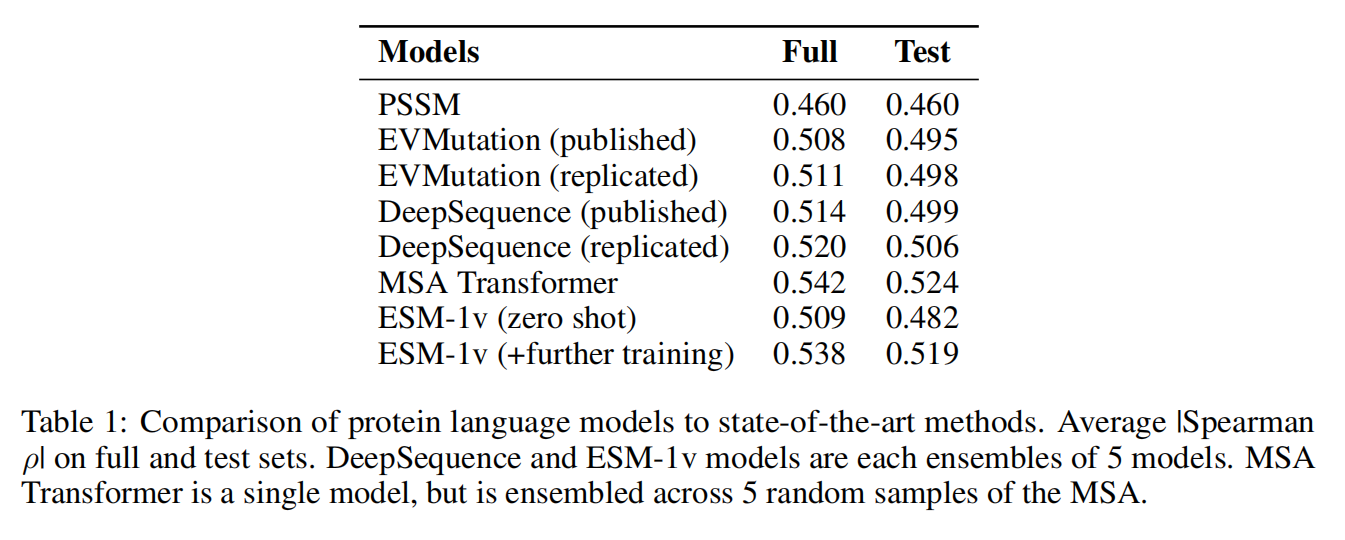
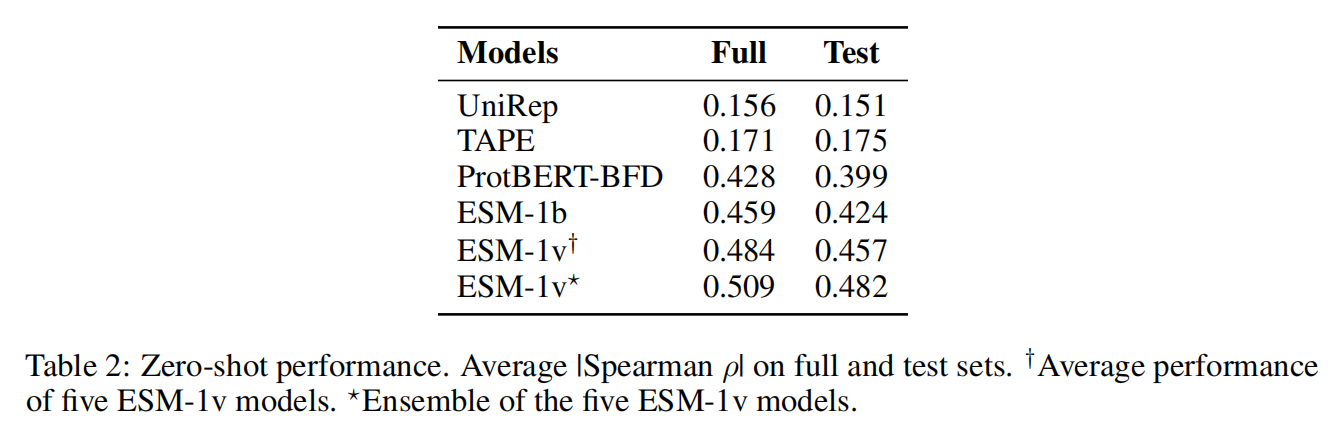
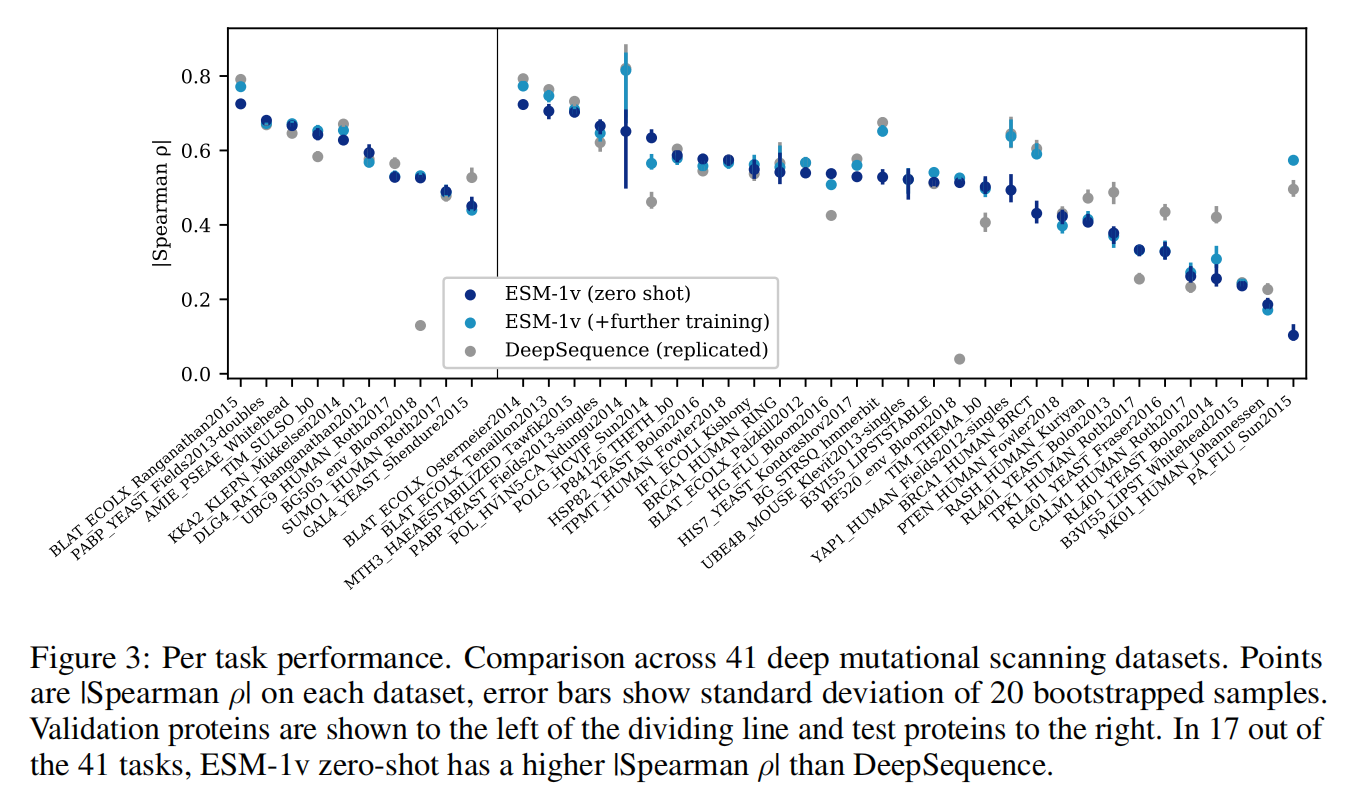
Result