论文地址:Embeddings from protein language models predict conservation and variant effects
VESPA:保守性预测+BLOSUM+PLM embedding
Abstract
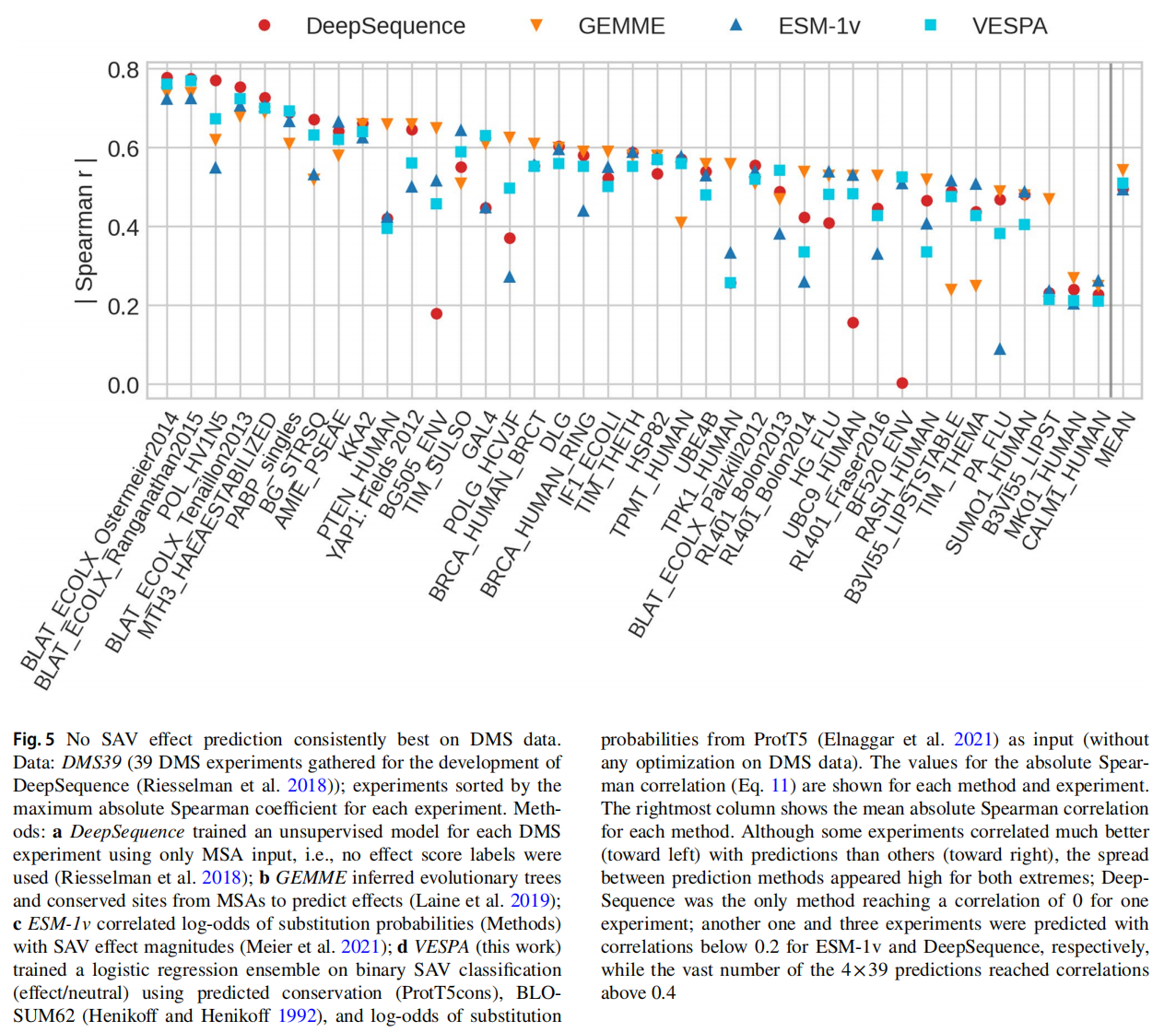
SAVs(single amino acid variants)。本文使用蛋白质语言模型表征来预测序列保守性和SAV影响,而没有多重序列比对(MSAs)。将保守性预测,BLOSUM62矩阵喝plm mask重建概率输入到LR ensemble中来做variant effect score prediction任务,不需要在DMS数据上进一步优化。在一个标准的39个DMS数据集上进行比较,没有一个方法是完全超过了其他方法。最后本文研究了四种人类蛋白的DMS实验中的binary effect prediction
Introduction
即使在现有的实验中,仍存在内在问题:
- 在 vitro DMS 数据可以很好地捕捉到基于分子功能的突变效应,但对生物过程(例如疾病的发生)的影响则不够敏感。例如,虽然在线数据库如 OMIM 中包含了与疾病相关的突变信息,但在 MaveDB 中则不包含。
- 大多数蛋白质具有多个结构域,因此它们很可能具有多种不同的分子功能。然而,每个实验方法往往只能测量对其中一种功能的影响。
- 在 vivo 中,蛋白质功能可能会受到多种影响,这些影响可能无法通过 vitro 实验复制。
换句话说,虽然 vitro 实验可以提供有价值的信息,但我们仍需要谨慎解释实验结果,特别是在探索与疾病相关的突变时。我们需要结合不同的实验方法和生物信息学工具,以全面理解蛋白质的结构和功能,同时需要结合 vivo 实验来更好地模拟真实生物过程
分析了使用DMS(Esposito等人2019年)和PMD(Kappata等人1999年)的实验数据,使用预先训练的pLMs嵌入来预测sav对蛋白质功能的影响,重点是分子功能的影响
来自预训练的plm的embedding不变,然后再在有注释的数据集第二步监督训练。评估了两个独立的监督预测任务:conservation和SAV effects
先使用plm作为静态的特征编码器,然后训练一个LR来预测SAV,还集成使用了最好的保守预测器(ProtT5cons)和替换分数BLOSUM62和ProtT5的替换概率。其实这里的替换概率已经和DMS scores相关了,但把另外两个东西结合起来可以提升效果
Methods
Data sets
总的来说使用了五个数据集,ConSurf10k用来训练和评估残基保守性预测,Eff10k用来训练SAV effect prediction,PMD4k和DMS4是测试集评估二元SAV effects,回归的effect score用DMS39评估
ConSurf10k assessed conservation.
ConSurf-DB有89,673条蛋白,经过一致性,分辨率,残基数一系列清洗得到10,507条蛋白
Eff10k assessed SAV effects
SNAP2 development set有100,737条二元SAV-effect annotations(neutral: 38,700, effect: 61,037),来源于9594条蛋白质
把蛋白是一个cluster的放在相同的CV(cross-validation)分割里
PMD4k assessed binary SAV effects
从PMD提取注释(no change是neutral,功能增加或减少视为effect),得到51,817条二分类SAV(neutral: 13,638, effect: 38,179),来源于4061条蛋白质
DMS4 sampled large-scale DMS in vitro experiments annotating binary SAV efects
从四条人类蛋白质提取的二元分类数据集,在数据集分布均值95%视为neural,尾端的5%视为effect,可以调整这个比例
DMS39 collected DMS experiments annotating continuous SAV efects
DeepSequence的43DMS实验的子集
Input features
plm的embedding,替换概率和BLOSUM62的替换分数
Method development
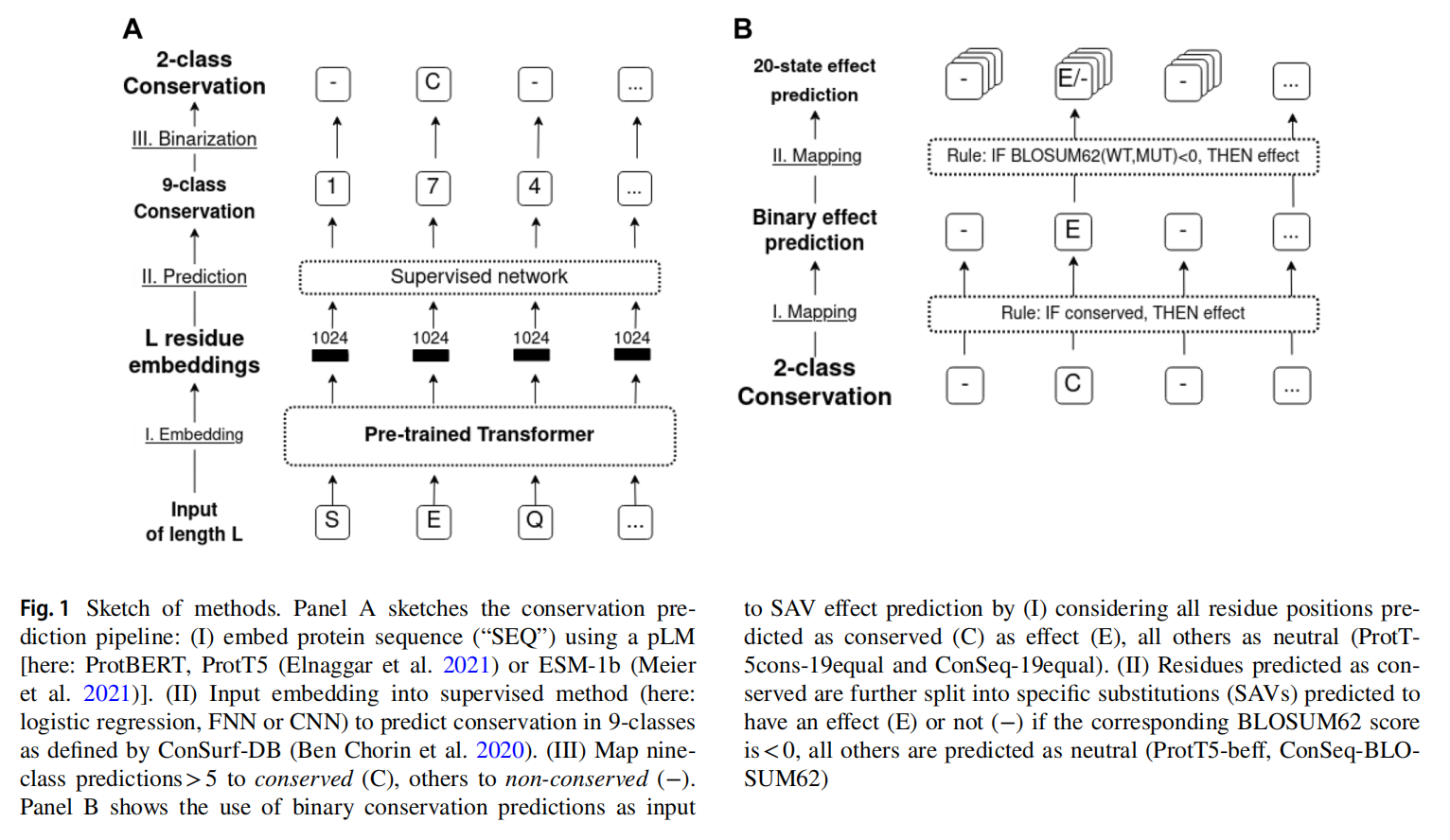
Conservation prediction
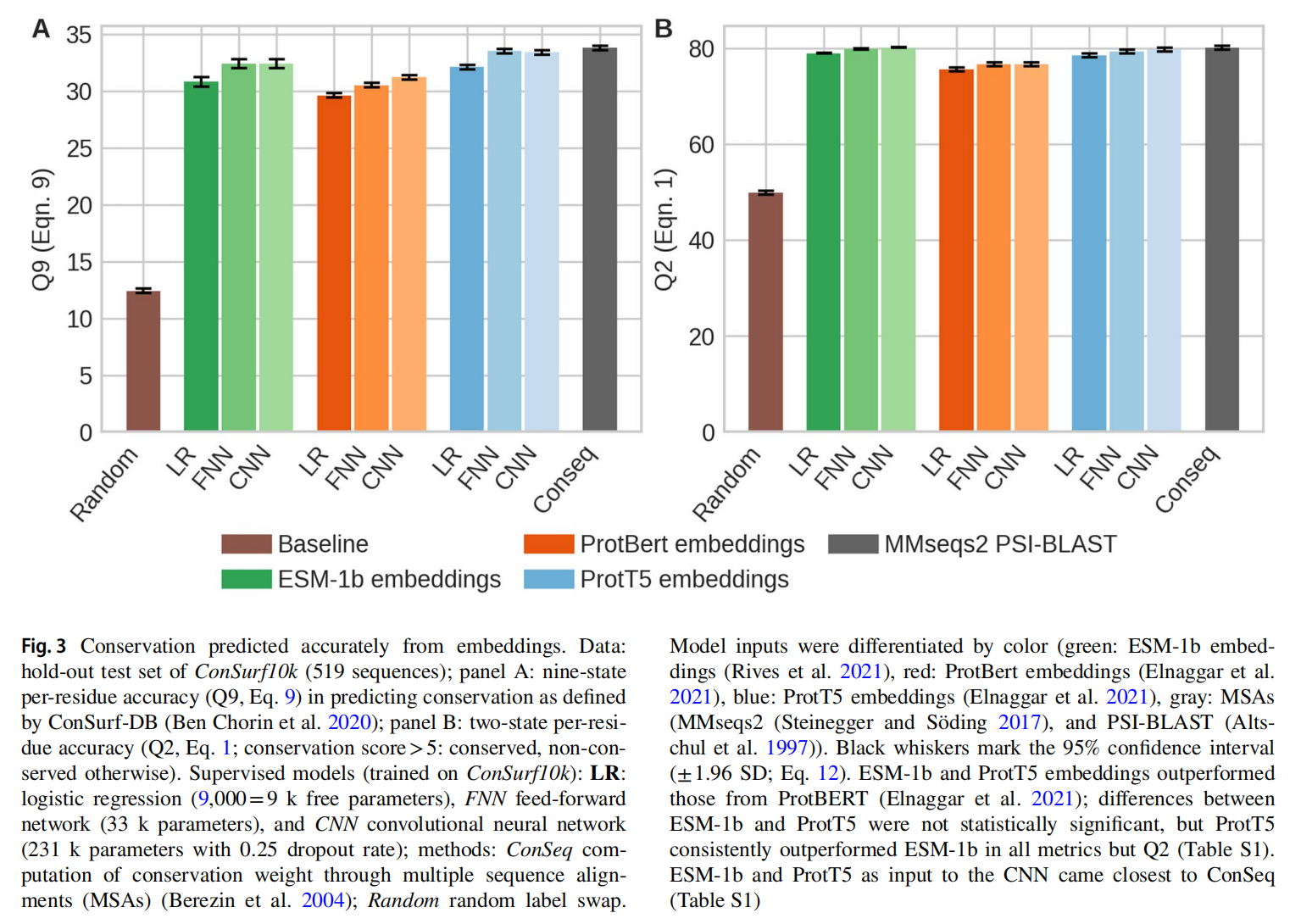
使用plm模型embedding作为输入,训练三个有监督分类器。先是9分类的保守性预测,测试了几个模型:1.LR;2.两层FFN+RELU;3.两层CNN,window size=7+RELU
最好的模型是在ProtT5上训练的CNN称为ProtT5cons
Relu-based binary SAV effect prediction
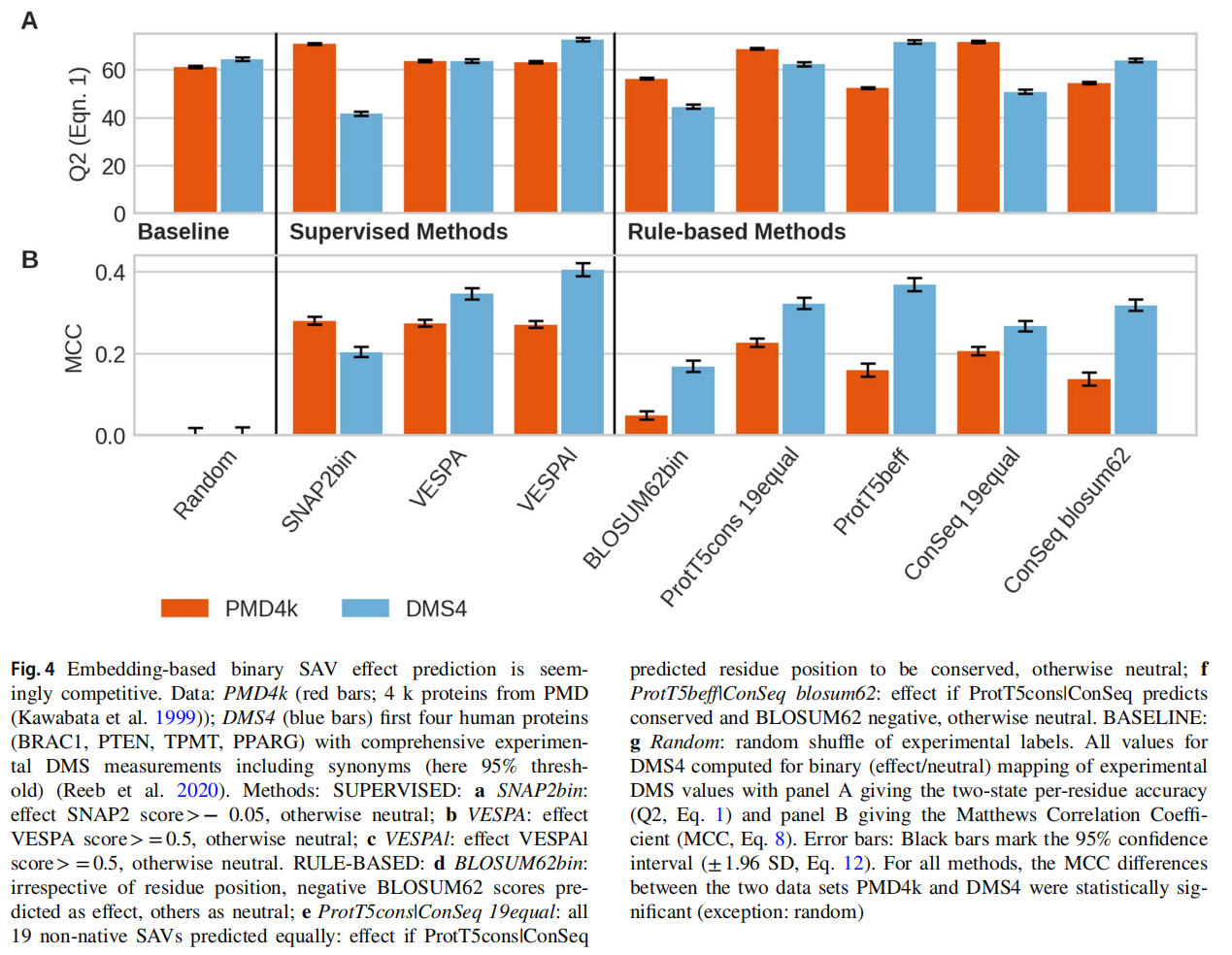
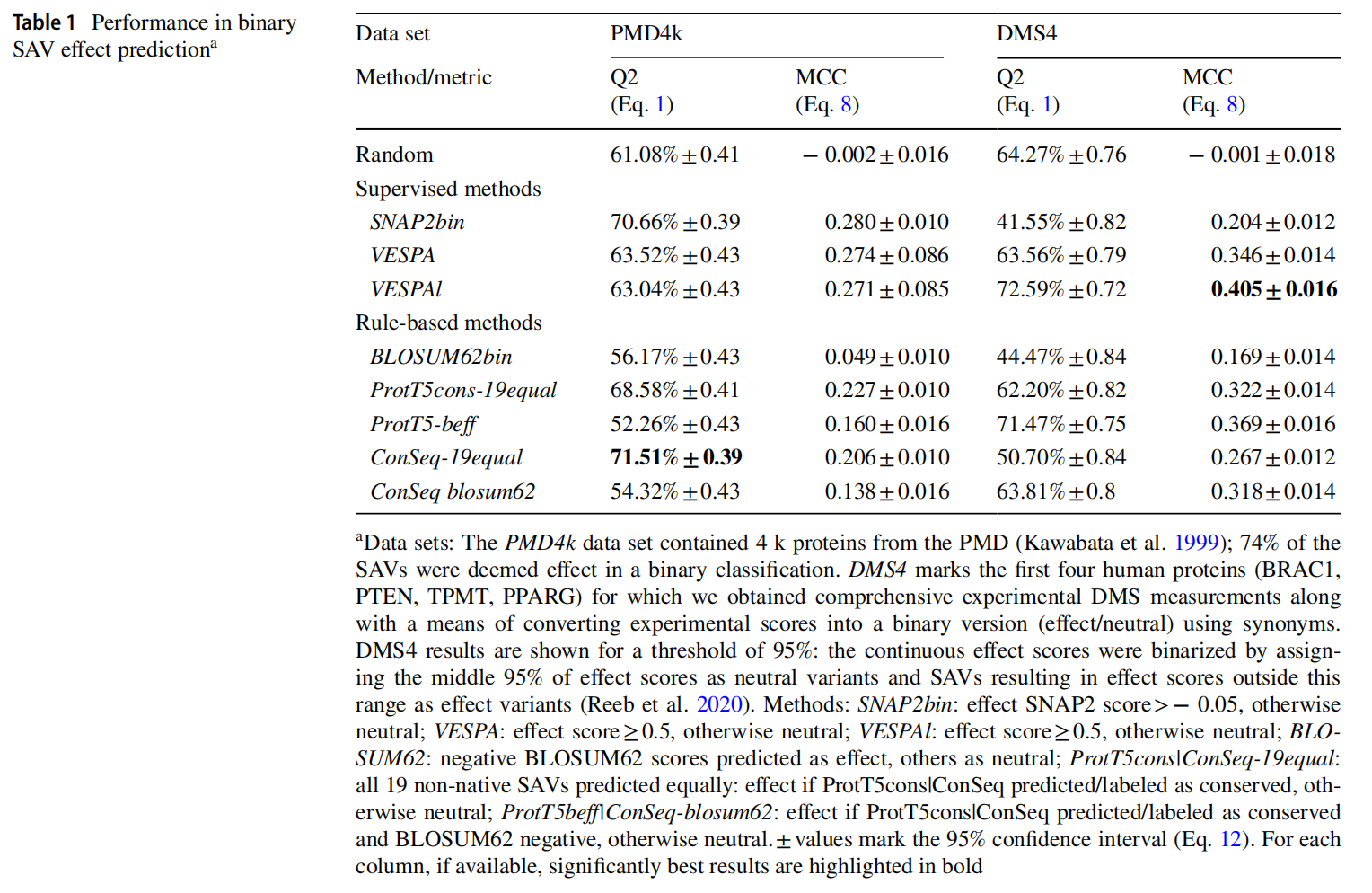
该方法首先使用了一个简单的阈值方法,将ProtT5cons输出的保守性评分(conservation score)大于5的残基标记为“effect”,其他标记为“neutral”。然后,该方法结合了SIFT中使用的BLOSUM62替代得分,对预测结果进行了优化。这个优化方法被称为BLOSUM62bin,被认为是一种简单直观的基线方法。在该方法中,那些比预期更不可能发生突变(在BLOSUM62中具有负值)的突变被标记为“effect”,其他被标记为“neutral”。
最后,将两个基于规则的分类器进行结合,形成了第三种方法ProtT5bef。在这个方法中,如果ProtT5cons预测的保守性评分大于5且BLOSUM62替代得分为负,则标记为“effect”,否则标记为“neutral”。这种方法将来自ProtT5cons和BLOSUM62的位置感知信息和变异感知信息进行了合并,预测了突变的二进制分类(effect/neutral)结果,而不需要使用任何实验数据对突变效应进行优化
Supervised prediction of SAV effect scores
训练了一个balanced LR 集成模型,使用的SciKit里的交叉验证,在Eff10k数据集上
VESPA<0.5是neutral,>=0.5是effect
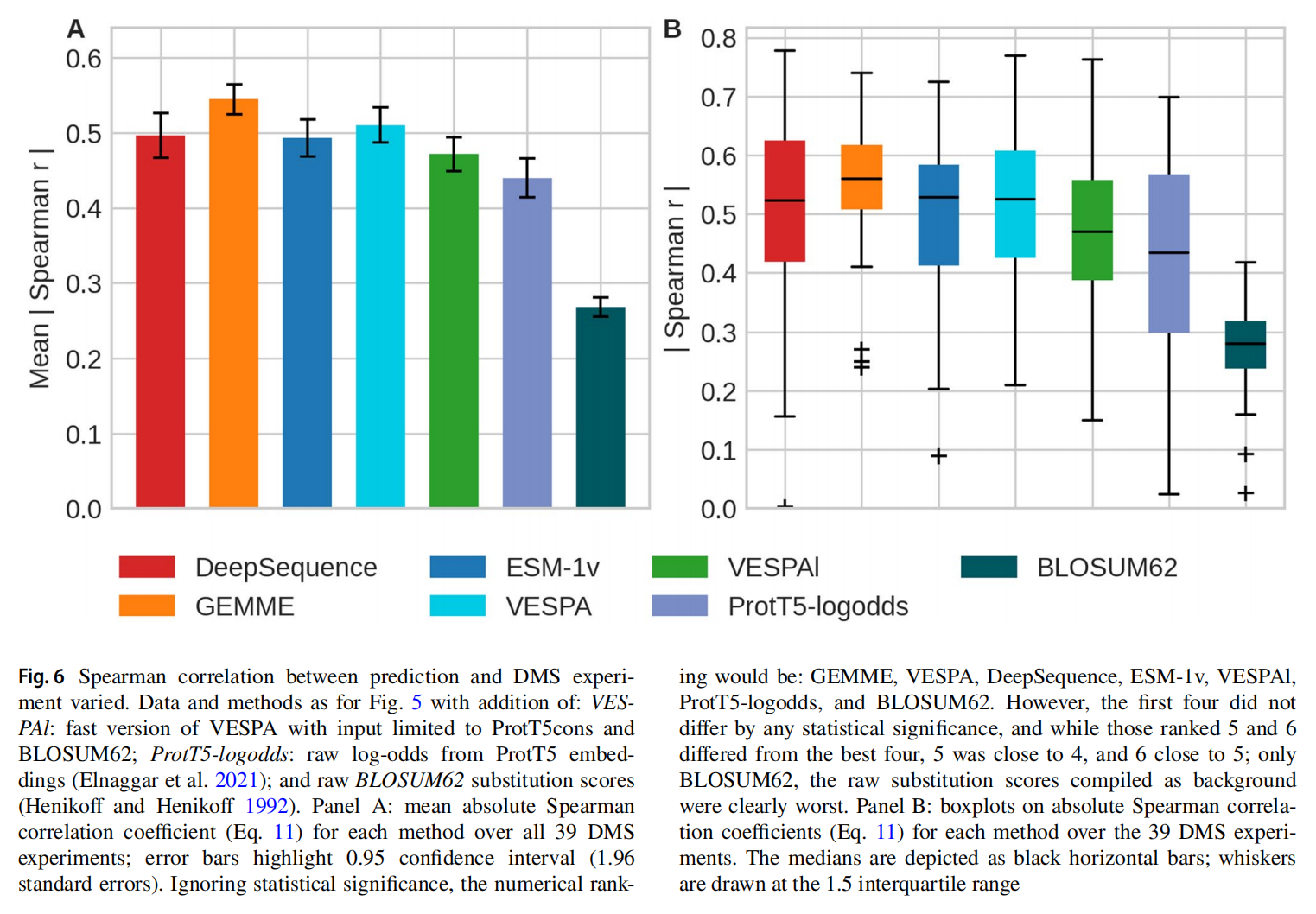
Results