论文地址:Ankh:Optimized Protein Language Model Unlocks General-Purpose Modelling
Ankh:蛋白质语言模型调参
Abstract
随着蛋白质语言模型的进步,寻求针对蛋白质特性的优化方法来提高性能。虽然语言模型的规模和表征的丰富已经得到了验证,但还是在寻求一种数据高效,开销减少,知识驱动的优化方法,通过从msaking,架构,预训练数据设计了超过20个实验,从protein-specific的实验来融入Insight,构建解释语言模型。提出了Ankh,一个通用目的的PLM,在谷歌TPU-v4上训练,以更少的参数取得了SOTA(<10%预训练,<7%推理,<30%嵌入维度)。在机构和功能的Benchmark上进行了测试,进一步在high-N和OneN输入数据尺度上提供了蛋白质变异生成分析,其中Ankh成功地学习了蛋白质的进化保守-突变趋势,并引入了功能多样性,同时保留了关键的结构-功能特征
Introduction
通常情况下,人们倾向于认为模型的大小(参数量)与其学习的表示能力和性能之间存在正相关关系,即模型越大,其学习的表示越丰富。然而,这种观点被指出是具有误导性的。其中一个误导源自对大规模参数的语言模型进行观察。这些模型经过大量的参数训练和训练步骤,但仍然表现出明显的学习梯度,被认为是欠拟合
这种情况下,观察到的模型尽管具有大量的参数,但其表现并不如预期的那样良好。这可能表明模型的规模不是决定其学习能力和表示丰富性的唯一因素。其他因素,例如数据质量、训练方法、学习率等,都可能对模型的性能和拟合程度产生影响
大模型好,但是也增加了研究创新的门槛和限制了可扩展性,然而追求大也不是唯一的方向,比如大的数据集其实比不上一个小的高质量数据集
Ankn在两类实验上验证,一个是在结构和功能的benchmark上取得了SOTA,以及在hign-N(family based)和One-N(single sequence-based)的蛋白质工程上进行了分析;其次是民意调查模型性能,优化模型设计,开发和部署等
Results
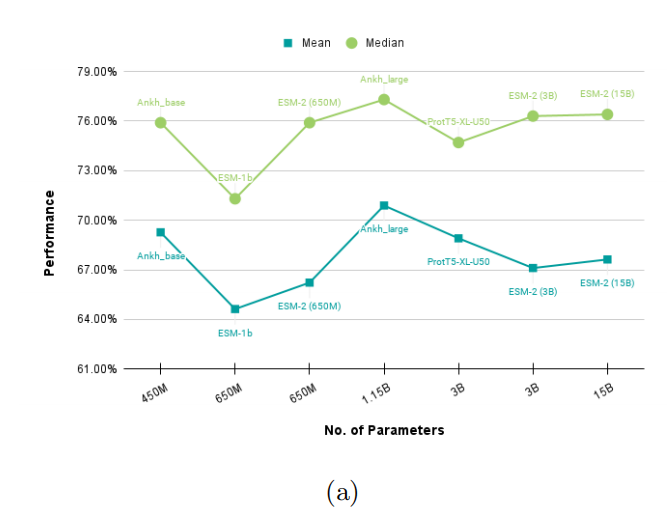
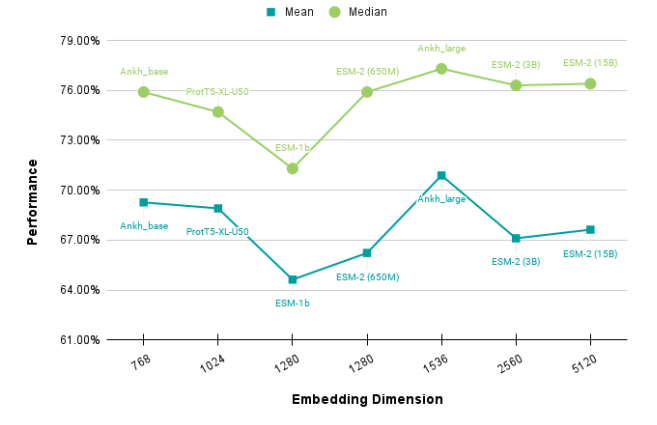
Utmost Computational Efficiency and Utmost Performance
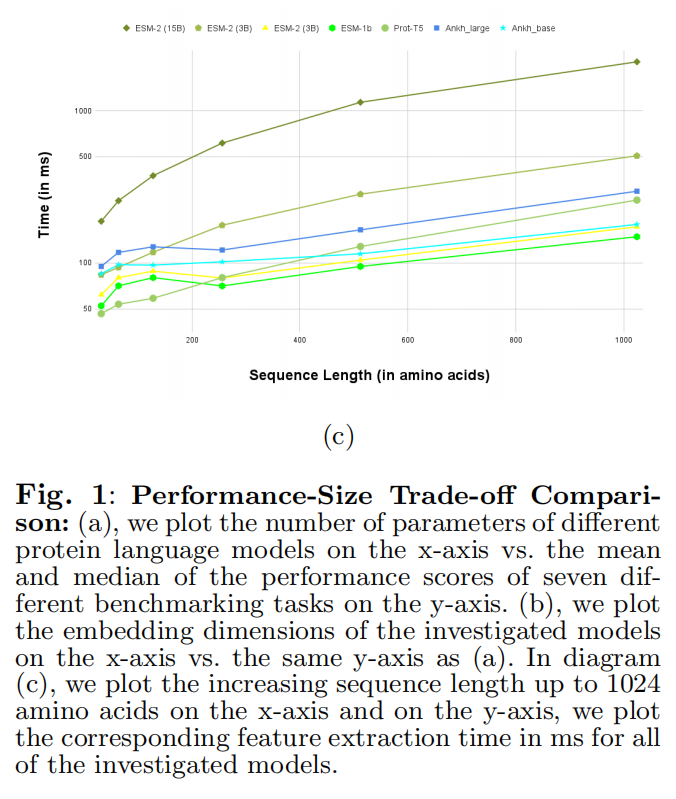
Protein Generation Made Attainable
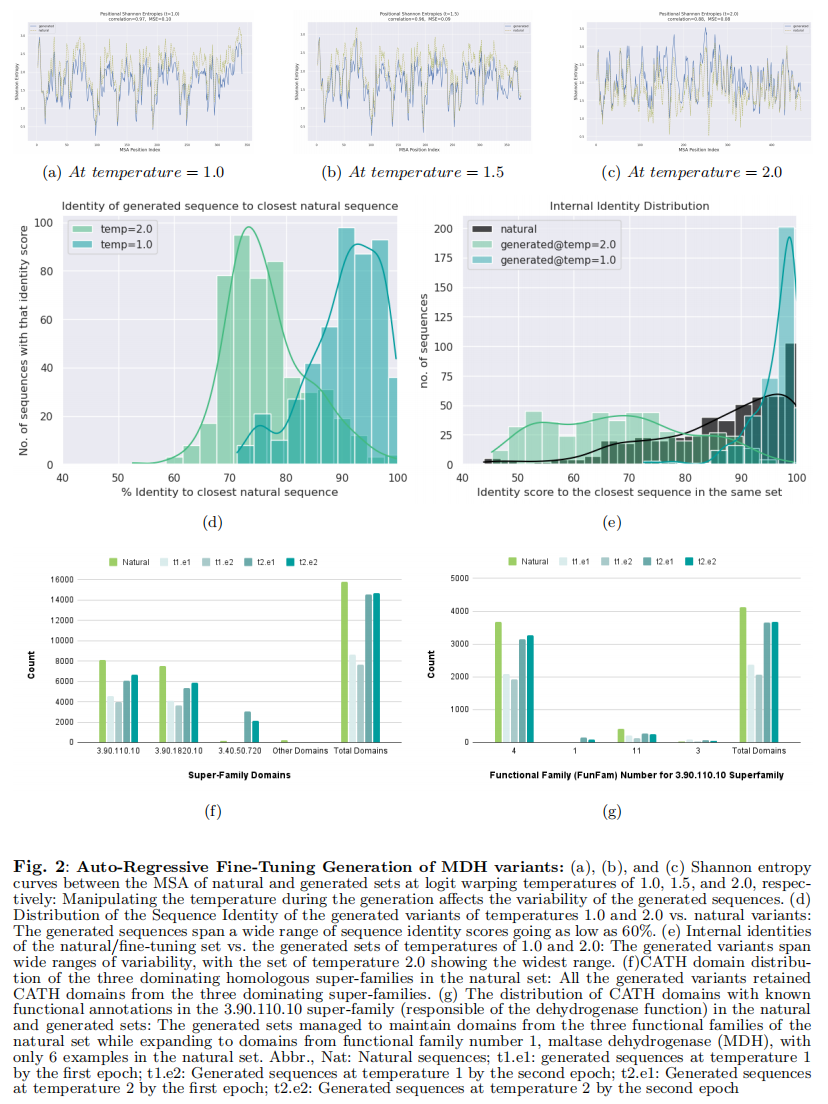
Auto-Regressive Fine-Tuning for High-N Generation
用超参数t来控制生成结果的多样性,高的t会给不同候选回复都有较高的概率,低的t会给最可能的回复较高的概率
用MDH天然蛋白上进行微调,设置了三种t生成每组500条序列,用香农熵的变化来报告微调数据的MSA和三种温度设置的关联,香农熵的低熵值反映了控制保留功能的保守区域,而高熵值反映了突变率较低的保守区域
Masked Language Modeling (MLM) for One-N Generation
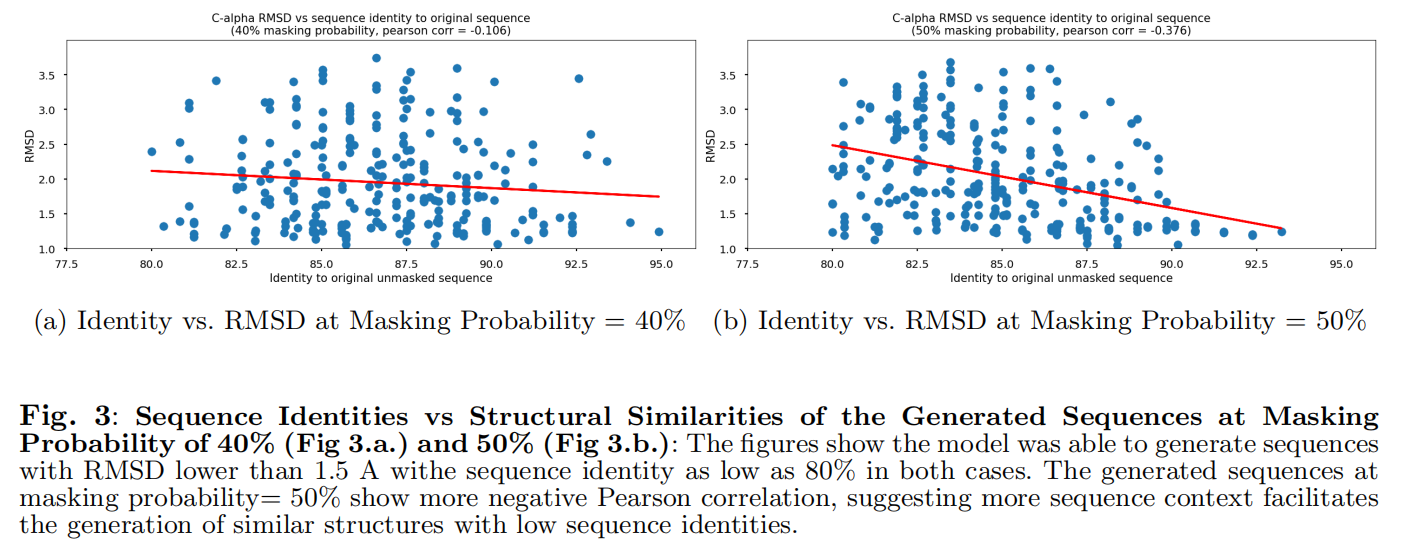
使用colabfold的2个模型和3个周期来预测生成的变体的三维结构,希望使用MLM生成的序列得到的预测结构,和实验的3D结构有较低的序列相似性和RMSD
在两种情况下,该模型都能够生成RMSD低于1.5 A的序列,且序列一致性均低至80%
Knowledge-Guided Optimization in Action
把knowledge-guided experimentation定义为针对蛋白质特定任务进行探索,同时保持单一的独立变量,即遮蔽策略和概率、模型架构以及预训练数据集
- 蛋白质特定实验:这些实验是为了针对蛋白质相关任务进行优化和调整。蛋白质是复杂的生物分子,具有独特的结构和功能特性,因此需要专门的实验来针对其特定需求进行设计。在这种情况下,实验可能涉及不同的遮蔽策略和概率、模型架构以及预训练数据集的选择,以满足蛋白质任务的要求
- 单一独立变量:在进行实验时,通过保持单一独立变量的变化,可以更好地理解不同因素对模型性能的影响。在这里,遮蔽策略和概率、模型架构以及预训练数据集被视为独立变量,通过对它们进行变化和控制,来评估它们对计算效率和下游性能的影响
Discussion
Results Interpertation
在应用序列上下文嵌入时,需根据任务特定的需求来选择和设计适当的架构和层,以获得更好的性能和效果
Results Implications
提出了基于蛋白质知识的优化方法,该方法通过在模型生命周期中对软件和硬件组件进行优化,以提高模型性能,通过这种知识引导的优化方法,可以在显著减少计算资源的情况下达到卓越的性能
通常人们倾向于认为提高模型性能需要更大的模型和更多的数据,但这种相关性并非绝对,其实可以通过有效的优化方法来提高模型的性能,而不是盲目地依赖更大的模型规模或数据规模
Results Limitation
这个工作里汇报了几个局限性,测试改变激活函数,维度观察对优化的影响
因为没有一个单独的模型是在所有任务数值上都SOTA,所以在每个任务上选了表现最好的模型这个版本传递给下一个任务
Recommendations
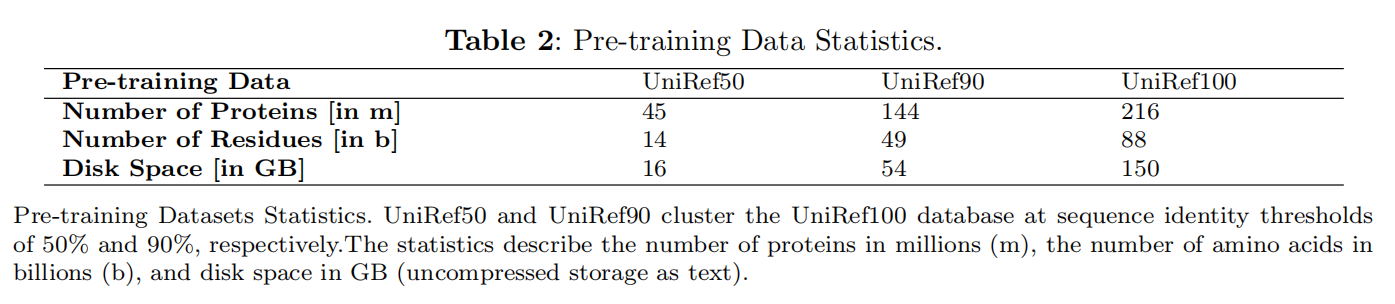
预训练数据集的选择与后续任务测试数据集的协调性,使用UniRef50进行预训练比使用UniRef90、UniRef100和BFD更优的结果,这是由于UniRef50具有较低的冗余性。冗余性的具体定义与应用领域相关(例如,使用所有可用的人类蛋白质在考虑人类蛋白质长度分布时具有较低的冗余性,但在尝试预测这些蛋白质的结合位点时则具有较高的冗余性)
Future Work
Ankh作为优化通用蛋白质语言模型的初始版本。该版本旨在作为预训练基础,将在未来的工作中针对高影响力和高价值的蛋白质建模任务进行专门优化和详细分析(例如,完整的原子分辨率三维结构预测、蛋白质生成等)
Methods
Self-Supervised Learning
Pre-training Datasets
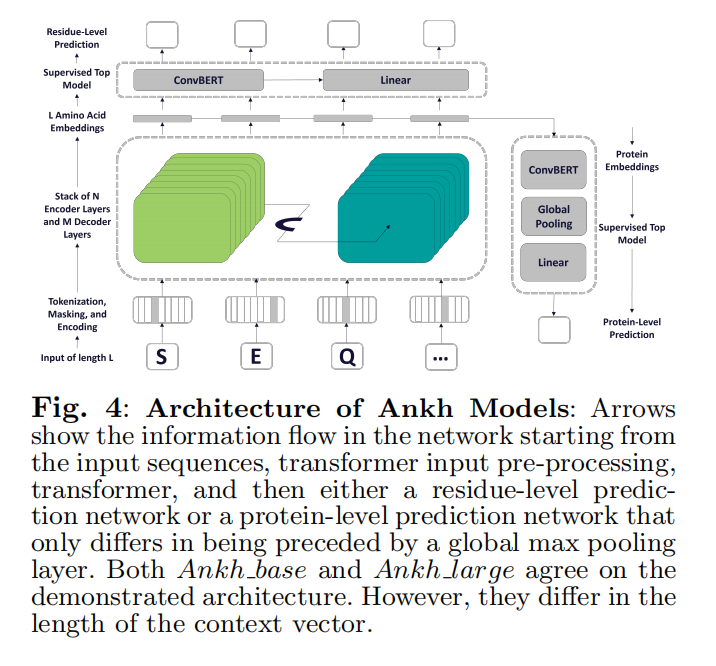
Pre-trained Model Encoder-Decoder Transformer
选择encoder-decoder的架构不仅是性能做好,而且和实验自变量兼容比如mask和architecture
Downstream Tasks

Tasks and Datasets
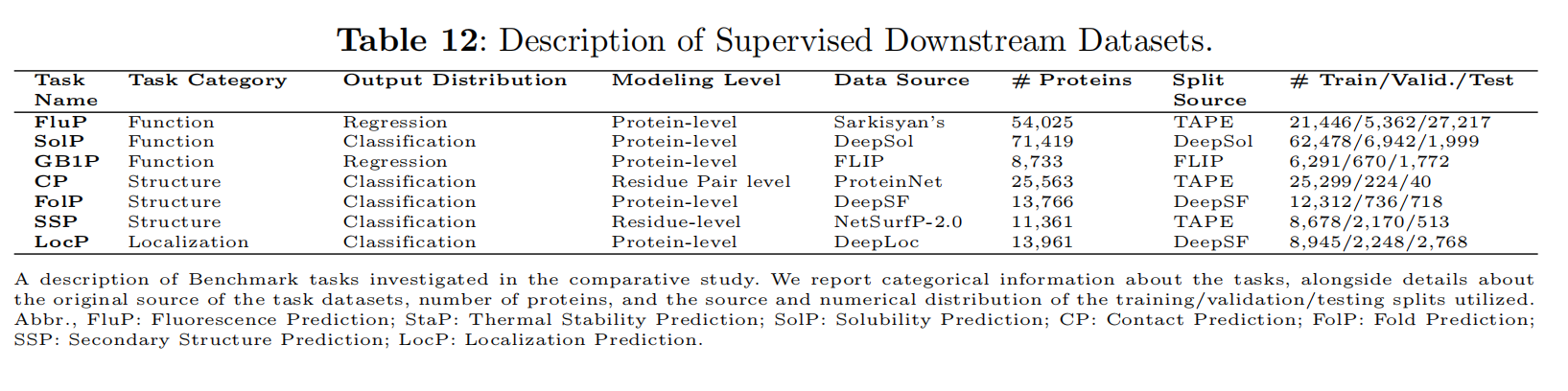
Protein Function Prediction
- Fluorescence Prediction (FluP):回归任务评估绿色荧光蛋白的荧光强度
- Solubility Prediction (SolP):分类任务评估一组不同的蛋白质的二进制标签是否具有可溶性
- GB1 Fitness Prediction (GB1):回归任务评估在FLIP基准中管理的4个特定位置的突变后GB1结合的适应度
Protein Structure Prediction
- Contact Prediction (CP):二分类任务,即预测蛋白质中的残基对是否在其三维结构中相互接触(通常使用8Å的距离阈值进行定义)
- Fold Prediction (FolP):分类任务,将完整的蛋白质序列分类到1194种可能的蛋白质折叠结构中
- Secondary Structure Prediction (SSP):分类任务,蛋白质中的每个氨基酸残基被分类到其二级结构折叠中,有两个难度级别:3类和8类。二级结构包含有关功能域的重要信息,并且通常被用于通过多序列比对来捕捉进化信息
- Embedding-based Annotation Transfer (EAT):传统上,蛋白质注释的转移是通过基于同源性的推断(Homology-based inference,HBI)在序列空间中进行的,从已标记(实验注释)的蛋白质向未标记的蛋白质进行转移。现在可以做基于嵌入的注释转移方法
Protein Localization Prediction
- Sub-cellular localization prediction (LocP):分类任务评估一个蛋白质的定位为10个亚细胞类
Generation of Novel Protein Sequences
- High-N (Family-Based Variant Generation):选择 malate dehydrogenase (MDH)
- One-Shot (Single Sequence Variant Generation):单链SARS-Cov-2纳米抗体(nanobody),该纳米抗体在2022年6月之后被添加到CoV-AbDab数据集中。这样做是为了确保生成的序列是模型在无监督训练中没有见过的新序列。研究进行了七个独立的虚拟生成实验,使用了七个不同的纳米抗体(Nb-007、F6、Nb 1-23、Nb 1-25、Nb 2-62、Nb 2-65和Nb 2-67)。所有选定的纳米抗体都具有经过实验证实的结构,以便将它们与生成的候选纳米抗体的预测结构进行比较
Downstream Model: ConvBERT
最顶层的监督模型有两类:
- 一类是ProtTrans中所做的实验将cnn作为顶部/下游模型/层,这些模型被证明在与自我注意相结合时表现更好
- 另一类是linear layers,加上激活函数和softmax
- 当然还有第三种,但不是所有情况都共享的,只有回归或二分类任务,在有监督的网络前使用一个max pooling
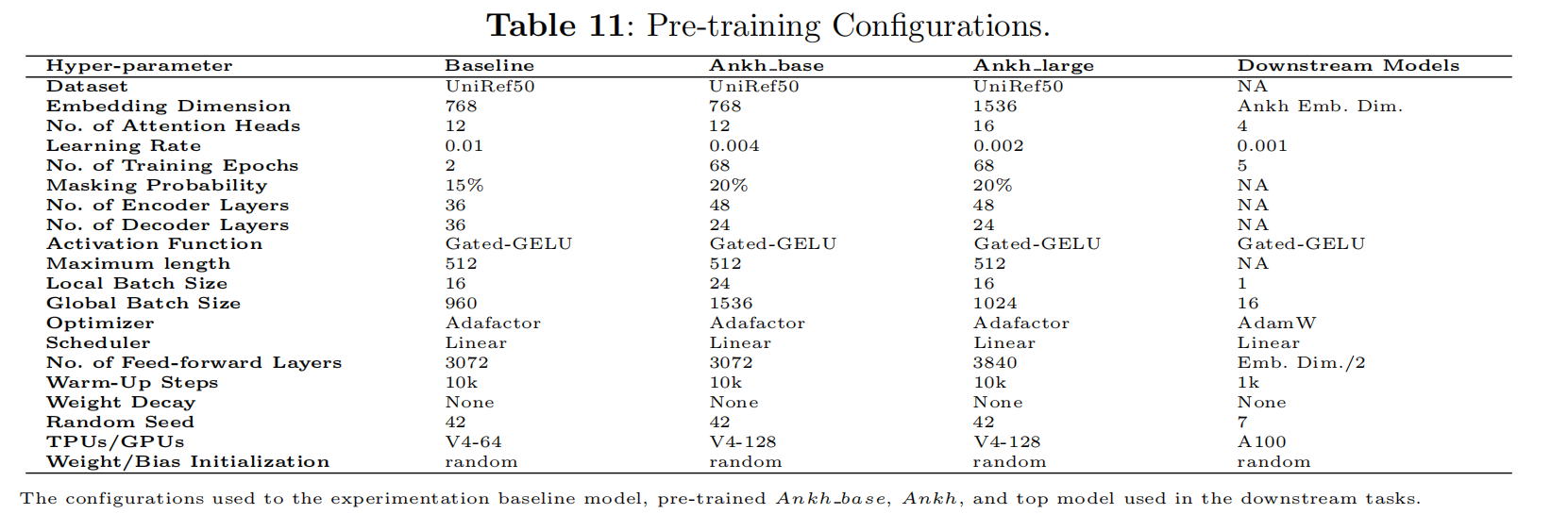
一般来说,对特定于任务的优化,不同的模型具有不同的超参数和配置集,可以导致更好的下游性能。本文统一了这个最好模型的设置,可以实现更好的广义性能
Variant Generation Model
对基于家族的auto-regressive微调和单蛋白生成的MLM
自回归微调的时候冻住了encoder只改变decoder,训练了2个epoch,最大序列长度256,最大prompt长度20,lr3e-4,epsilon value e-8,train batchsize 4,evaluation batchsize 8,beam search 10,temperature value 1,1.5,2。先改变temperature再用beam search采样
MLM用了两组参数,50%MLM和t=1,40%MLM和t=1.5,beam search 30
Computational Power (Software & Hardware)
Flax & Jax,TPU
Data & Model Experimentation
Masking
- Masking Strategy
-
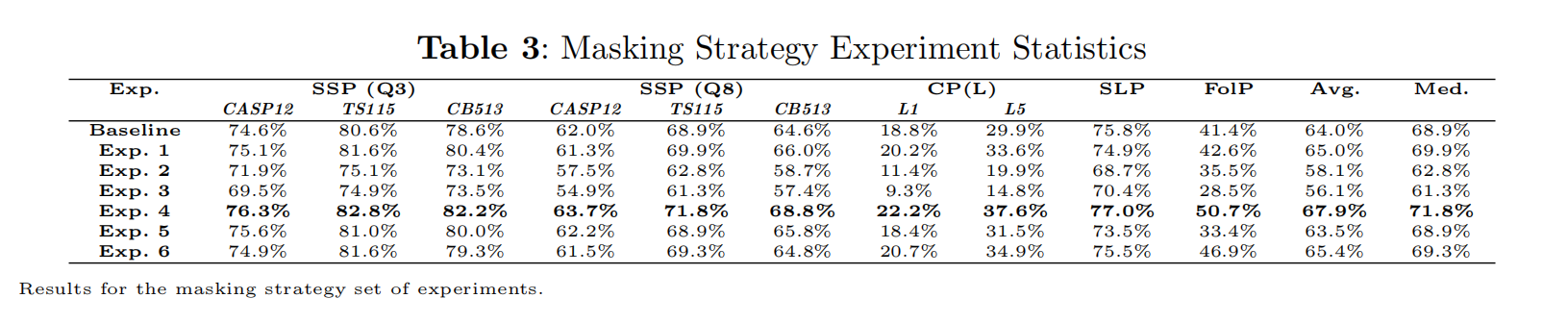
- Exp1
- 1-gram mask,每个出现的氨基酸至少被mask一次,比如 “ABCAAAAAAA…A”长度是20,按15%掩码,那么mask一个A,一个B和一个C
- 有性能提升
- Exp2
- 3-gram mask,比如“ABCDEFG”,如果D选中了,那么CDE都被mask
- 所有下游任务都性能下降
- Exp3
- 1-gram mask,在msak的地方算loss,比如 “ABCDEFG”,选择mask了DF,那么只在DF上算Loss,而不是重建整个input
- 所有下游任务性能下降
- Exp4
- 1-Gram Span Partial Demasking/Reconstruction:改变了输入和目标之间的映射方式,如果输入序列是“ABCDEFG”,标记“C”和“G”被掩码,那么输入序列会被表示为“A, B, [MASK0], D, E, F, [MASK1]”,然后被重构为“[MASK0], C, [MASK1], G”
- 相较于exp有性能提升
- Exp5
- 把exp4用在exp1上面,比如 “ABCAAAAAAA…A”长度是20,输入是“[MASK0], [MASK1], [MASK2], A, A, A, A, …, A”,重构为 “A, B, C, [MASK0]”,后面这个[MSAK0]是单个token映射到17个输入token
- 性能下降
- Exp6
- exp4的变体,输入是 “ABCDEFG” ,选择CDE做mask,输入 “A, B, [MASK0], F, G”重建为 “[MASK0], [MASK1], [MASK2]”
- 没用
- exp4最有用,然后沿用到剩下的实验
-
- Masking Probability
-
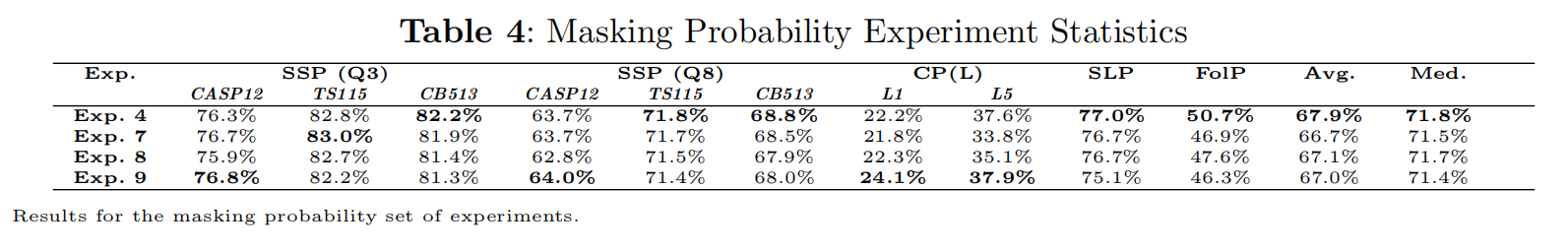
- exp7:10%
- exp8:20%
- exp9:30%
-
Architecture
-
-
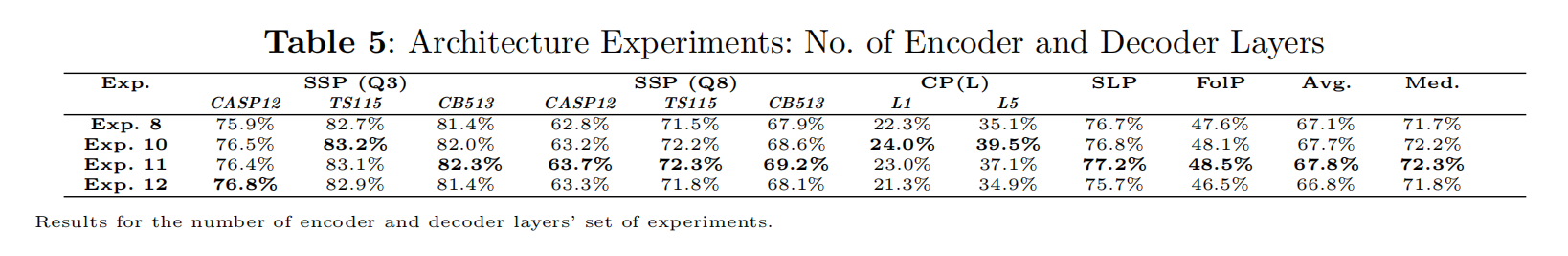
Number of Encoder-Decoder layers
- 对于Prot-T5而言,使用decoder并没有对下游任务产生显著提升,但是删掉可以减少一半的推理成本,所以做了编码器大于解码器的实验(不对称编码-解码)
- exp10:54encoder-18decoder
- exp11:48encoder-24decoder
- exp12:24encoder-48decoder
-
Depth vs. Width Variation layers
-
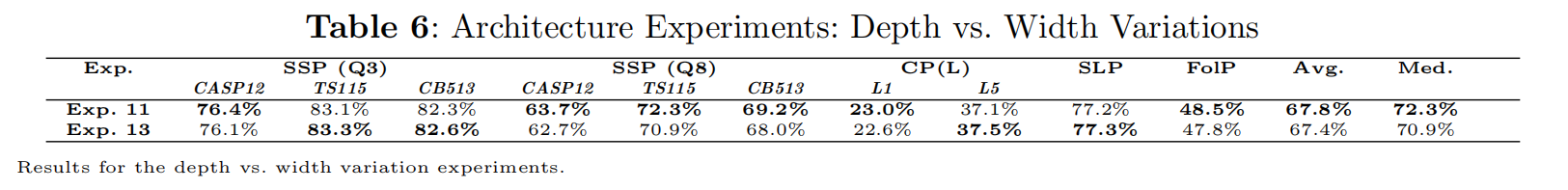
- exp13:embedding 768到1024,48encdoer-24decoder到24encoder-12decoder
-
-
Activation Function
-
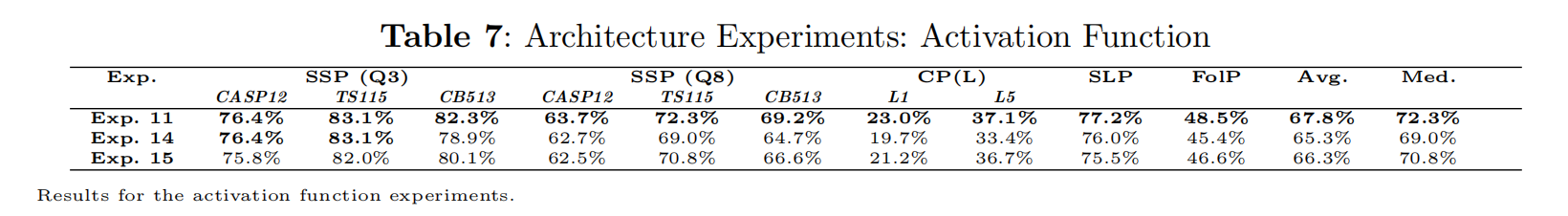
- 使用经典的Relu测试了两个组合,先前用的Gated-GELU
- exp14
- 62-layer encoder and 11-layer decoder with an embedding dimension of 768
- exp15
- 48- layer encoder and 24-layer decoder, also with an embedding dimension of 768
-
-
Relative Positional Embedding
-
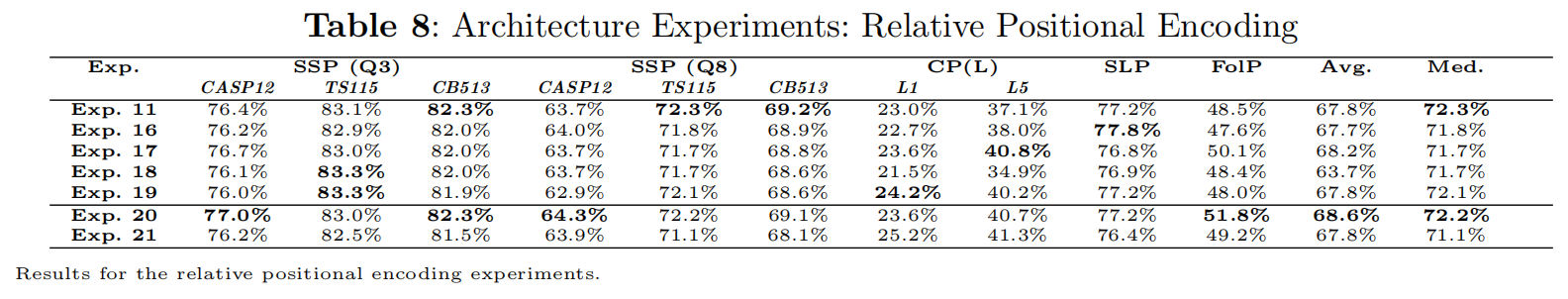
- 使用了一个简化的相对位置编码
- exp16:嵌入维度32,offset 256
- exp17:嵌入维度32,offset 64,发现64比默认的128和更大的256都好
- exp18:嵌入维度64,offset 64
- exp19:嵌入维度16,offset 64
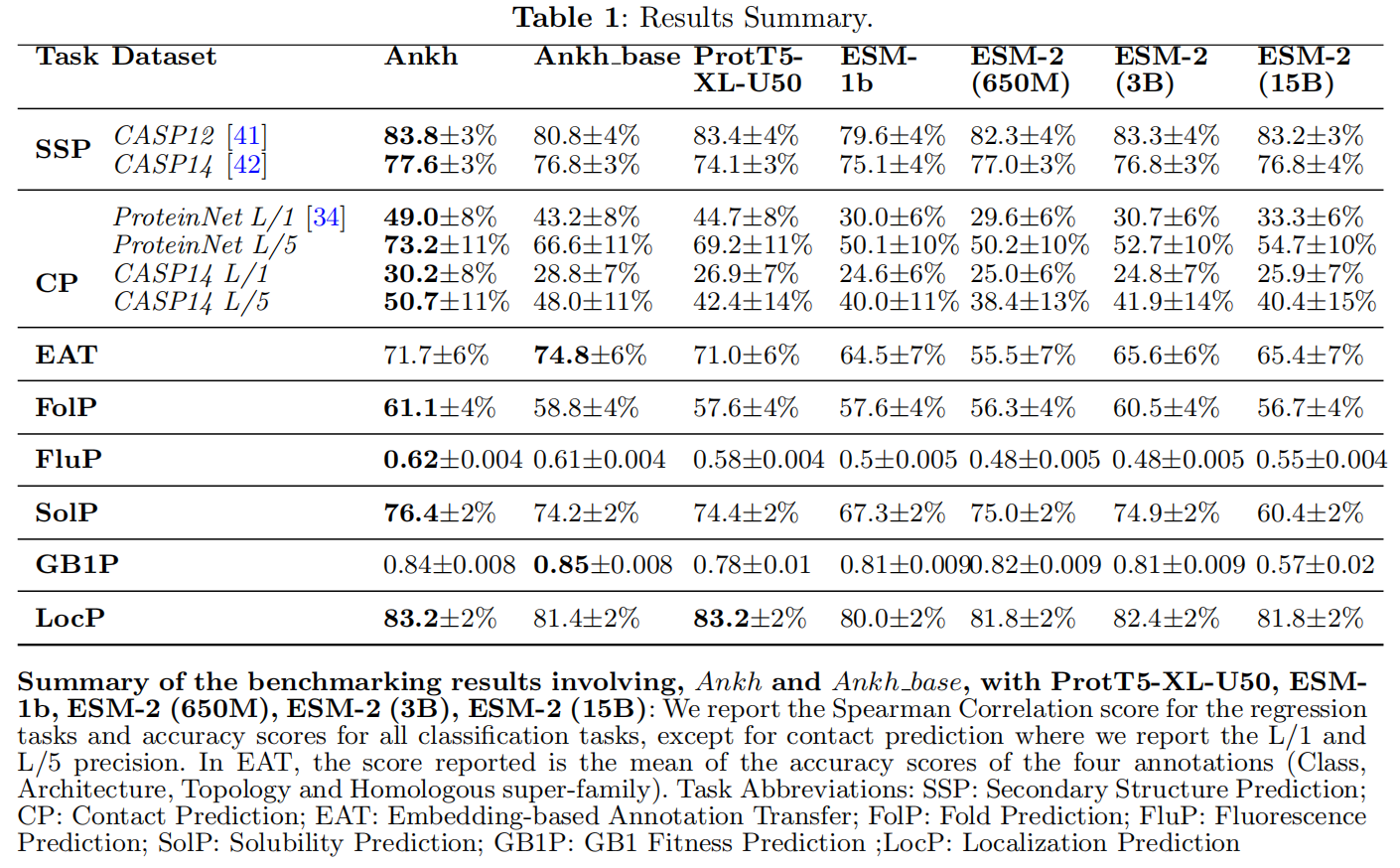
- exp20:嵌入维度64,offset 128
- exp21:嵌入维度128,offset 256
-
-
Weight Typing
-
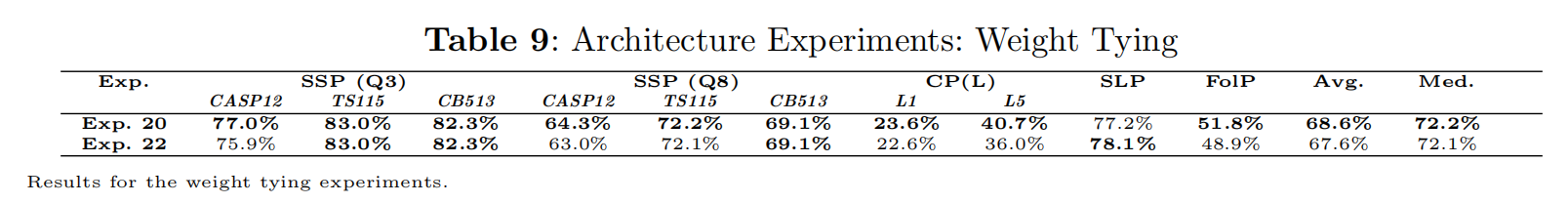
- 是否绑定embedding的权重
-
Dataset