论文地址:Structure-informed Language Models Are Protein Designers
LM-DESIGN:结构模型输入序列模型精炼蛋白质设计序列
Abstract
基于序列的蛋白质语言模型(plm)的通用方法,将轻量级的结构adapter丢到plm中,在推理过程中,进行迭代再细化,以有效地优化生成的蛋白质序列
Introduction
基于结构的蛋白质设计方法主要受限于:
-
通过实验确定的蛋白质结构数据有限
-
结构上有不确定性区域
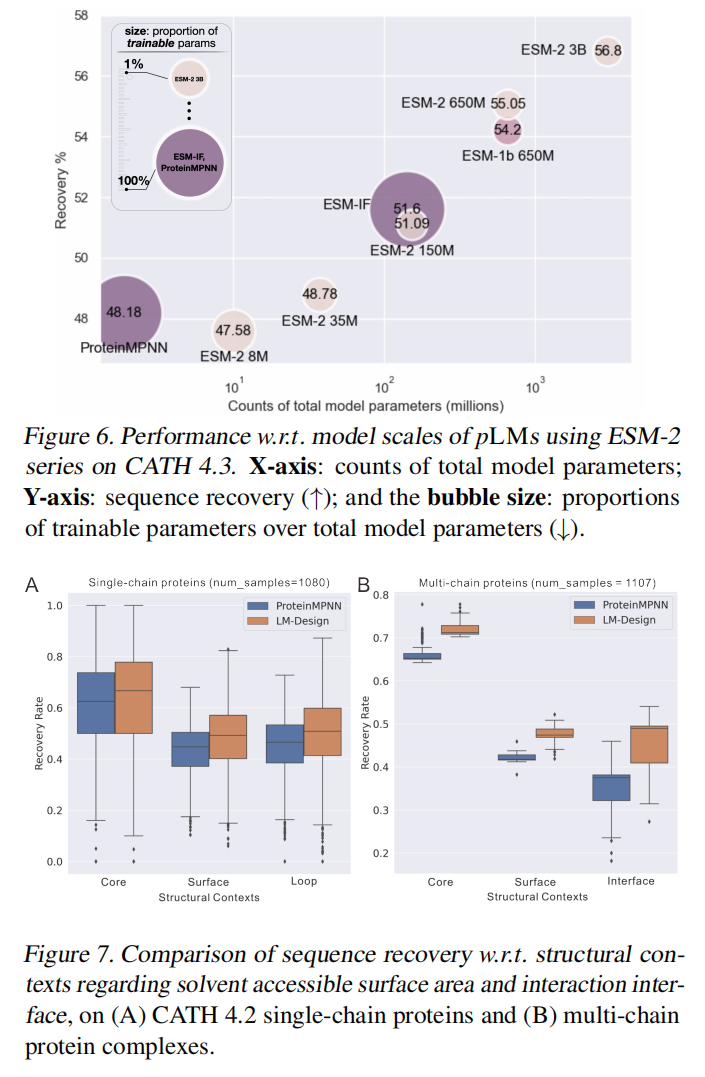
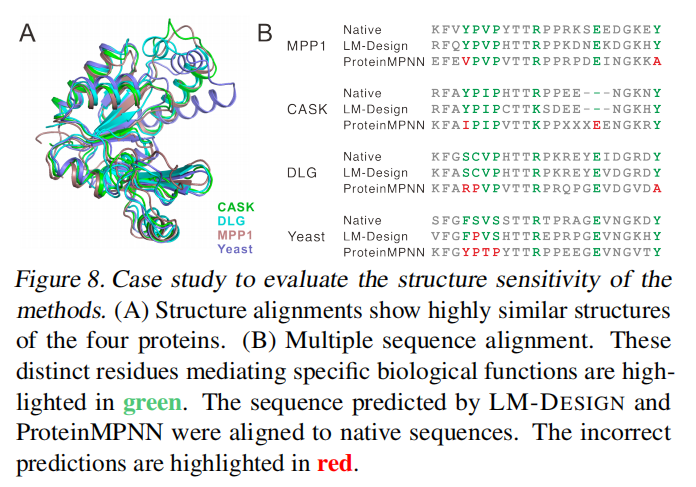
从生物学的角度来看,蛋白质结构有时没有足够的信息,特别是对于那些灵活的区域,如环和暴露的表面。在这些区域中,假设残差恒等式与结构上下文的相关性较小,而序列知识更有用,但在很大程度上被忽略了。我们验证了这一假设,并发现现有的纯基于结构的方法容易为这些区域产生功能无效的序列
所以用结构模型来提示序列模型生成序列
Reprogramming pLMs for Structure-based Protein Design with Structure Surgery
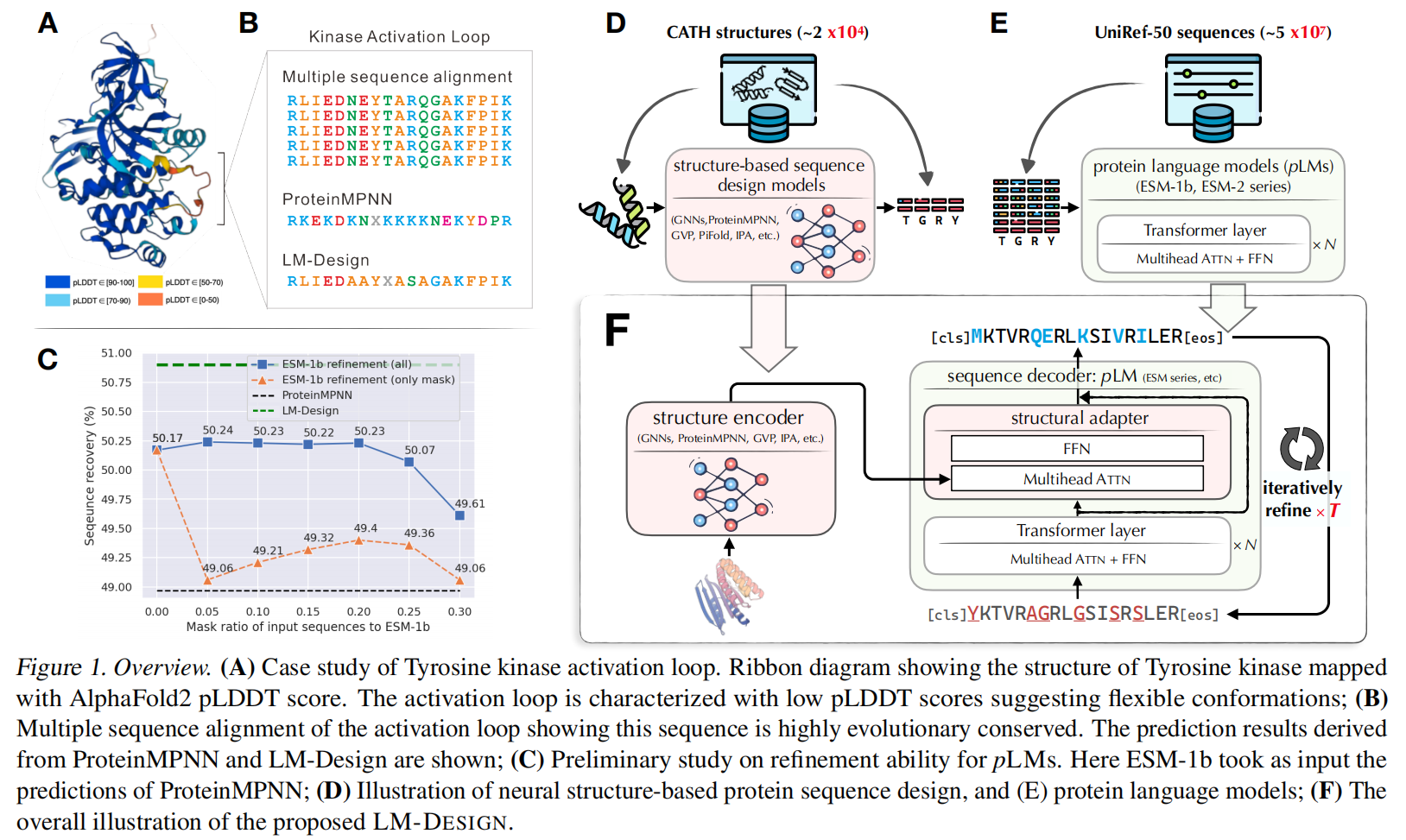
增加这种structual adapter是模型无关的方法
Training
去噪自编码方式,使用conditional masked language modeling(CMLM),用这个目标函数只看到左侧的氨基酸对于序列生成任务而言更好
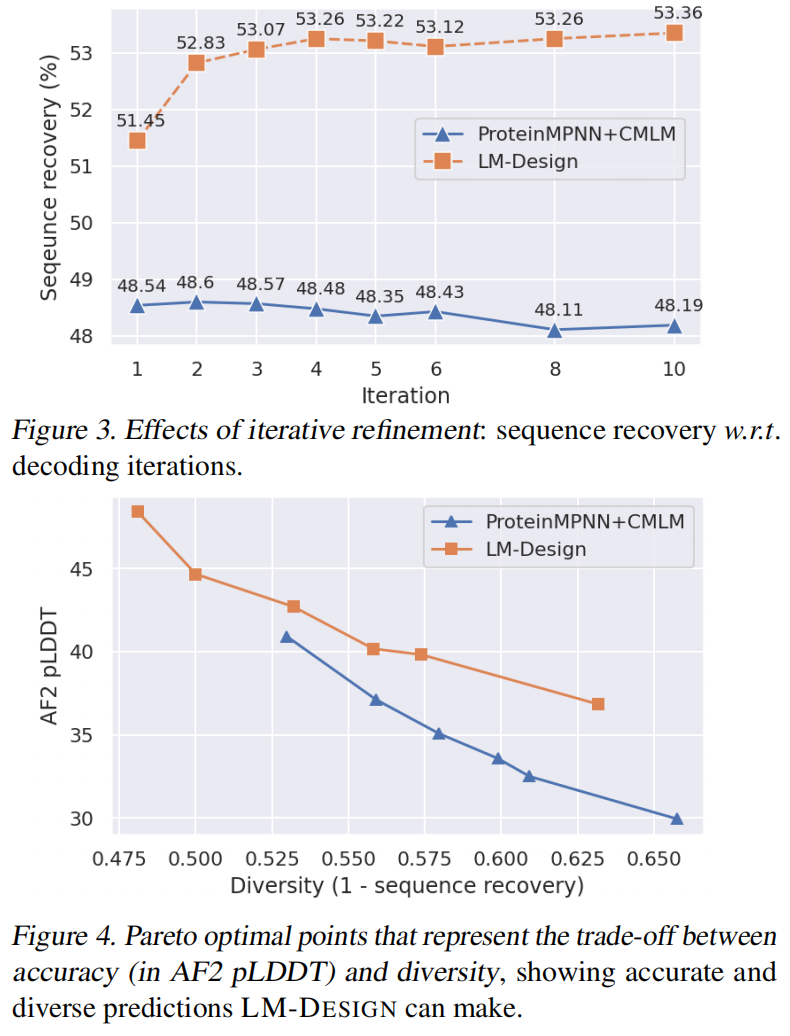
Inference with Iterative Refinement
从结构模型用一个线性头映射到20个氨基酸得到的序列作为初始序列,反复迭代T次直到不再优化
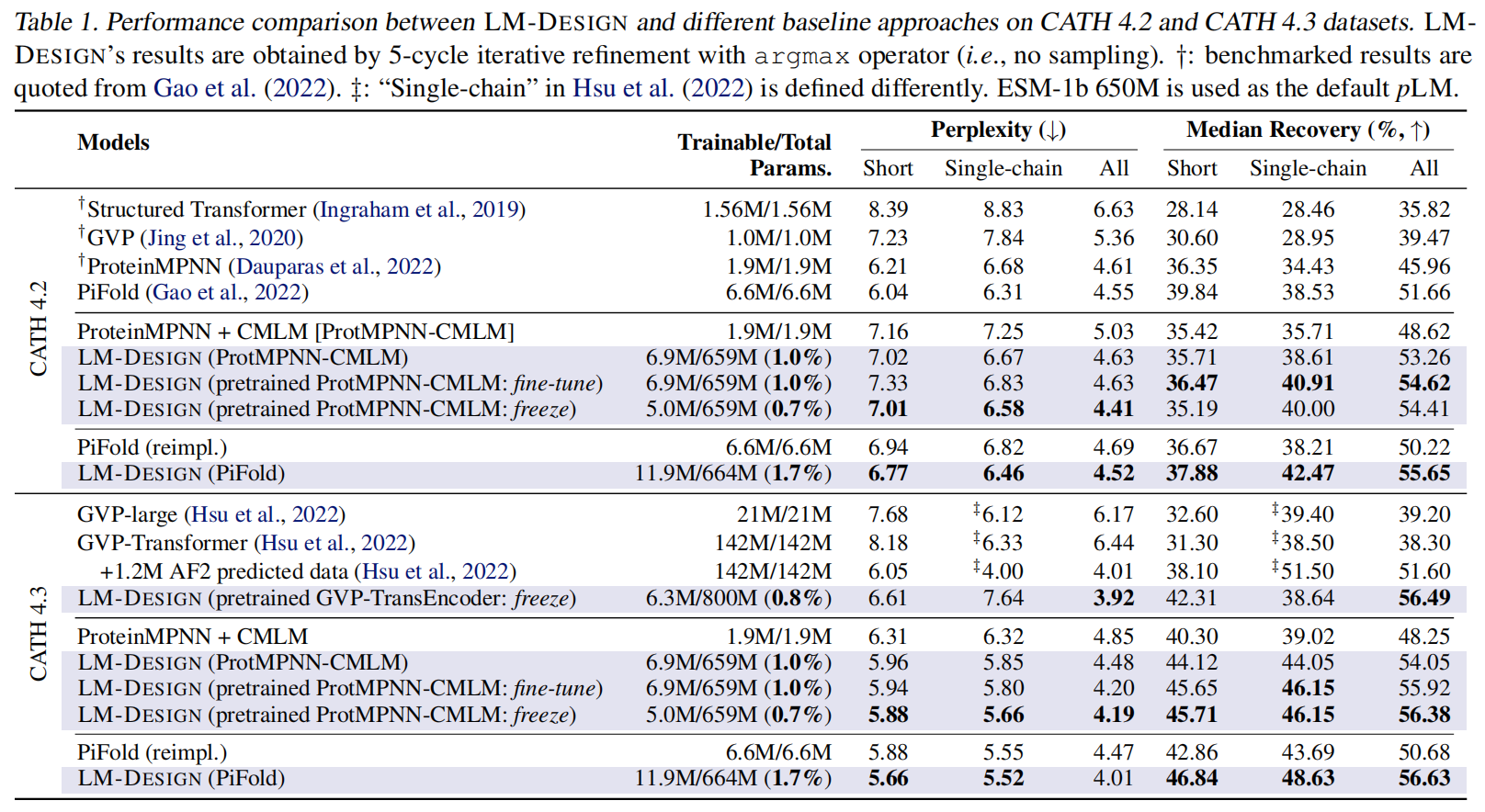
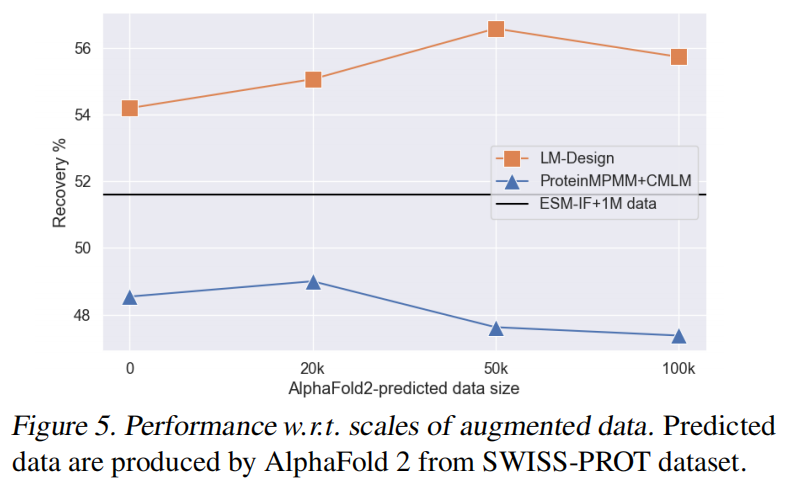
Experiments