论文地址:PoET:A generative model of protein families as sequences-of-sequences
PoET:两层casual attention建模同源序列做突变预测
Abstract
目前蛋白质语言模型大多是从MSA中训练得到的,这就导致了迁移性很差。提出PoET学习基于整个蛋白质家族的自回归模型,是一种检索增强的语言模型,可以从短的上下文长度进行推断,以及很好的推广到小家族去,通过在序列内顺序建模,序列间的顺序不变性,能够扩展到超出训练中使用的上下文长
Introduction
生成模型在大规模蛋白质语料上训练后可以通过采样生成现实的序列,也可以通过预测蛋白质变体的relative fitness来提高蛋白质序列变体效果
通常来说,基于家族的模型从蛋白质家族中学习到进化信息往往通过MSA或homo sequences,但是这对于蛋白质家族较少的蛋白无效且没办法迁移到别的蛋白上。使用MSA时是假设MSA是准确的,并且无法建模插入、删除的情况
作者提出PoET,通过学习在数千万个自然蛋白质序列簇中生成相关的蛋白质序列,能够概括跨蛋白质家族的进化过程,并避免与MSAs条件作用相关的问题。这是基于transformer layer的序列内order-dependence和序列间order-independence的方法
主要特性:
- 一个基于检索增强的蛋白质语言模型,可以条件性建模感兴趣的蛋白质家族,并且使用到新的蛋白质上的时候不需要再训练
- 自回归生成模型,不需要MSA因此不会有插入等对齐问题
- 在较短的context环境下也能对一些小蛋白家族有很好的泛化性
- 可以对任意序列进行打分
PoET
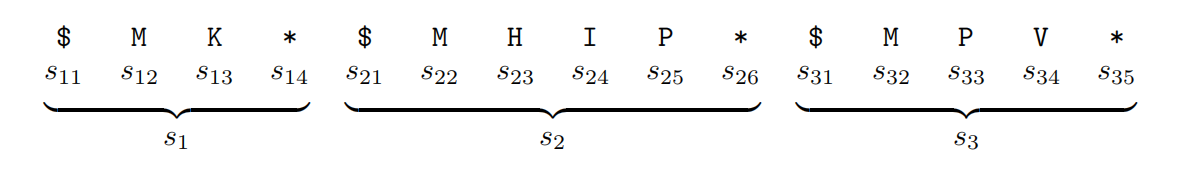
PoET是建模蛋白质家族分布的自回归模型,$P=(X=x)$ 其中 $x=s_1,s_2,…,s_n$ 是从同一个家族来的n条序列,下面这个例子就是长为4,6,5的三条序列
PoET生成每个token的概率分解如下
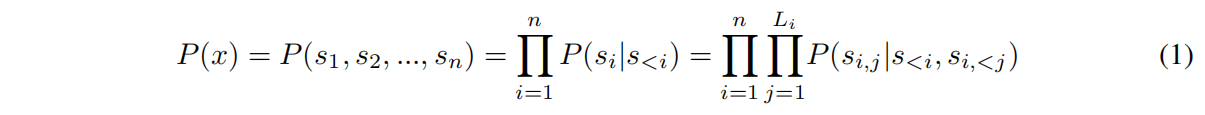
Model Architecture
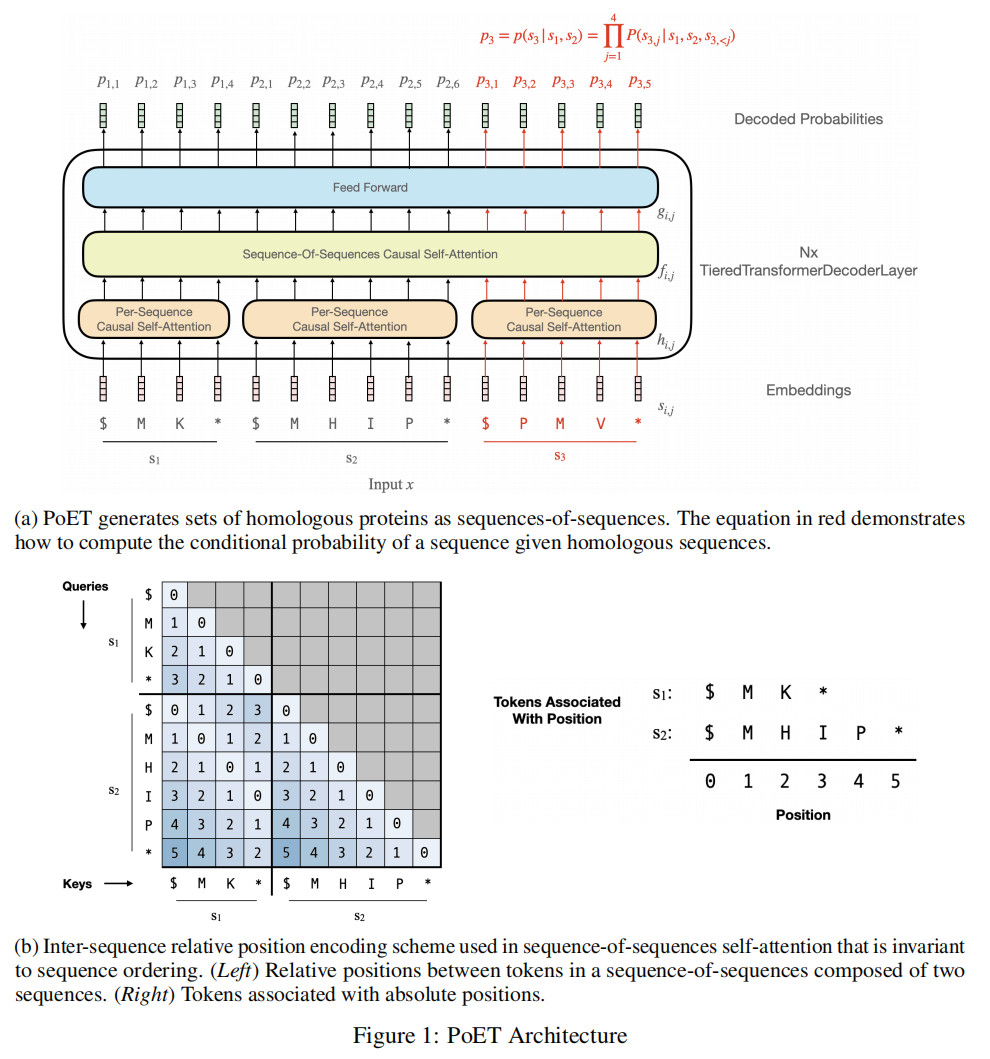
两个attention module:within-sequence module和between-sequence module
Input Embedding
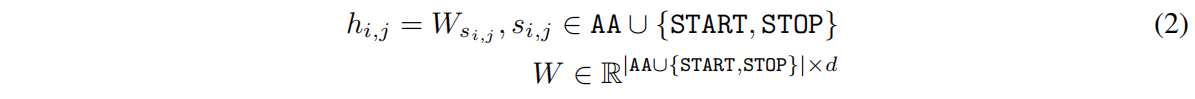
AA是20个标准氨基酸,d是embedding dimension
Tiered Transformer Decoder Layers
用N层TieredTransformerDecoderLayer叠加起来
使用了一种新的位置编码方式,比如 $f_{1,1}$ 和 $f_{2,1}$ 之间的相对位置是0,如图1b,有两个好处
- 同源序列蛋白中相似的的位置应该有更相似的分布
- 将最大相对位置编码数限制在了一个序列长度以内,而不是会有一串序列,这样能够更好的泛化到更长的sequences-of-sequences上
Decoded Probabilities
最后的output经过线性头投影分类得到logits
Training Data
在29M的同源序列集上训练,删除了少于10条同源序列的集合,为了避免对大的同源序列集过拟合,采样的时候与这个同源序列集成反比
Protein Variant Fitness Prediction
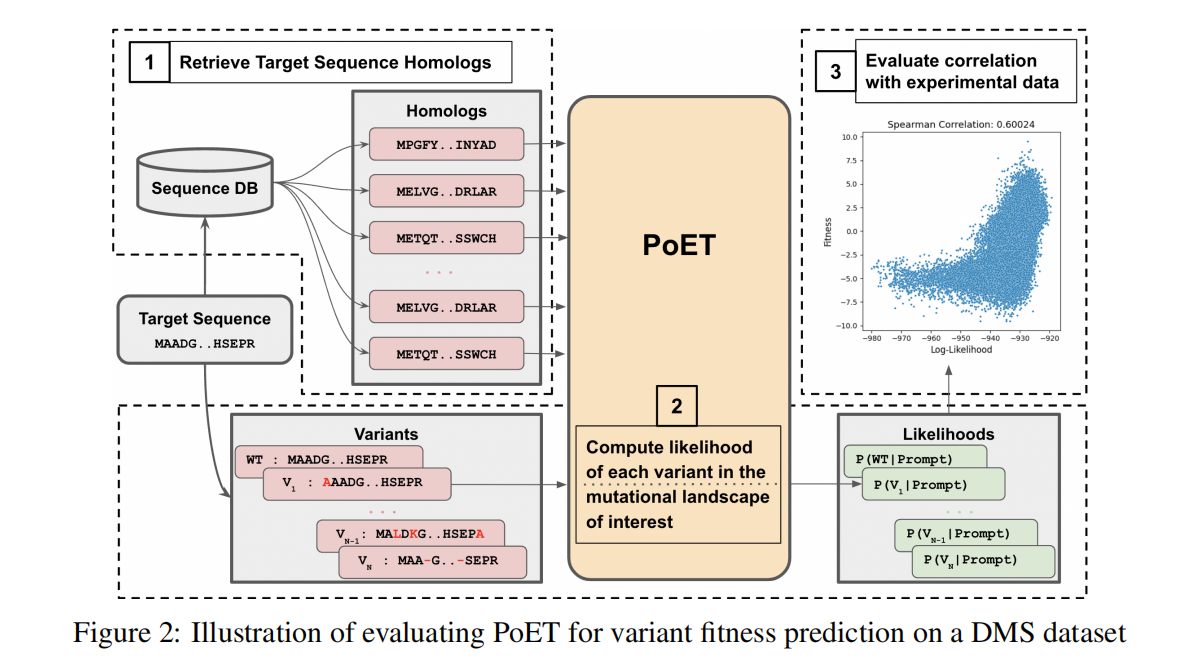
Benchmarking using deep mutational scans
在proteingym上评估,有87个substitution和7个indel
Fitness Prediction with PoET
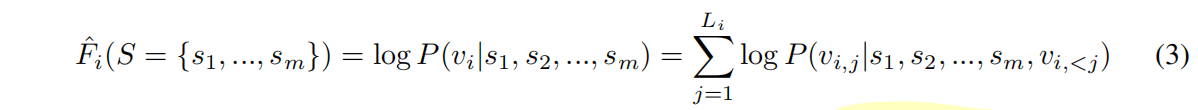
集合S是从Uniref100里面检索出来的
基于验证集的表现,根据以下三点来挑选最好的方法:
- 检索同源序列
- 采样和过滤同源序列来获得一个合理的上下文长度
- 从不同的下采样同源序列中集成conditional log-likelihood
集成如下:
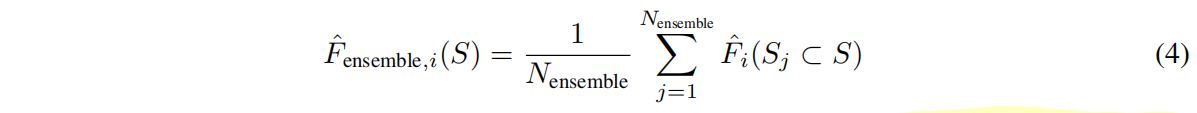
子集 $S_j$ 是不同序列相似度 {1*.0,* 0*.95,* 0*.90,* 0*.70,* 0*.50},context lenth {6144,* 12288*,* 24576},N_ensemble=15
Experiments
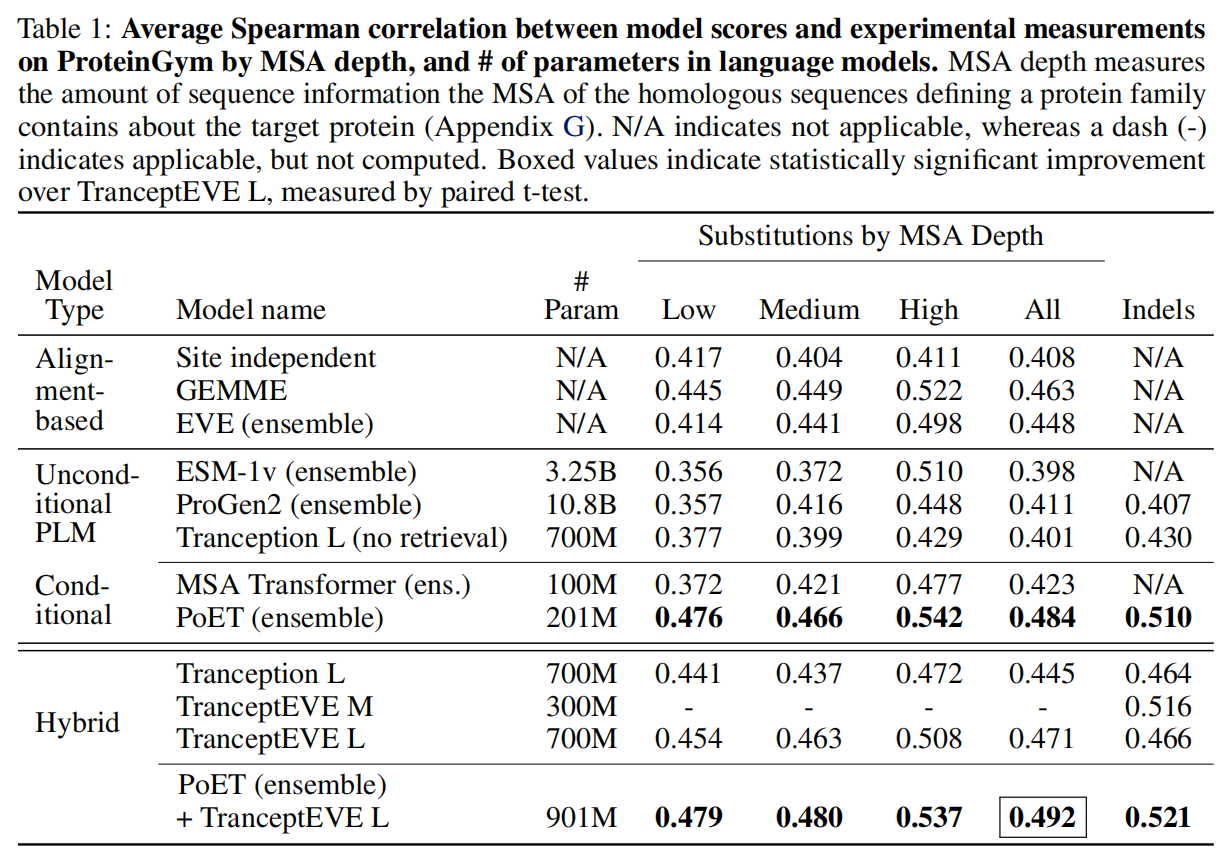
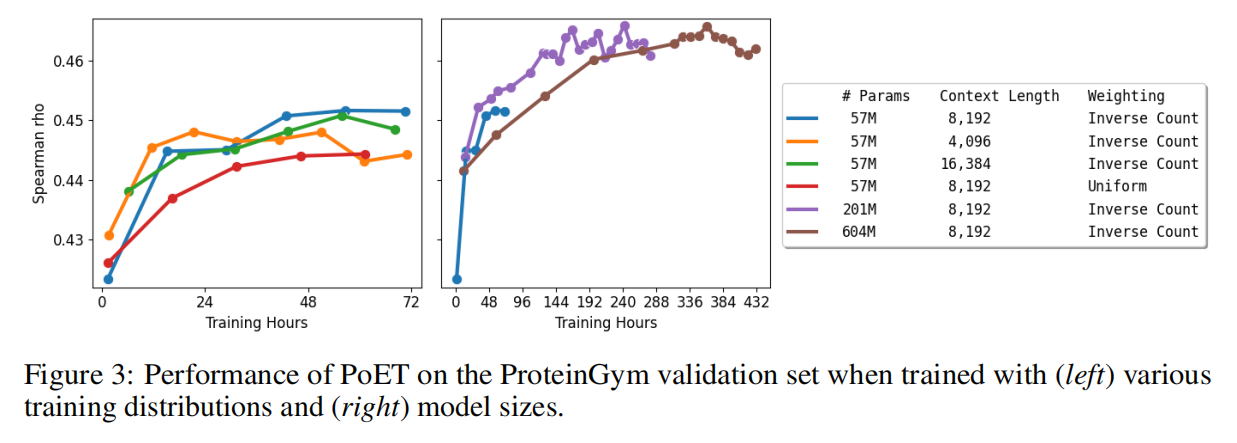
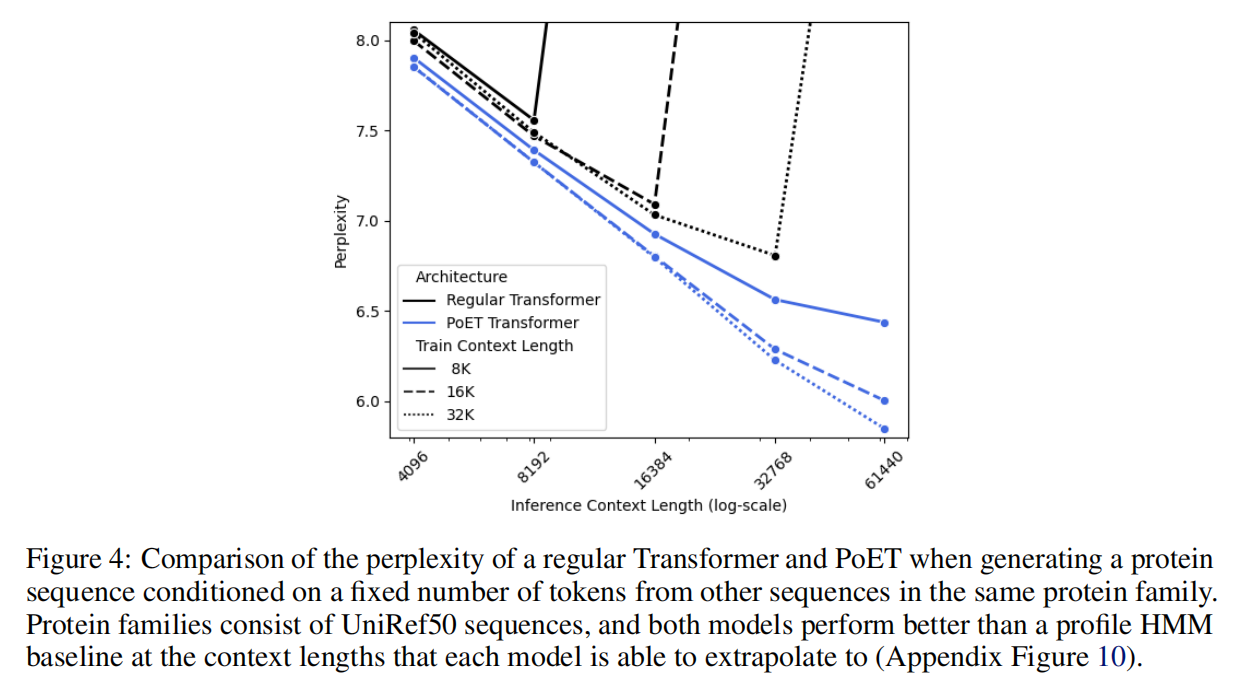
传统的transformer架构不能泛化到超过context长度


