论文地址:https://academic.oup.com/nar/article/50/W1/W228/6576357
论文网站:https://services.healthtech.dtu.dk/services/DeepLoc-2.0/
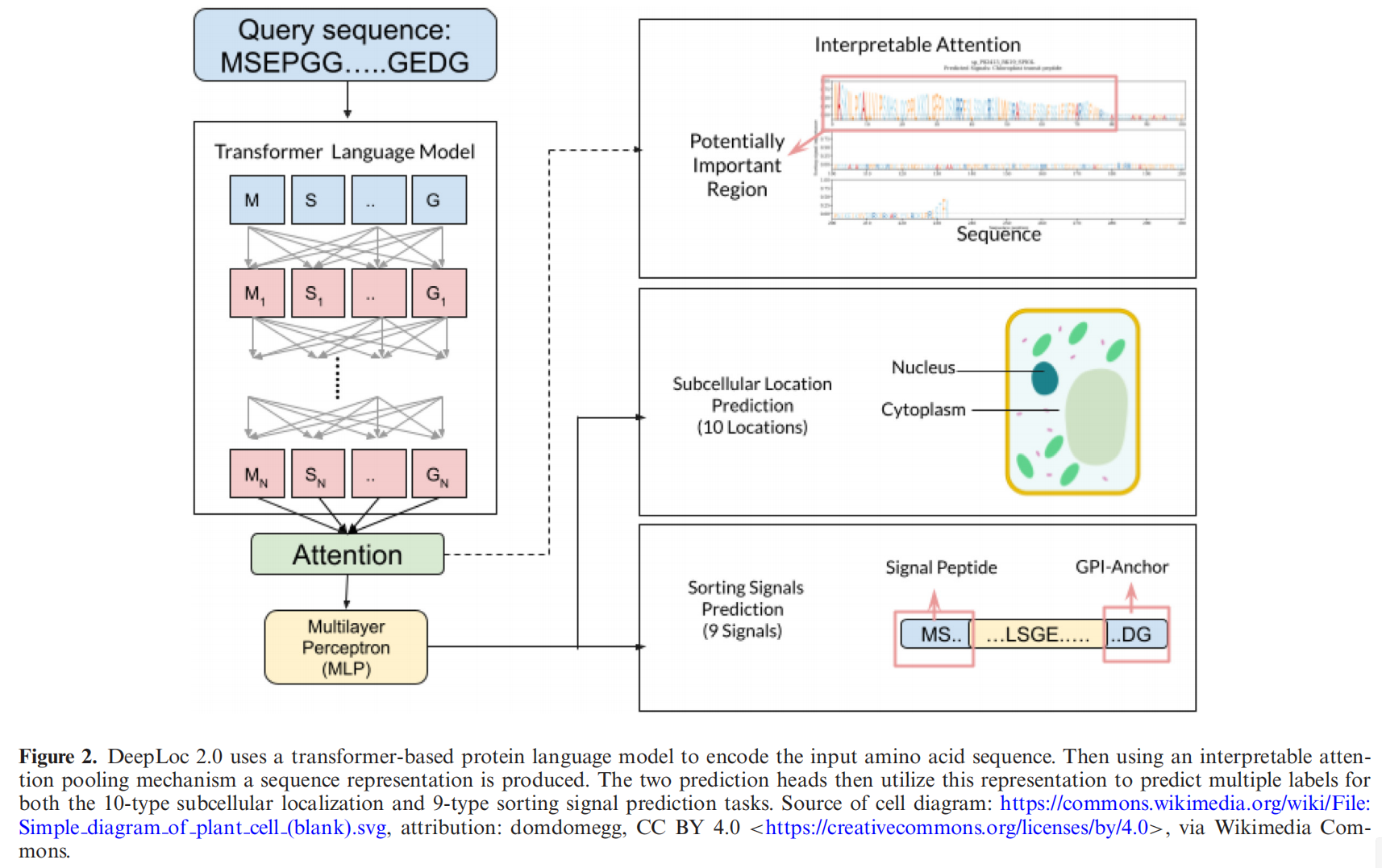
DeepLoc-2.0:预测亚细胞定位和蛋白质排序信号
Abstract
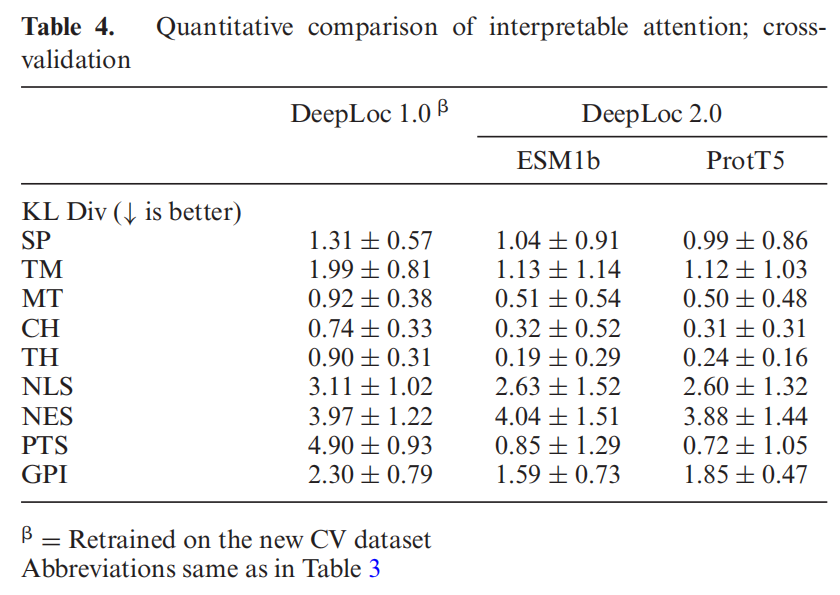
蛋白质亚细胞定位的预测对蛋白质组学的研究具有重要意义。提出了DeepLoc-2,具有多定位预测。为了进行训练和验证,我们通过严格的同源性划分来管理真核生物和人类的多位置蛋白数据集,并从文献中汇编的排序信号信息。提供了两种更好的可解释性的方法:沿着序列的注意输出和对9种不同类型的蛋白质分类信号的高度准确的预测,发现注意输出与排序信号的位置有很好的相关性
Introduction
识别不同细胞室中的蛋白定位在功能注释中起着关键作用。它还有助于识别药物靶点,并理解与异常亚细胞定位相关的疾病。已知一些蛋白质定位于多个细胞隔间。一些生物学机制已经被确定来解释定位过程,其中涉及到被称为排序信号的短序列
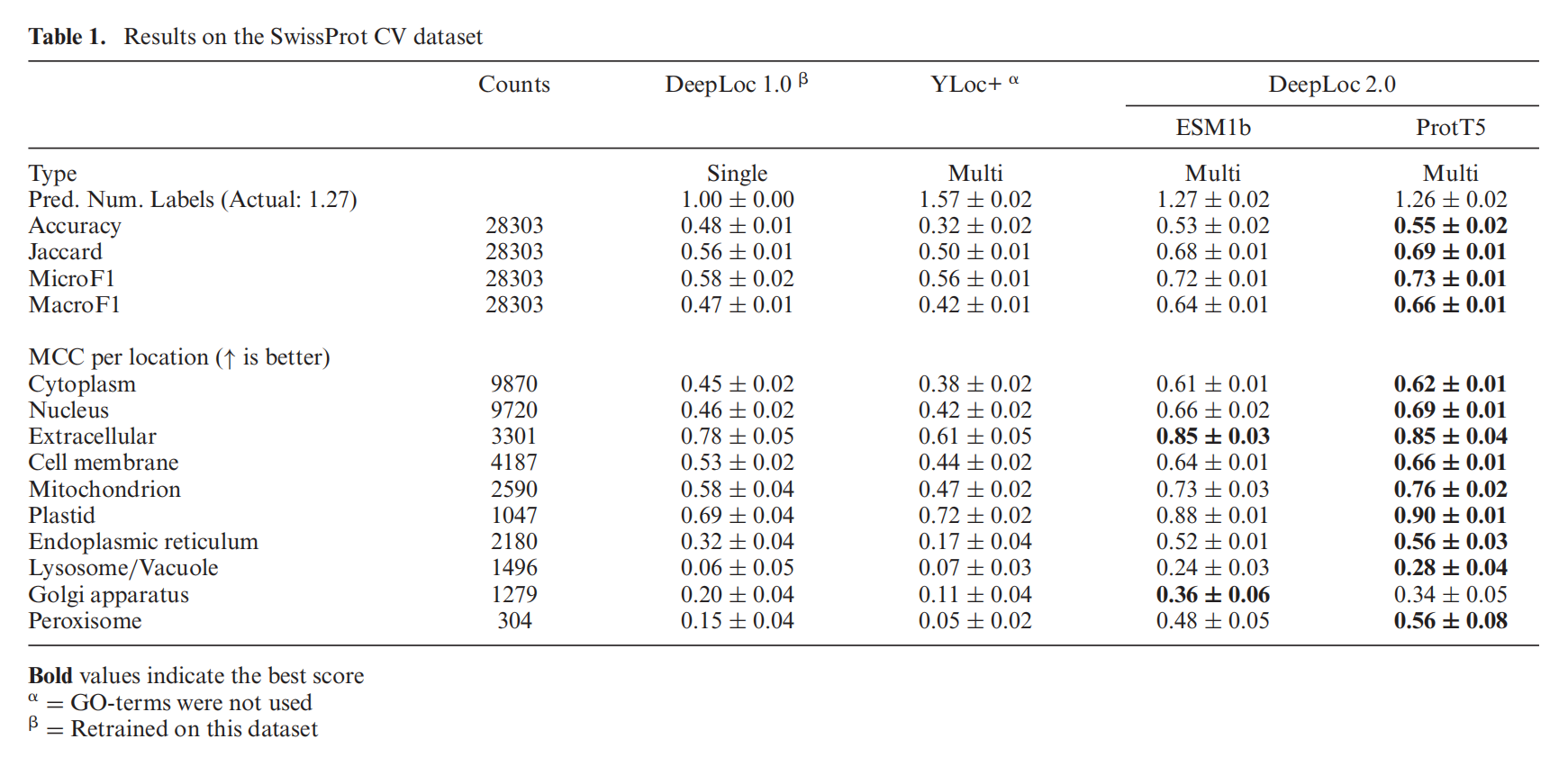
SwissProt localization dataset
蛋白质数据提取自UniProt数据库版本2021_03。然后使用以下标准对蛋白质序列和定位注释进行过滤:真核生物,而不是片段(这些片段可能有n端或c端排序信号缺失),编码在细胞核中,>40个氨基酸和实验注释(ECO:0000269)亚细胞定位。这些蛋白质可以分为这十个位置中的一个或多个:细胞质、细胞核、胞外、细胞膜、线粒体、质体、内质网、溶酶体/液泡、高尔基体、过氧化物酶体
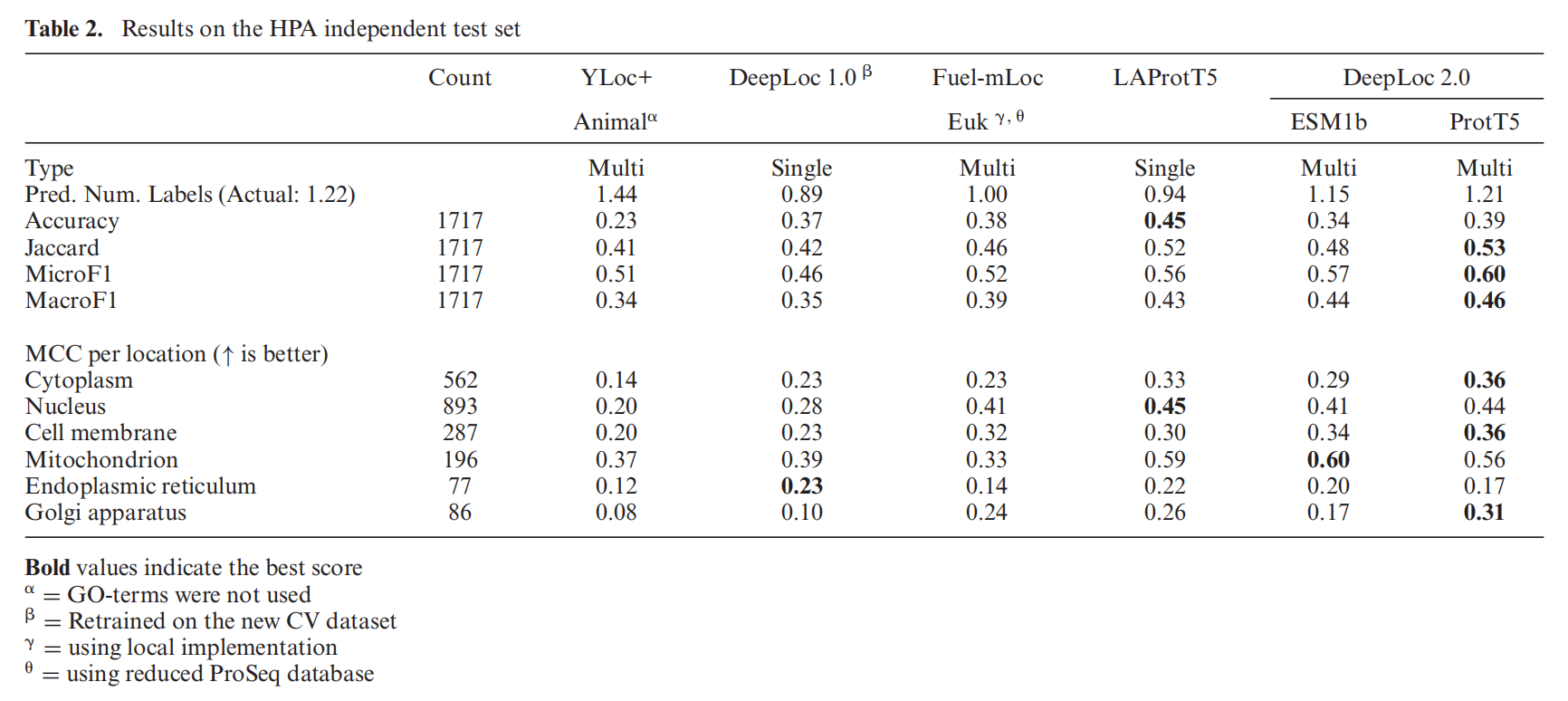
Human protein atlas
人类蛋白图谱(HPA)项目利用共聚焦显微镜提供了人类蛋白的亚细胞定位。根据文献中的抗体验证和实验证据等各种标准,这些注释提供了四个可靠性标签:增强、支持、批准和不确定(Enhanced, Supported, Approved, and Uncertain)。我们只考虑独立测试集的增强的和支持的注释,因为这些注释是最可靠的标签。该数据集被确保与上述数据集没有任何具有>30%全局序列标识的序列,并用于独立验证。
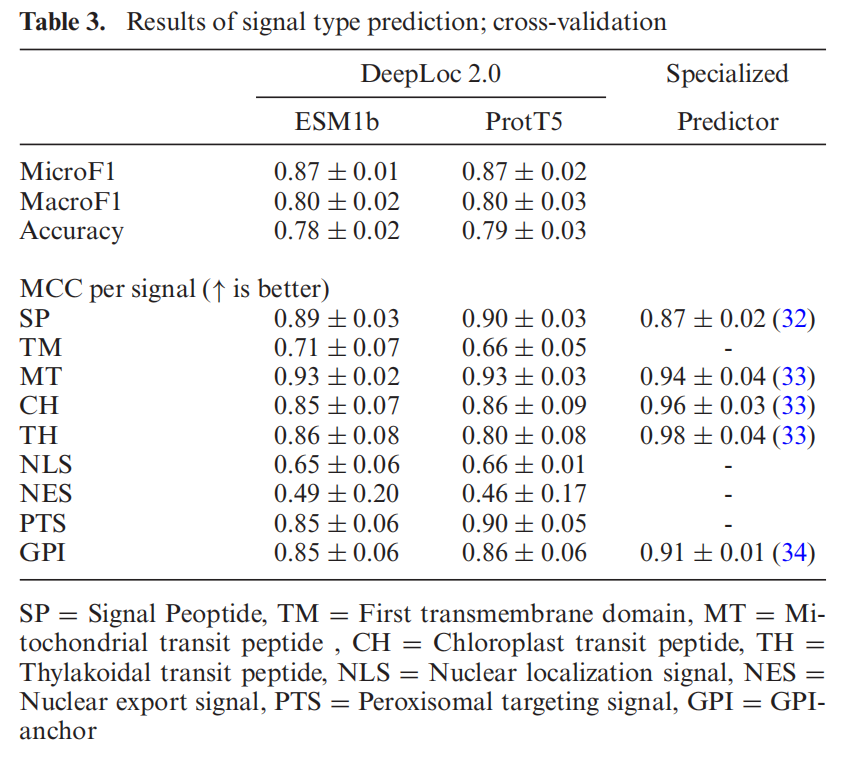
Sorting signals
经过实验验证的注释排序信号主要来自文献,排除了在我们构建的SwissProt定位数据集中不存在的蛋白质
DeepLoc 2.0 Overview