论文地址:Protein generation with evolutionary diffusion:sequence is all you need
EvoDiff:序列diffusion做蛋白质设计
Abstract
目前的生成式diffusion model可以生成看似合理的蛋白质,但SOTA方法都限制了生成在一个小的蛋白质设计空间,本文提出了一个通用的diffusion framework EvoDiff,结合了进化数据可控制的生成蛋白质。EvoDiff生成高保真度、多样化和结构合理的蛋白质,覆盖自然序列和功能空间。重要的是,EvoDiff可以生成基于结构的模型无法获得的蛋白质,比如那些具有无序区域的蛋白质,同时保持设计功能结构基序支架的能力,这证明了基于序列的公式的普遍性
Introduction
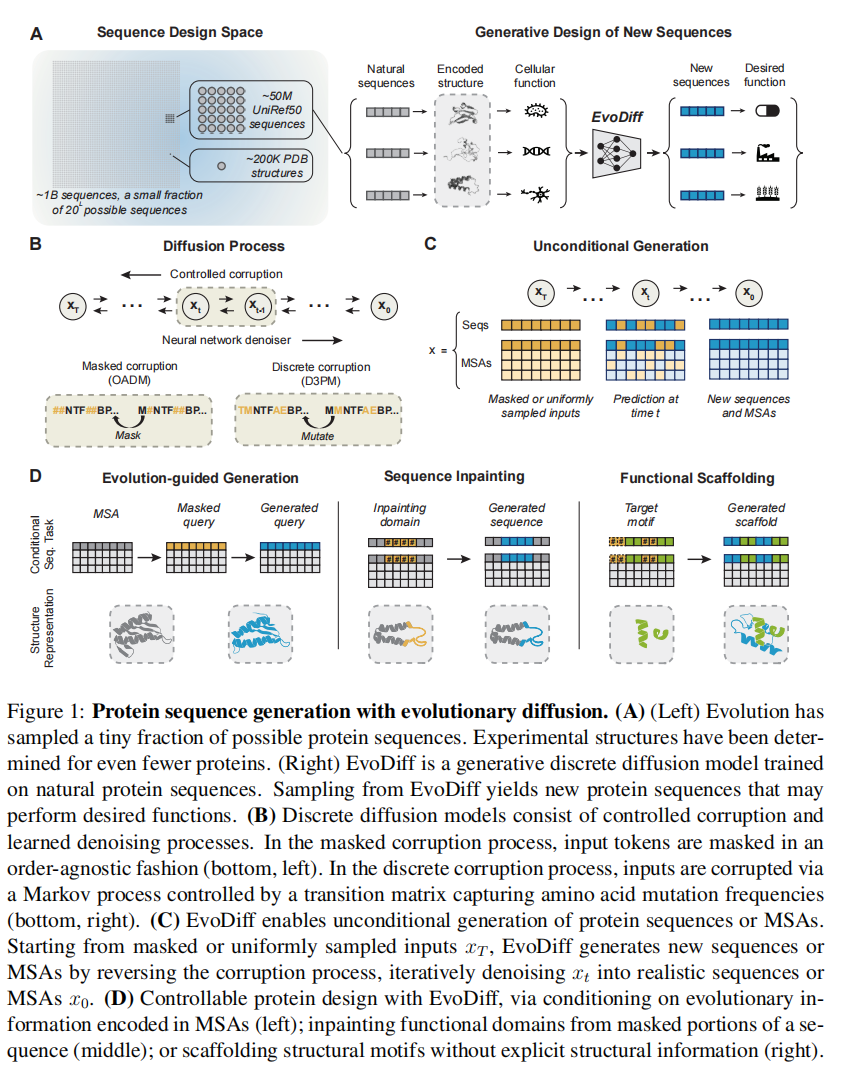
相比较于结构而言,序列才是更加全面的蛋白质设计搜索空间
序列通过结构构象的集合和氨基酸本身所产生的化学作用来决定功能。然而,并不是每个蛋白质都能折叠成一个静态的结构,很多情况下基于结构的设计是不可行的,因为功能不是由静态结构调节的。比如一些intrinsically disordered regions (IDRs),因此,具有x射线晶体学特征的静态结构是对在序列空间中捕获的信息的不完全蒸馏。另一个问题是结构数据太少,PDB中只有200K,不能表明出天然序列的完整性
本文提出的方法结合了进化相关数据,仅使用序列进行可控的蛋白质序列生成。使用discrete diffusion framework来生成序列,还建立了在msa上训练的离散扩散模型,以利用这一额外的进化信息层来生成新的单个序列
通过一系列的生成任务(图1D)评估了EvoDiff-Seq和EvoDiff-MSA模型,以证明它们在可控蛋白质设计方面的能力
Discrete diffusion models of protein sequence
研究了在离散数据模式上扩散的两种正向过程,以确定哪一种过程最有效,EvoDiff-OADM or EvoDiff-D3PM
EvoDiff sequence models在42M的UniRef50上训练,使用CARP蛋白质掩蔽语言模型中引入的扩展卷积神经网络架构。EvoDoffOADM比两个基于统一的转移矩阵的EvoDiff-D3PM变体更准确地学习重建测试集
EvoDiff MSA models用MSA Transformer在OpenFold数据集上训练,通过随机采样序列(“随机”)或贪婪地最大化序列多样性(“Max”),将msa下采样为512个残基,深度为64个序列。OADM损坏导致最低的验证集困惑,表明OADM模型能够最好地推广到新的msa
Structural plausibility of generated sequences
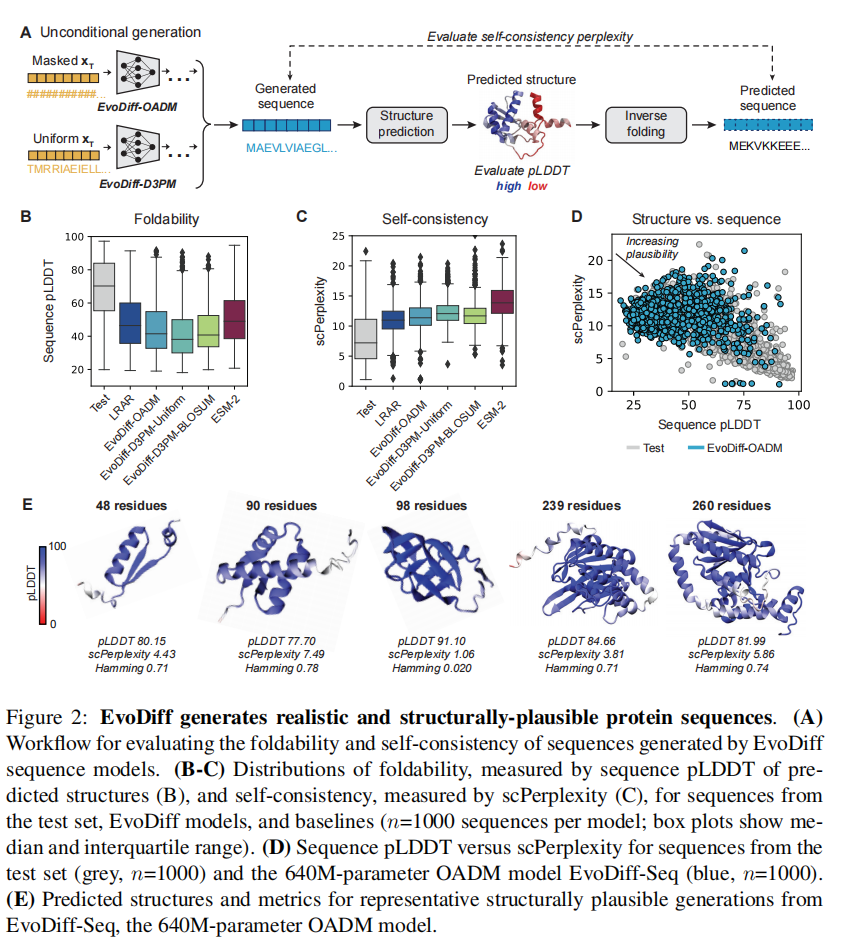
从每个EvoDiff序列模型中生成了1000个序列,其长度来自于训练集中长度的经验分布,生成结果与CARP自回归模型和ESM2的mask模型进行了比较
折结构的工具是OmegaFold,然后计算PLDDT,较低的分数一般在IDRs区域;还设计了一个self-consistency perplexity (scPerplexity),用ESM-IF从结构还原序列计算困惑度。因为ESM-IF也是在Uniref50上训练的,所以验证集可能有overlap,用ProteinMPNN
Biological properties of generated sequence distributions
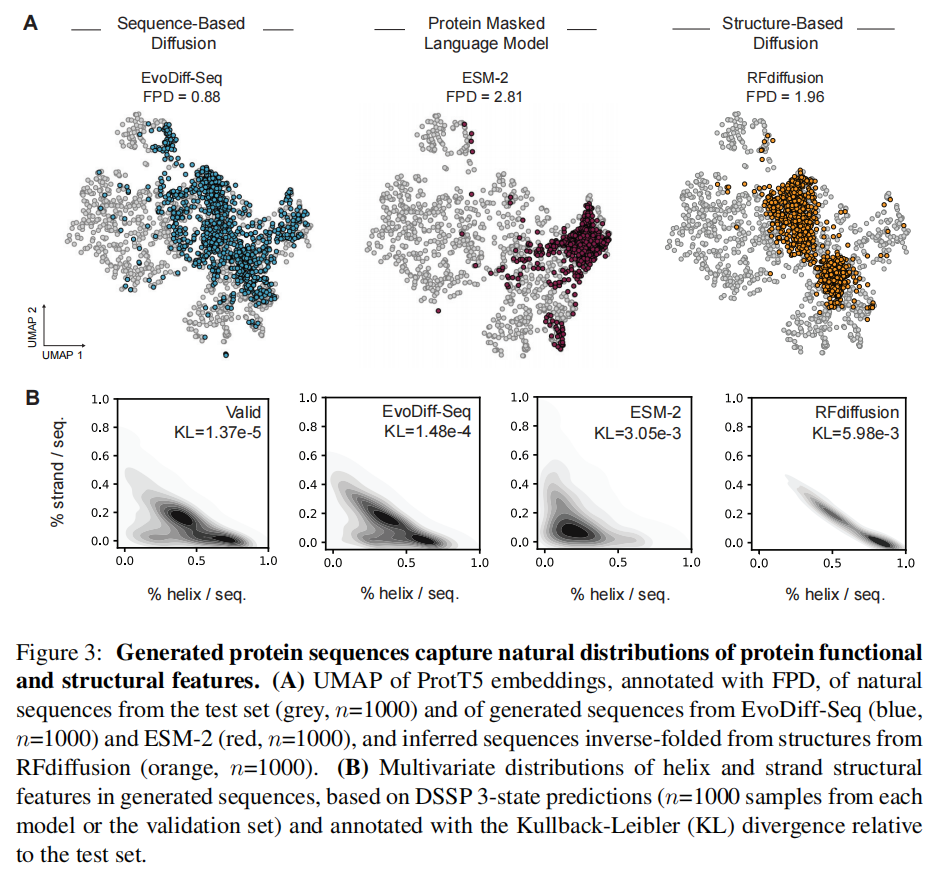
理想情况下,生成的序列应该捕获序列、结构和功能属性的自然分布,同时仍然具有彼此之间和与自然序列之间的差异
为了评估序列分布和功能属性分布的覆盖范围,使用ProtT5嵌入了每个生成的序列,将这个度量称为 Fr´echet ProtT5 distance (FPD),无论是定性还是定量,EvoDiff-Seq生成的蛋白质比最先进的蛋白质掩蔽语言模型(ESM-2)或最先进的结构扩散模型(RF扩散模型)的结构生成的预测序列更好地再现自然序列和功能多样性
为了评估生成序列中结构性质的分布,我们计算了三态二级结构,EvoDiff-Seq产生的链和无序区域的比例与自然序列更相似,而ESM-2和RFdiffusion都产生富含螺旋的蛋白质
Conditional sequence generation for controllable design
EvoDiff的OADM扩散框架通过固定一些子序列和替换剩余的子序列,提出了一种条件序列生成的自然方法。因为该模型被训练成生成具有任意解码顺序的蛋白质,所以这很容易通过简单地屏蔽和解码所需的部分来完成。我们将EvoDiff的能力应用于三种情况下的可控蛋白质设计:调节msa编码的进化信息,在绘制功能域,和支架功能结构基序
Evolution-guided protein generation with EvoDiff-MSA

从验证集中随机选择的250个msa中屏蔽查询序列,并使用EvoDiff-MSA新生成这些序列,然后用foldability和self-consistency评估质量
Generating intrinsically disordered regions
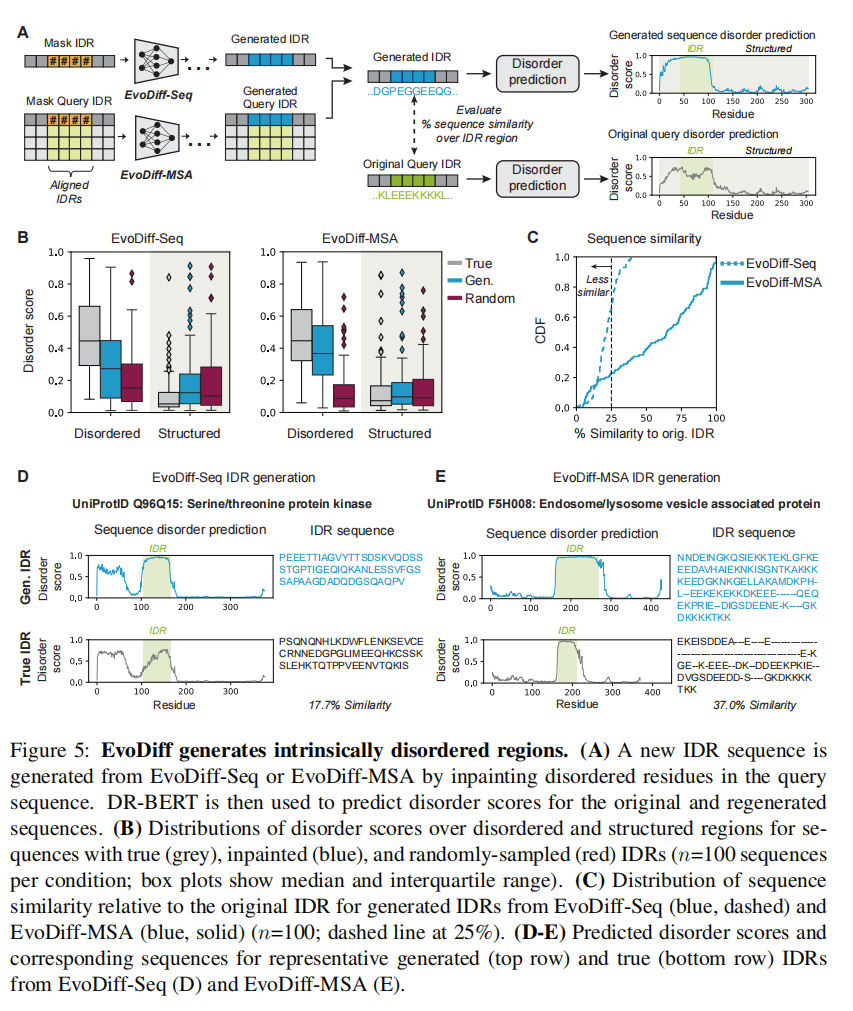
多达30%的真核蛋白含有至少一个IDR,而IDRs占真核生物蛋白质组中残基的40%以上,idr在细胞中发挥重要和多样的功能作用,通过缺乏结构直接促进,如蛋白质-蛋白质相互作用和信号转导
利用inpainting生成disorder区域,同时使用一个计算预测IDRs的数据集,这个数据集也构建了MSA
在使用EvoDiff通过插入绘制生成假定的idr之后,我们然后使用DR-BERT预测生成的和自然序列中的每个残基的无序分数
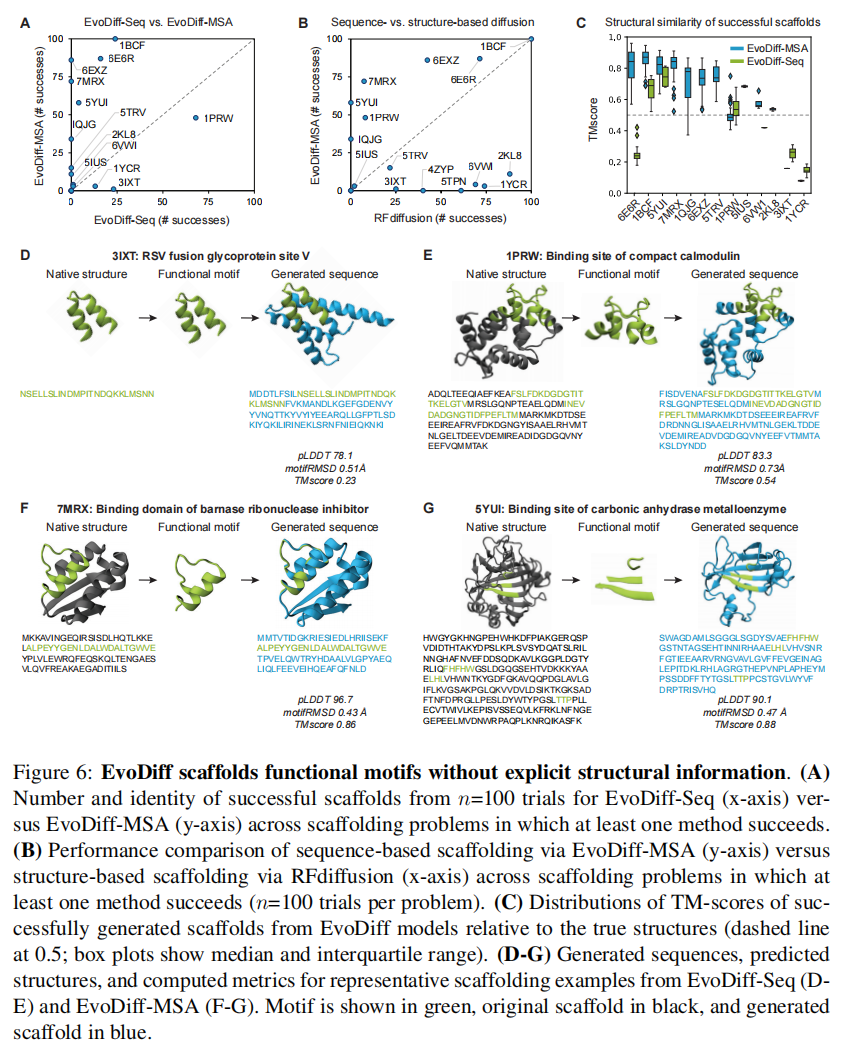
Scaffolding functional motifs with sequence information alone
鉴于固定的功能基序包含了基序的残基身份,研究了一个结构模型是否真的对基序支架是必要的
我们使用EvoDiff条件生成技术,通过固定功能motif,只提供motif的氨基酸序列作为条件信息,生成17个motif支架问题的支架,然后对其余序列进行解码
EvoDiff和RFdiffusion的特定问题成功率之间几乎没有相关性,而且两种方法的高成功率也很少,这表明EvoDiff可能与RFdiffusion具有正交强度
Discussion
EvoDiff是第一个证明扩散生成建模在进化尺度的蛋白质序列空间上的能力的深度学习框架。与之前在蛋白质结构和/或序列上训练扩散模型的尝试不同,EvoDiff是在所有自然序列的大样本上训练的,而不是在较小的蛋白质结构数据集或来自特定蛋白质家族的序列数据上训练的。以往在全局序列空间上训练的蛋白质生成模型要么是从左到右的自回归(LRAR)模型,要么是掩蔽语言模型(MLMs)
EvoDiff的OADM训练任务概括了LRAR和MLM的训练任务。具体来说,OADM设置通过考虑所有可能的解码顺序来推广LRAR,而MLM训练任务则相当于对OADM扩散过程的一个步骤进行训练


