论文地址:Learning sequence, structure, and function representations of proteins with language models
Protein-Vec:融合序列结构功能对比学习+MOE
Abstract
目前只有不到1%的蛋白有实验验证的注释信息,需要用计算方法来填补缺失的信息。使用了一个基于蛋白质语言模型的multi-aspect framework名为Protein-Vec,可以学习sequence-structure-function的表征,可以用于蛋白质序列检索和注释
Introduction
大量的基因和蛋白质基因注释方法依赖于通过同源性推断/检测的个映射注释(或注释子集),而远程同源性检测方法(inference/detection of homology)至今仍是生物信息学中最常用的工具,但是大量的蛋白是同源检测注释是失败的
通过建模不同的蛋白方面来进行计算蛋白质注释和功能预测,并在此过程中建立了一个多方面的信息检索系统,这个不同的方面包括
- protein function (encapsulated in Enzyme Commission numbers (EC) and Gene On-tology (GO) annotations
- protein domain sequence families (Pfam)
- gene domains (Gene3D)
- protein structure information (via TM-Scores)
每个aspect都有自己独立的困难和计算开销
- EC classification:最近的研究表明有40%的计算注释的酶是错的
- Pfam:人工管理的蛋白质结构域,但因为Pfam的家族包含了很多小的序列集合,这使得机器学习的方法学习变得困难
- Protein function prediction (GO) :有错误的GO注释,稀少的实验注释以及极端的多标签多分类问题
Results
将序列、结构和功能信息编码到这些蛋白质向量中中,通过使用对比学习和改进的损失函数训练混合专家神经网络
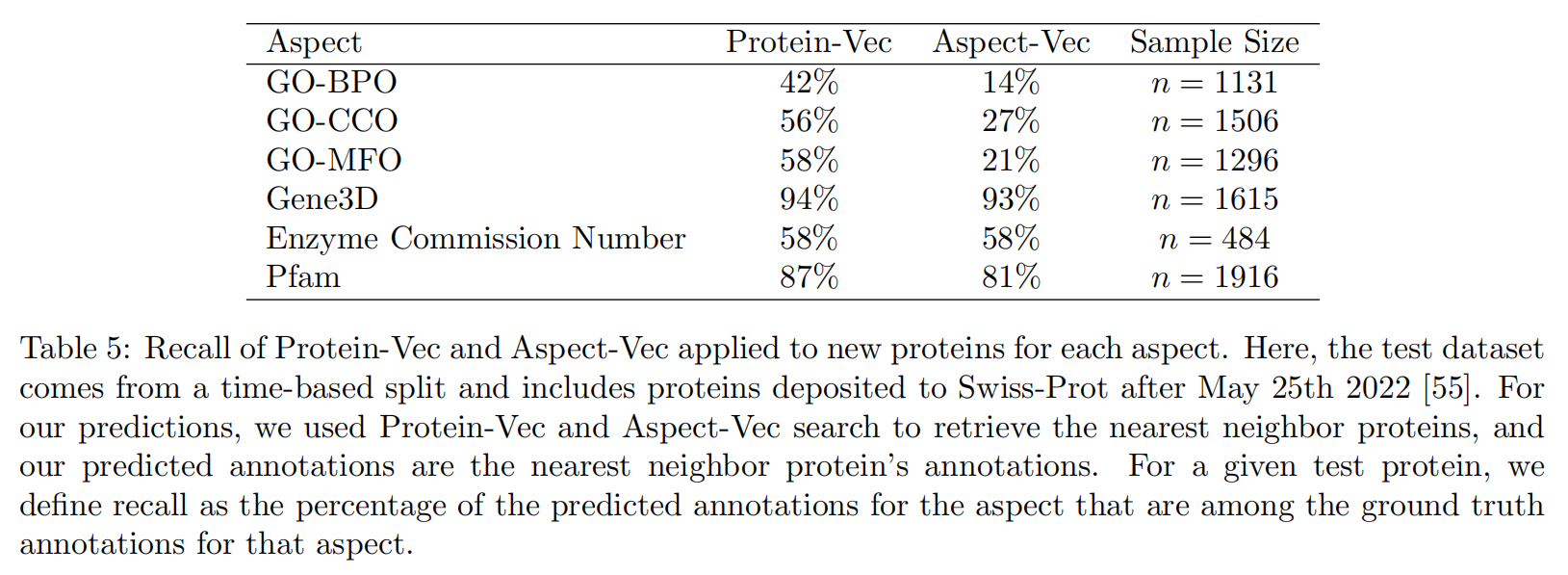
训练整个Protein-Vec模型之前,必须先训练single aspect models,称为Aspect-Vec models,包括:Enzyme Commission number (EC), Gene Ontology (GO), Protein Family (Pfam) , TM-Scores, and Gene3D domain annotations.
在训练Aspect-Vec和Protein-Vec模型的时候需要anchor protein,一个正样本和一个负样本,其中一个训练目标就是识别正负样本
每个Aspect-Vec训练完了后组合成一个multi-aspect model,Protein-Vec。创建一个矩阵把对于每个训练样本的Aspect-Vec的向量集合进去,mask没有指定的aspects,训练MOE来形成矩阵
Aspect-Vec: Single-aspect expert models
有7个:EC number, Gene Ontology molecular function (GO MFO), Gene Ontology biological process (GO BPO), Gene Ontology cellular component (GO CCO), Pfam, TM-Scores, and Gene3D domain annotations
Enzyme Commission number prediction for novel proteins

和CLEAN进行比较, Aspect-Vec model效果已经更好了
Pfam prediction for remote homologs
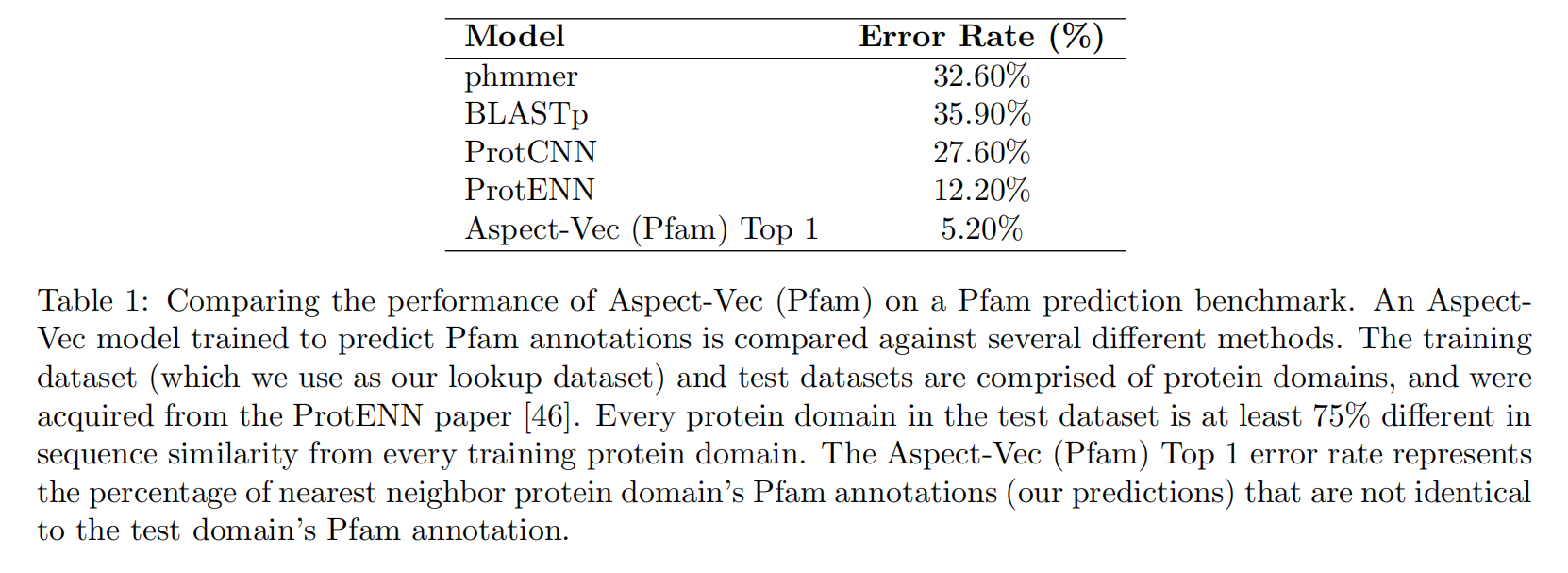
使用数据集是clustering Pfam-seed v32,是基于序列相似度低于25%
Predicting Gene-Ontology annotations for novel proteins
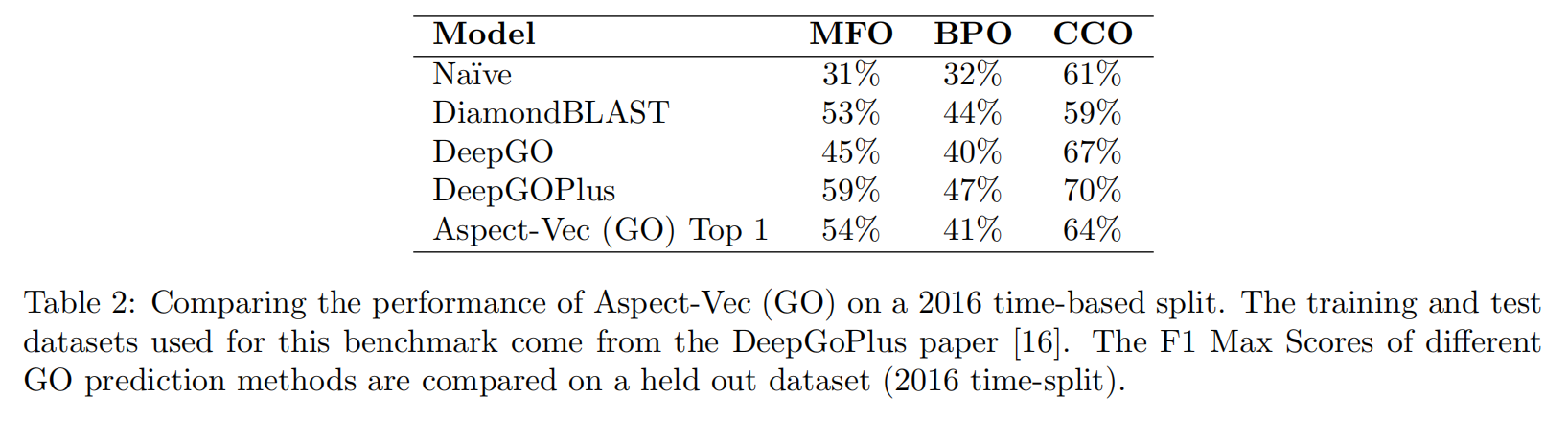
Predicting Gene3D domains: Diamond benchmark
Gene3D为蛋白质序列提供了CATH注释
DIAMOND在Diamond benchmark上有99%的top 1敏感度,TM-Vec是92%,而Gene3D Aspect-Vec model是83%
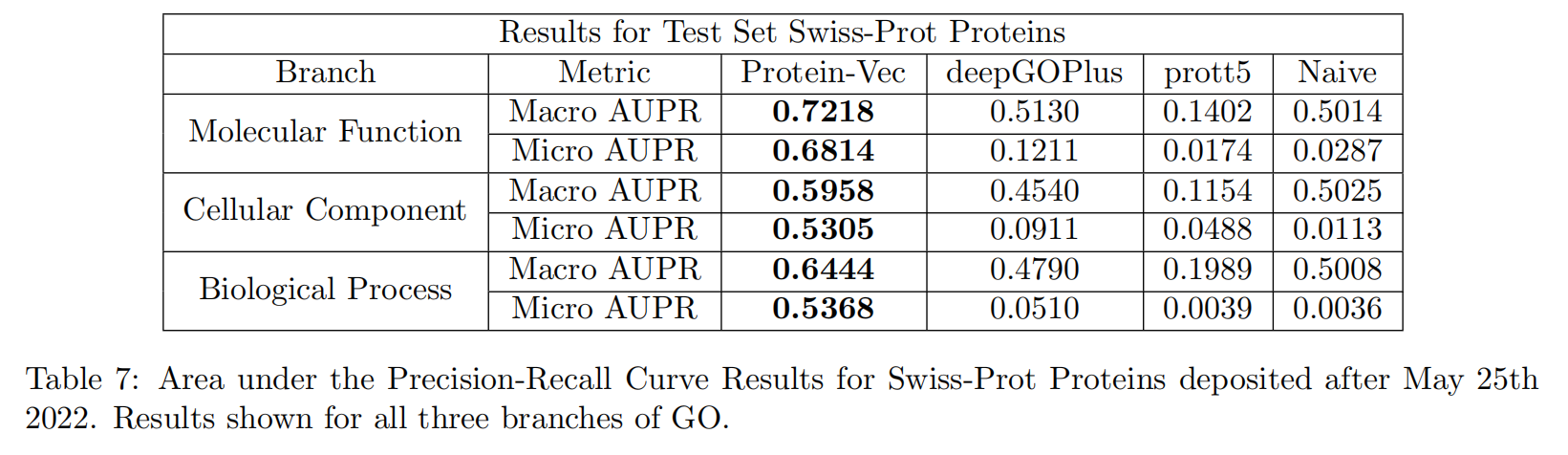
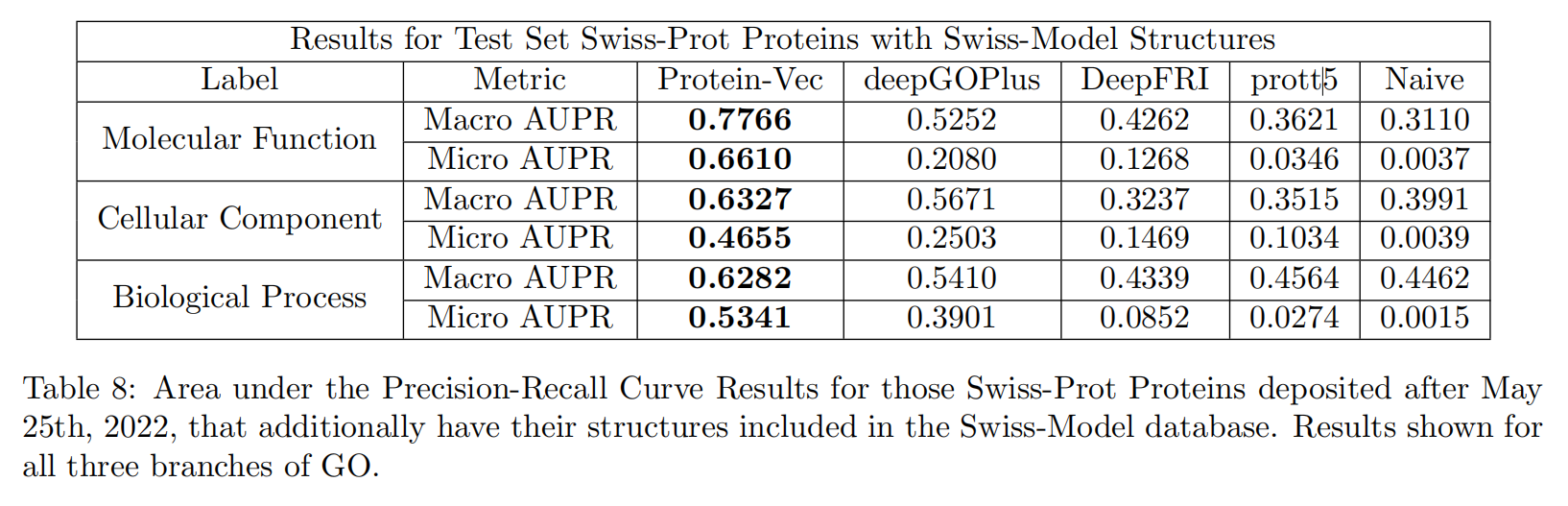
Protein-Vec: Combining sequence-structure-function aspects into one model
使用Protein-Vec编码了Swiss-Prot在May 25, 2022作为lookup database,有三个测试集:
- 基于时间分割的:在May 25, 2022之后的新蛋白
- 和训练集数据不超过50%相似度的
- 和训练集数据不超过20%相似度的
给定query,预测结果是查找数据库里最近的蛋白
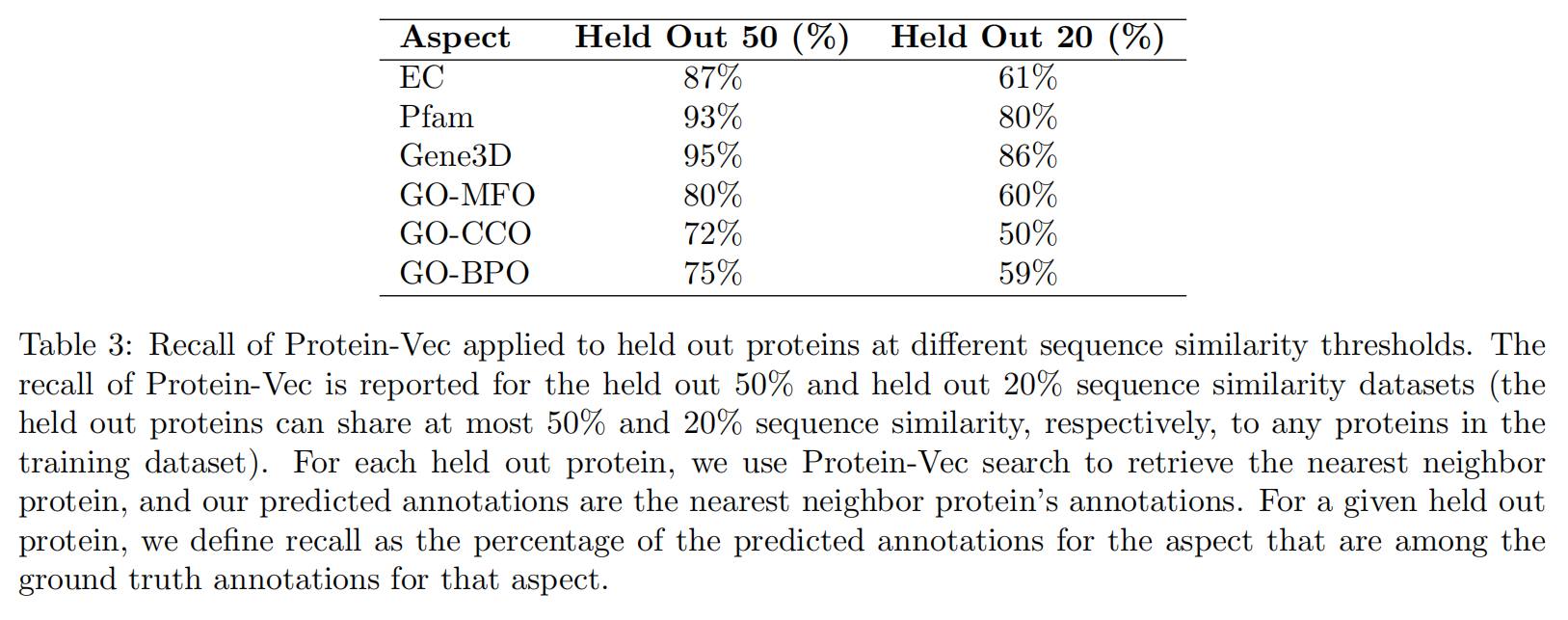
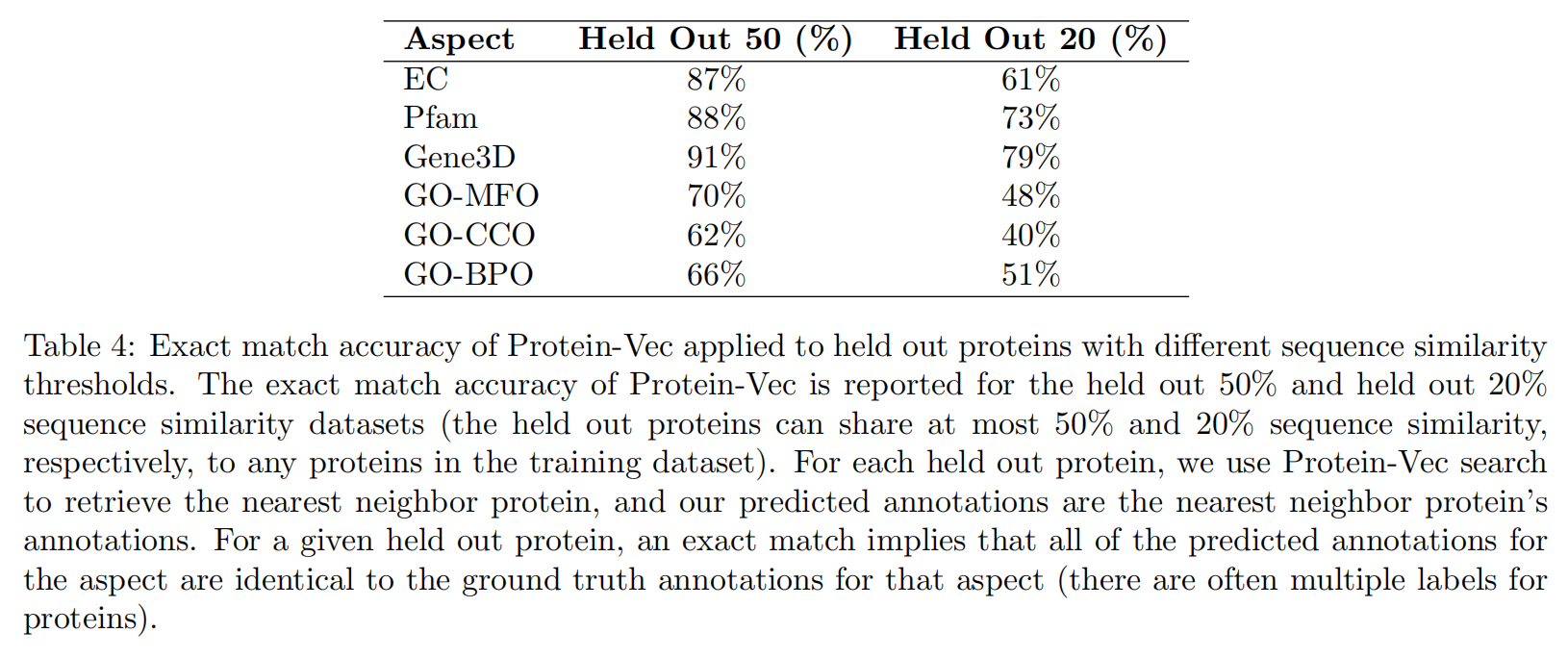
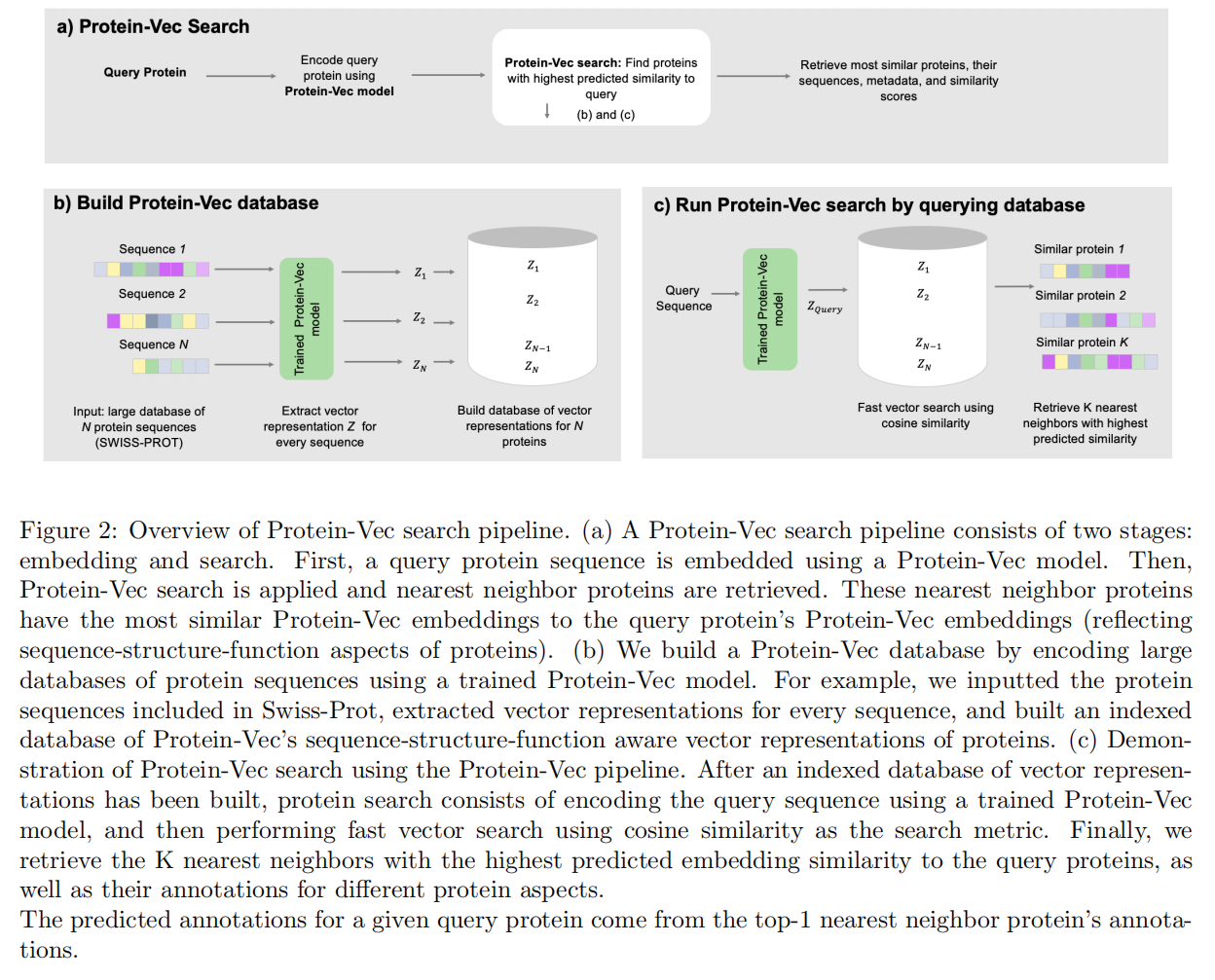
Online Methods


Aspect-Vec embedding model
每个Aspect-vec模型都是在蛋白质序列三元组上进行训练的,丢到ProtTrans (ProtT5-XL-Uniref50)里面,然后丢到Aspect-vec里面,fig3a
Protein-Vec embedding model
在训练阶段,按概率采样一些想要encode的aspect,然后生成向量,拼成一个Aspect-Vec矩阵,mask掉不需要的aspect,fig3b
使用了多目标的损失函数来训练Protein-Vec,如果训练样本有TM-score,那么还要预测TM-score
Protein-Vec search
使用FAISS作为向量搜索库
Protein-Vec dataset generation
需要生成序列、结构和功能的三方面组成anchor、正负样本的三元组,所有的数据都来源于Swiss-Prot
- 序列层面:
- Pfam和Gene3D
- 正样本:至少有一个注释和anchor一样
- 负样本:对于Pfam而言是建立hard negative,使用的Pfam clan information,有45%的负样本至少有一个相同的pfam clan;对于Gene3D,选择即没有同样的结构域,也没有同样的拓扑结构的
- 结构层面:
- 使用TM-align计算TM-score
- 功能层面:
- EC number和GO
- EC number:至少有一个EC number的4个层级一样就是正样本;有2,3个一样,但整体不一样就是负样本
- GO:使用GOGO工具生成三元组,对于每个GO component,正样本是有高的GO语言相似度的,负样本是低的,正样本是0.65,负样本0.25
最后有450M的三元组训练样本


