论文地址:Machine Learning Applied to Predicting Microorganism Growth Temperatures and Enzyme Catalytic Optima
Tome:机器学习二肽预测菌OGT和酶温度
Abstract
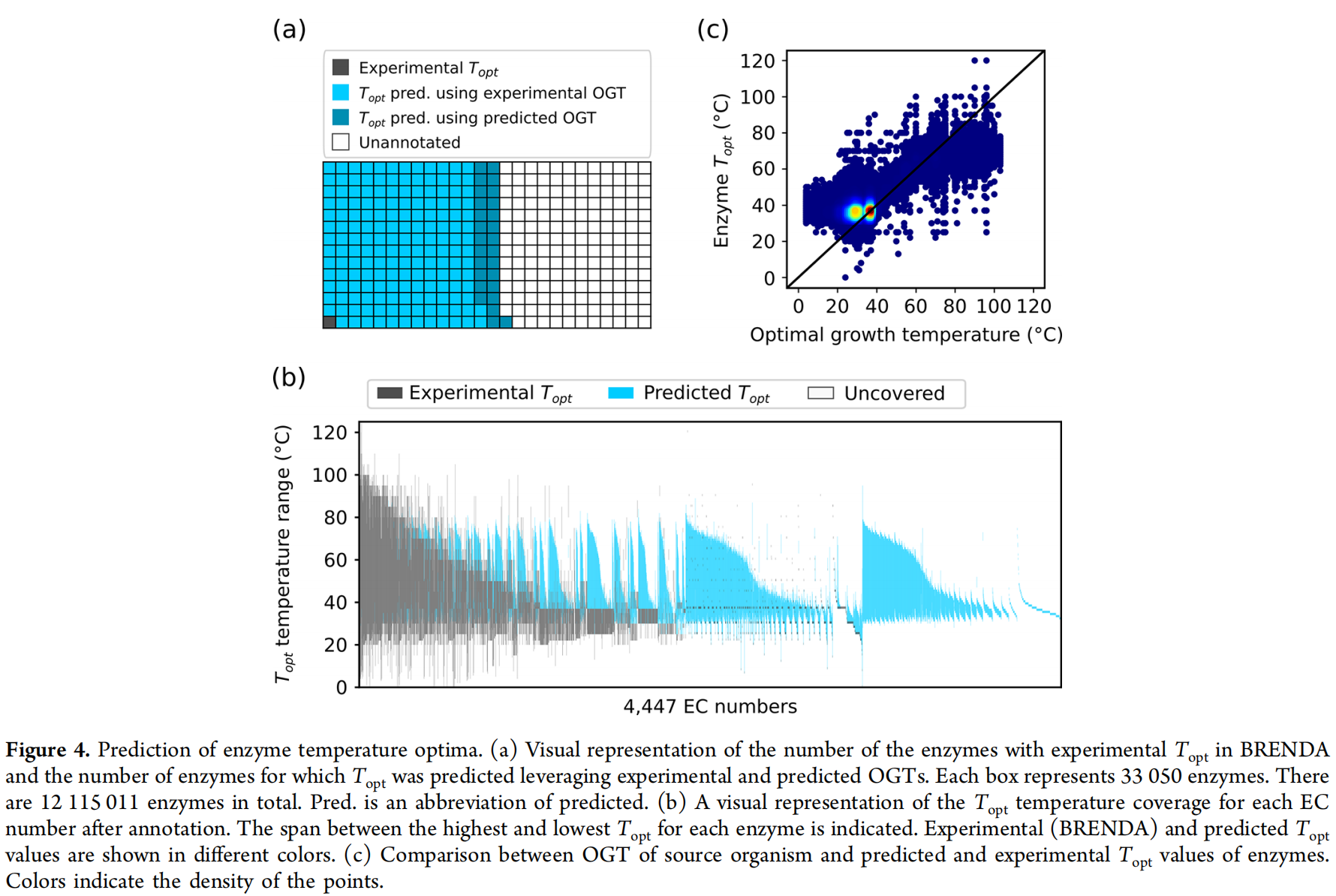
机器学习方法直接从细菌、古细菌和微生物真核生物的蛋白质组范围内的2-mer氨基酸组成中准确预测OGT。然后使用OGT数据结合酶的氨基酸序列,开发第二个机器学习模型用于预测酶的最佳催化温度(Topt)。预测了650万种酶的Topt,包括4447种酶类,并将得到的数据集提供给研究人员
Introduction
使用OGT数据面临着两项挑战:
- 很多实验的OGT数据存在大量私人数据不能轻易的获取,并且实验流程繁琐
- OGT预测酶是很粗糙的,酶可能比OGT高或者低很多,单个酶和OGT的spearman相关性只有0.48,嗜热菌的酶可能比想象中最适温度更低
使用氨基酸或2-mer氨基酸的组合预测OGT是可能的,比如Zeldovich发现,在由204个古细菌和细菌蛋白质组组成的数据集中,7个氨基酸I、V、Y、W、R、E和L的和分数与OGT的相关系数高达0.93
大部分的工作都是预测蛋白质突变的稳定性,少数工作预测蛋白质的热稳定性
本文预测蛋白质的温度为以下三步:
- 建立一个准确预测OGT的机器学习模型
- 使用OGT和序列信息提升预测酶稳定性的准确度
- 预测6.5M酶
Results and Discussion
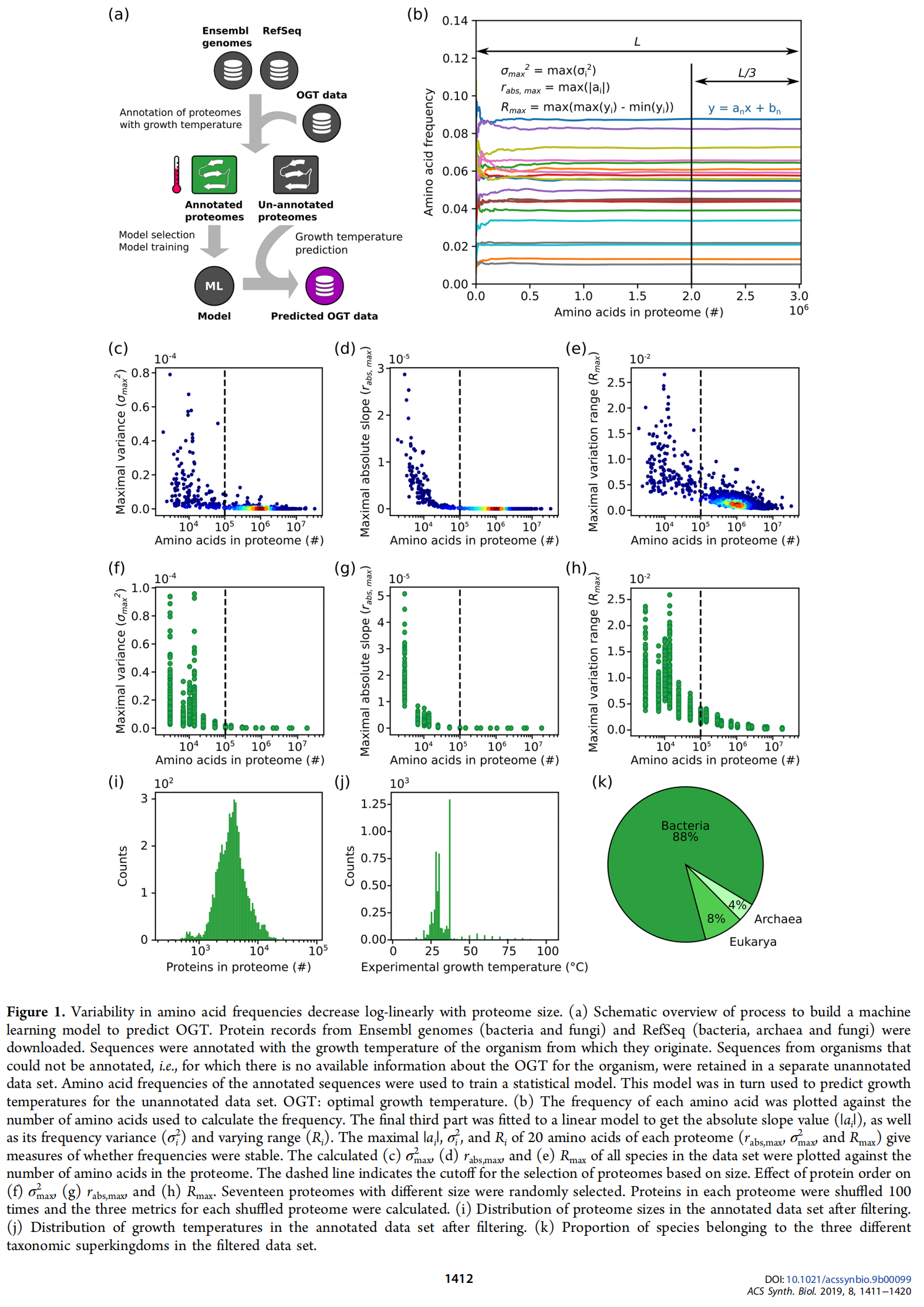
Collection of Optimal Growth Temperature and Proteomes of Microorganisms
从 https://doi.org/10.5281/zenodo.1175608 下载OGT数据
对于注释和未注释数据集中的每个生物体,我们计算了全局氨基酸单体和二肽频率,然而,数据集中的一些生物体只包含少量的蛋白质序列,因此从这些序列中获得的氨基酸组成可能不能代表完整蛋白质组的真实氨基酸组成。由于不清楚需要多少蛋白质序列才能获得稳定的氨基酸组成,设计了三个不同的指标来测试需要多少蛋白质序列数据才能获得稳定的氨基酸组成
OGT Can Be Accurately Predicted from Amino Acid Composition of the Proteome
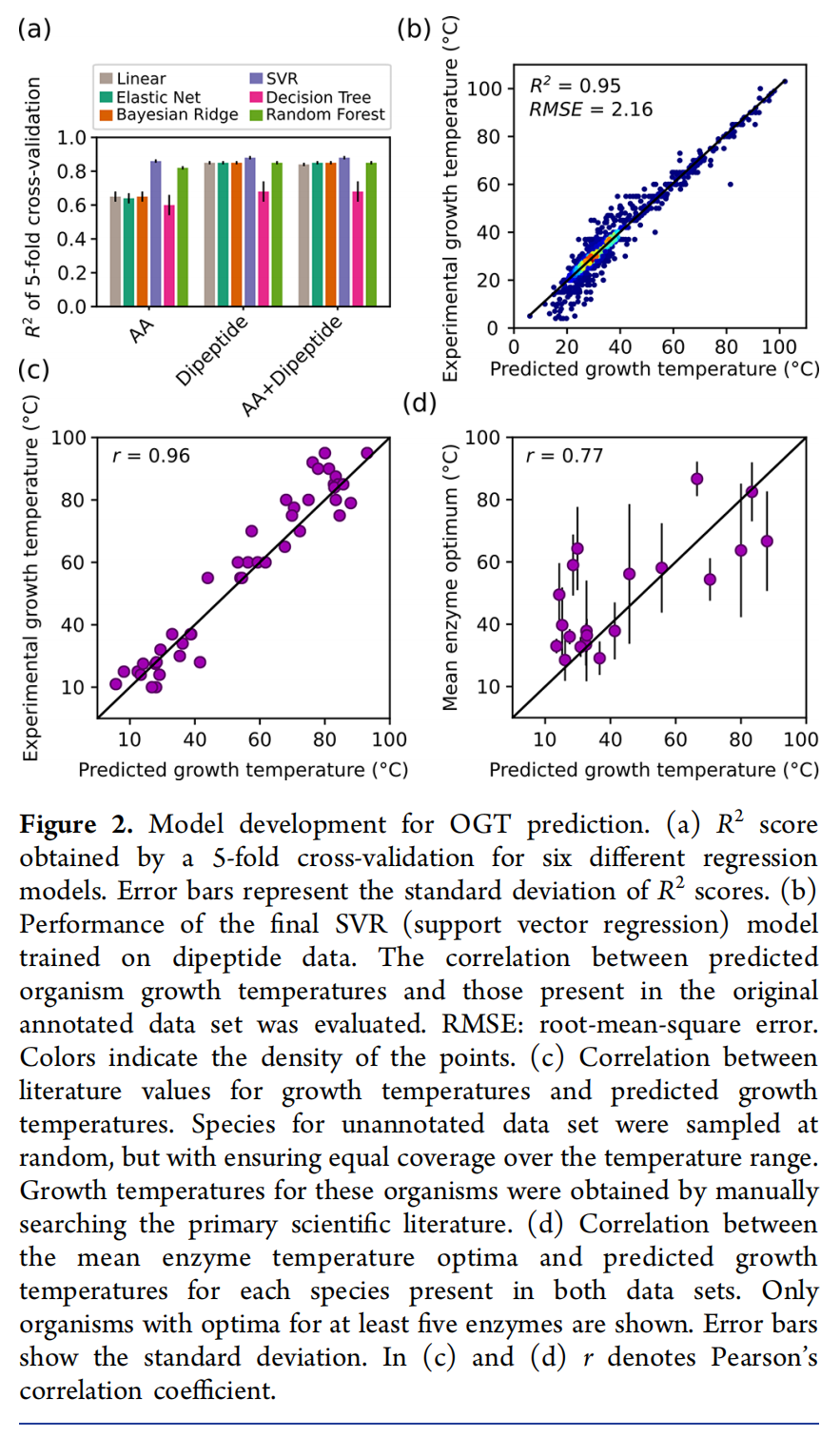
非线性模型远好于线性模型
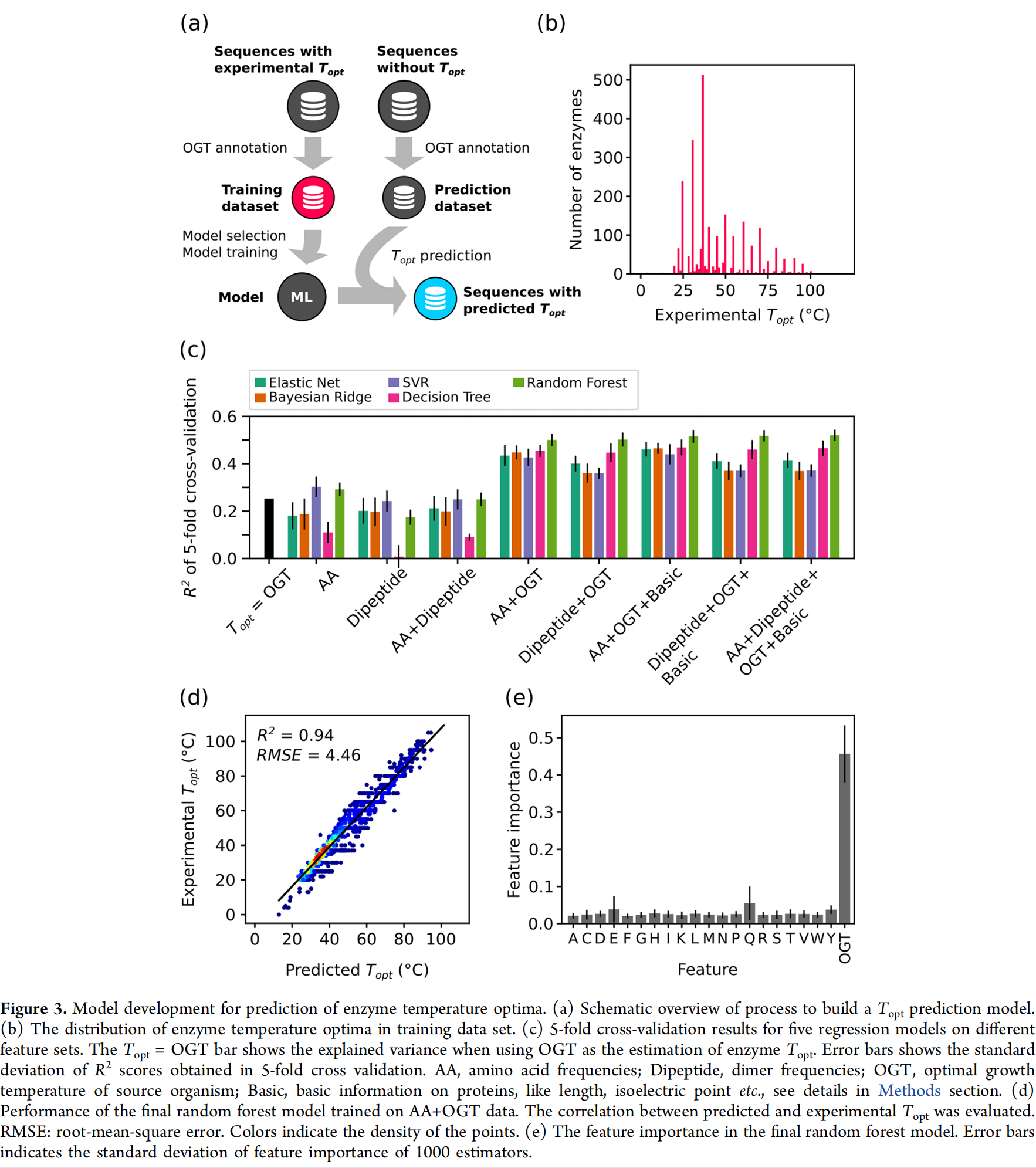
Improved Estimation of Enzyme Temperature Optima Using Machine Learning
Annotating Enzymes in BRENDA Using OGT and Predicted Topt
Methods
Estimation of Threshold
- 残基数与氨基酸频率线性回归的绝对斜率值(| ai |)
- 所选频率的频率方差(σi 2)
- 变化范围(Ri),最大频率与最小频率的差值(i表示20个氨基酸中的每个)
Machine Learning Workflow for OGT Model
使用单个氨基酸频率(AA)、二肽频率(二肽),或两个同时使用(AA+二肽):线性回归(线性)、bayesian ridge、elastic net、决策树、支持向量回归(SVR)和随机森林
Machine Learning Workflow for Topt Model
- 20个氨基酸频率(AA)
- 400个二肽频率(二肽)
- 其源生物的OGT
- 基本特征包括蛋白质长度、等电点、分子量、芳香性、instability index、gravy和三个二级结构单元的部分:螺旋、转和片。


