论文地址:Fast protein structure searching using structure graph embeddings
Progress:图神经网络+有监督对比学习
Abstract
相较于一级序列搜索,结构搜索能更有益于同源检测,功能注释和蛋白分类,一个快速准确的检索方法能够对Bioinformatics非常有效。训练了一个简单的图神经网络,使用有监督对比学习来获得蛋白质结构的低维度表征的方法名为Progres,准确率能和最好的方法比较,同时在AFDB中搜索TED domains使用CPU只用1/10秒
Introduction
蛋白质结构往往比序列更加保守,因此可以用于远端同源检测,蛋白质分类,推理功能等方面。目前最高精度的方法是基于坐标的比较比如Dali,但是数据库一旦变大就很慢
使用GNN方法在pre-embedded数据库进行余弦相似度计算是很快的,使用了有监督对比学习方法反应出对于蛋白质空间距离的理解
Results
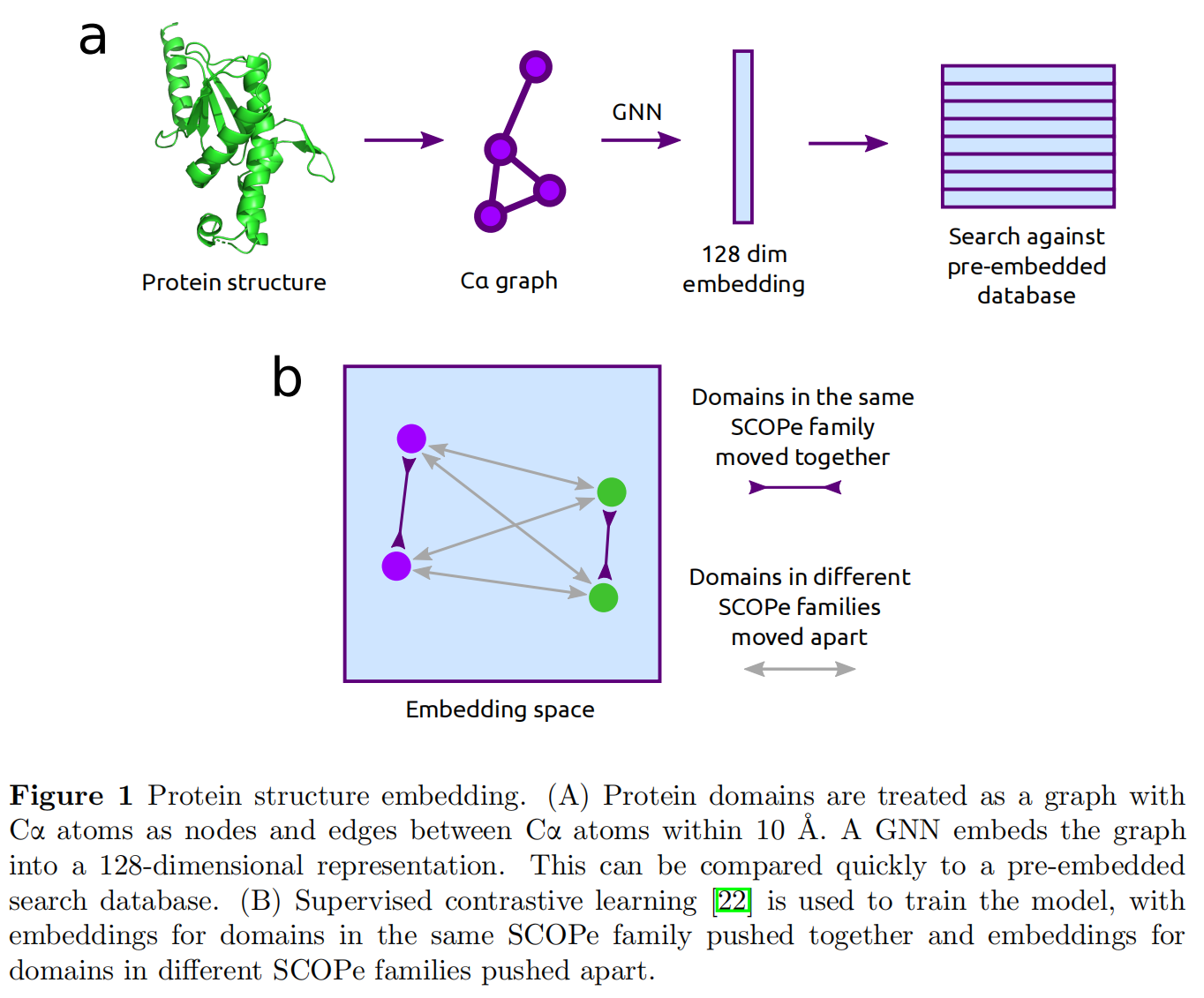
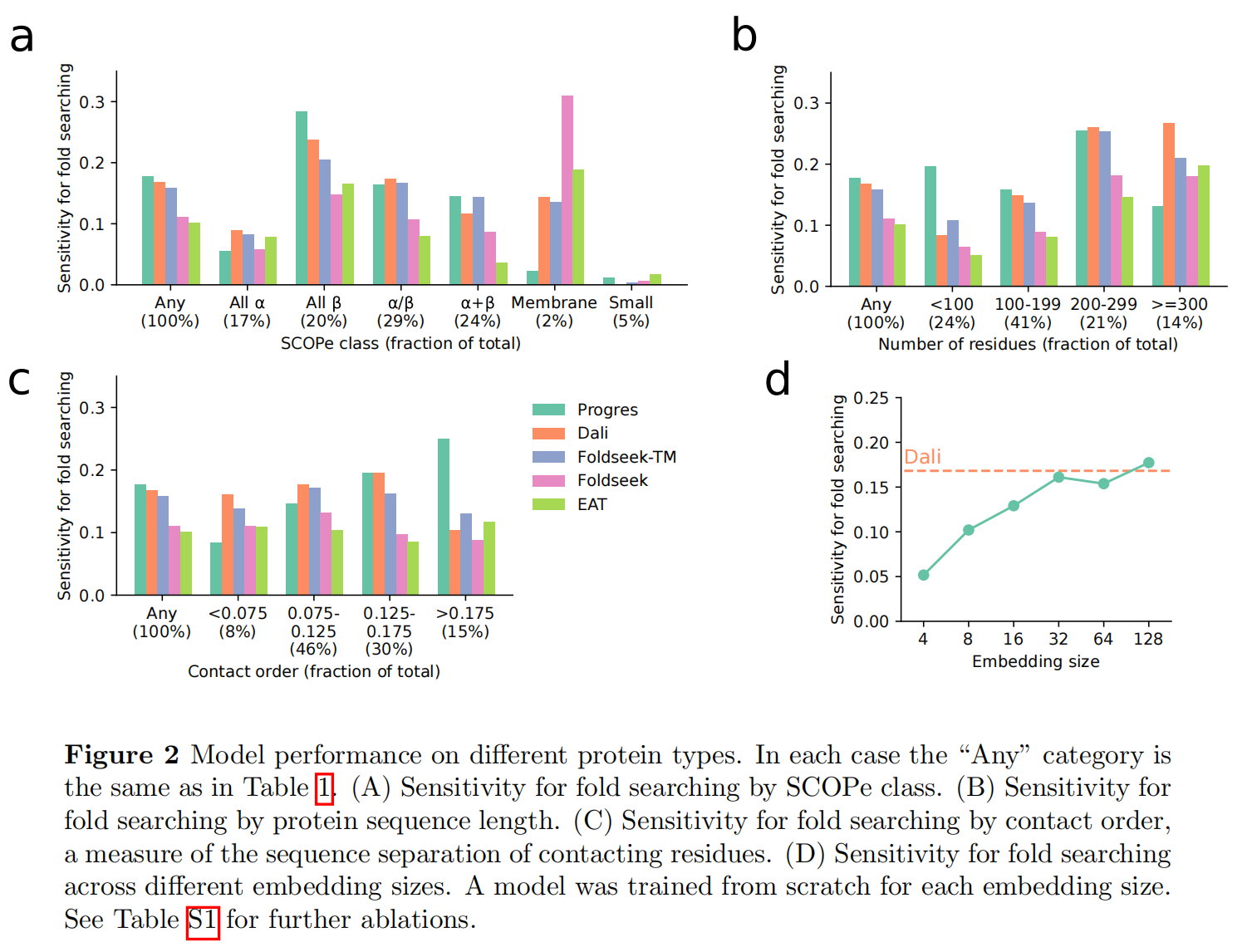
在SCOPe domains里面做有监督对比学习,使用 Sinusoidal position encoding,但是该方法无法给出结构alignment
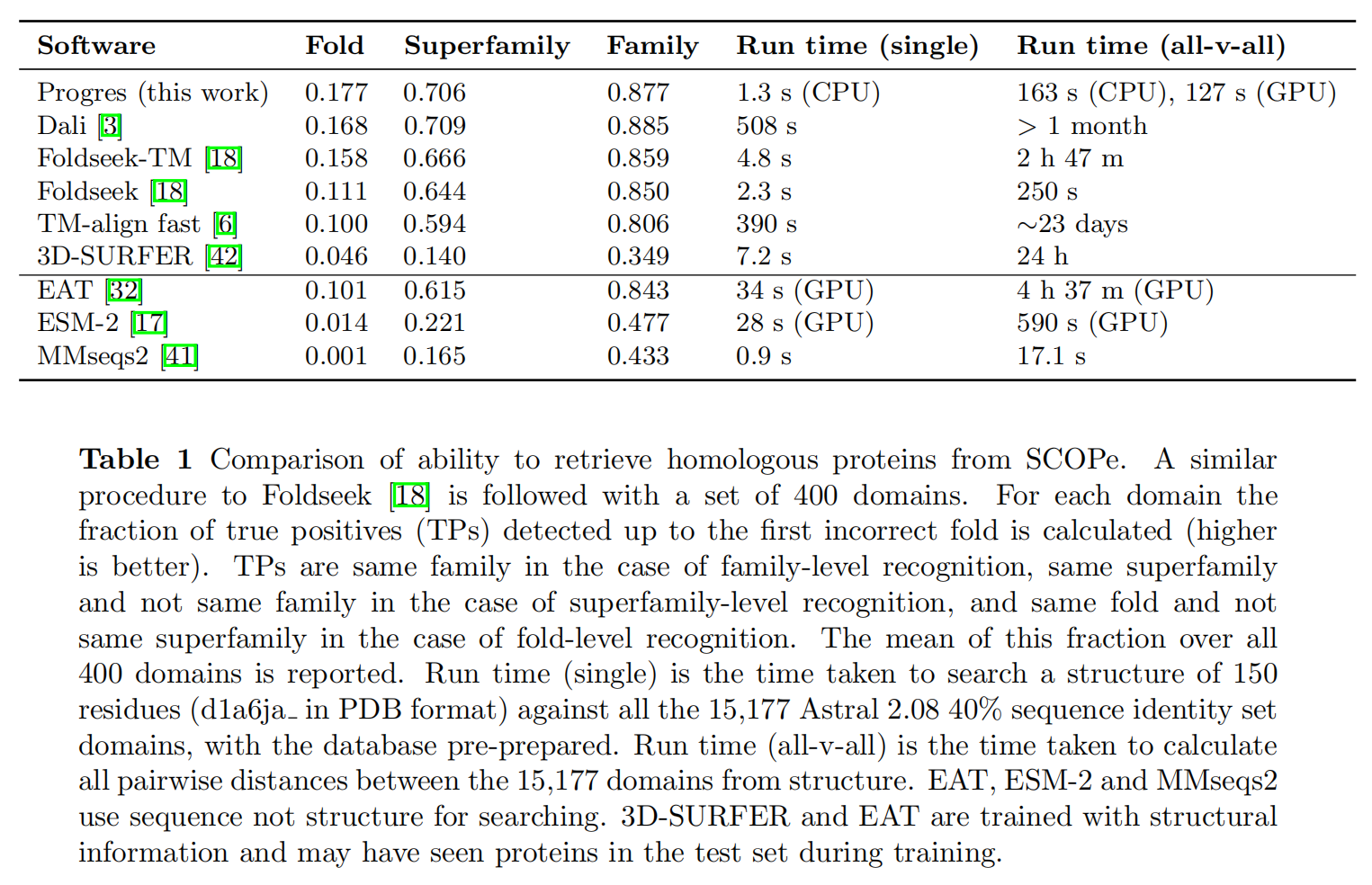
测试集是从Astral 2.08 40%的序列标识集中选择了一个随机的400个域集来进行测试,保持和SCOPe的独立且相似度低于30%
最近TED study将ADFB拆分出了domains,将domains进行50% sequence identity聚类后的embedding用FAISS进行向量检索,一共是53M的结构
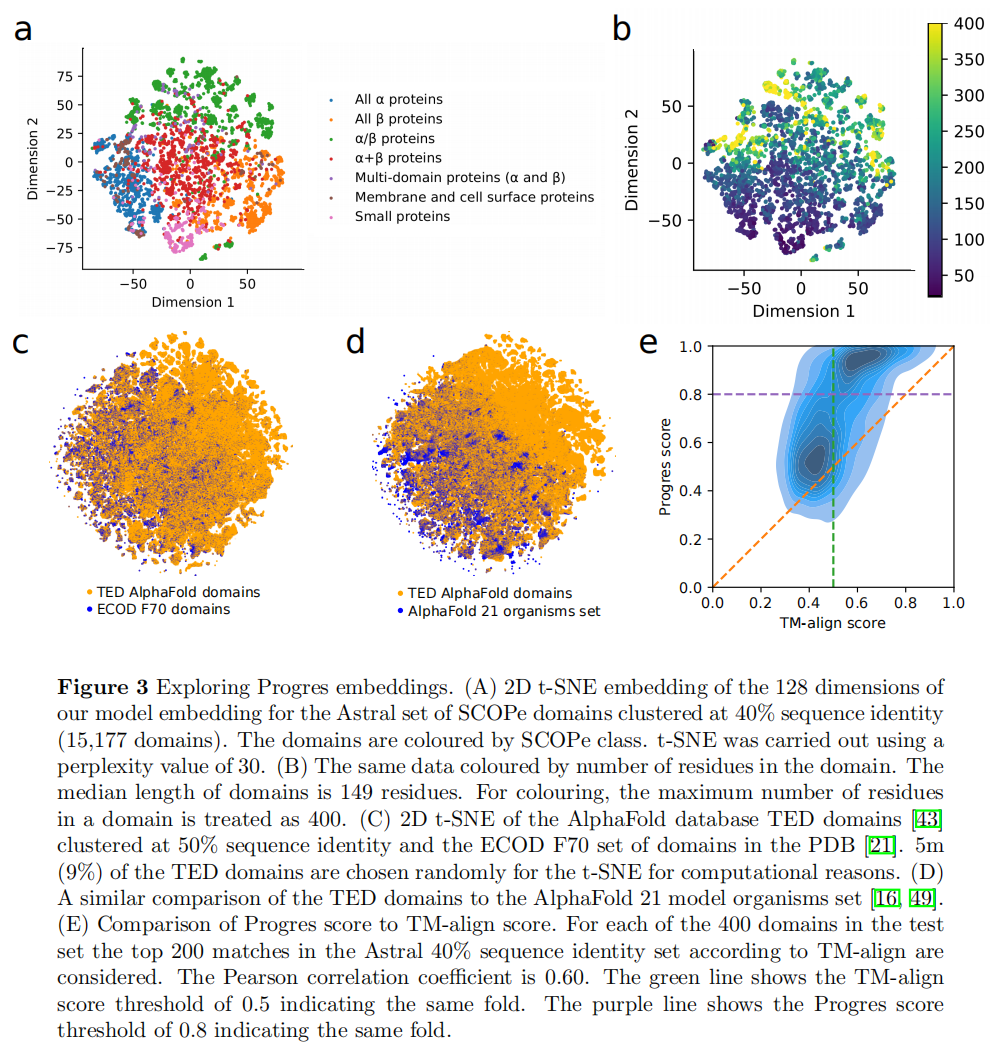
Discussion
模型是在domain上进行训练的,所以不能处理多个domain,长无序区域或复合物,需要依赖于一些现有方法比如Merizo,SWORD2和Chainsaw将query structures拆分为domain
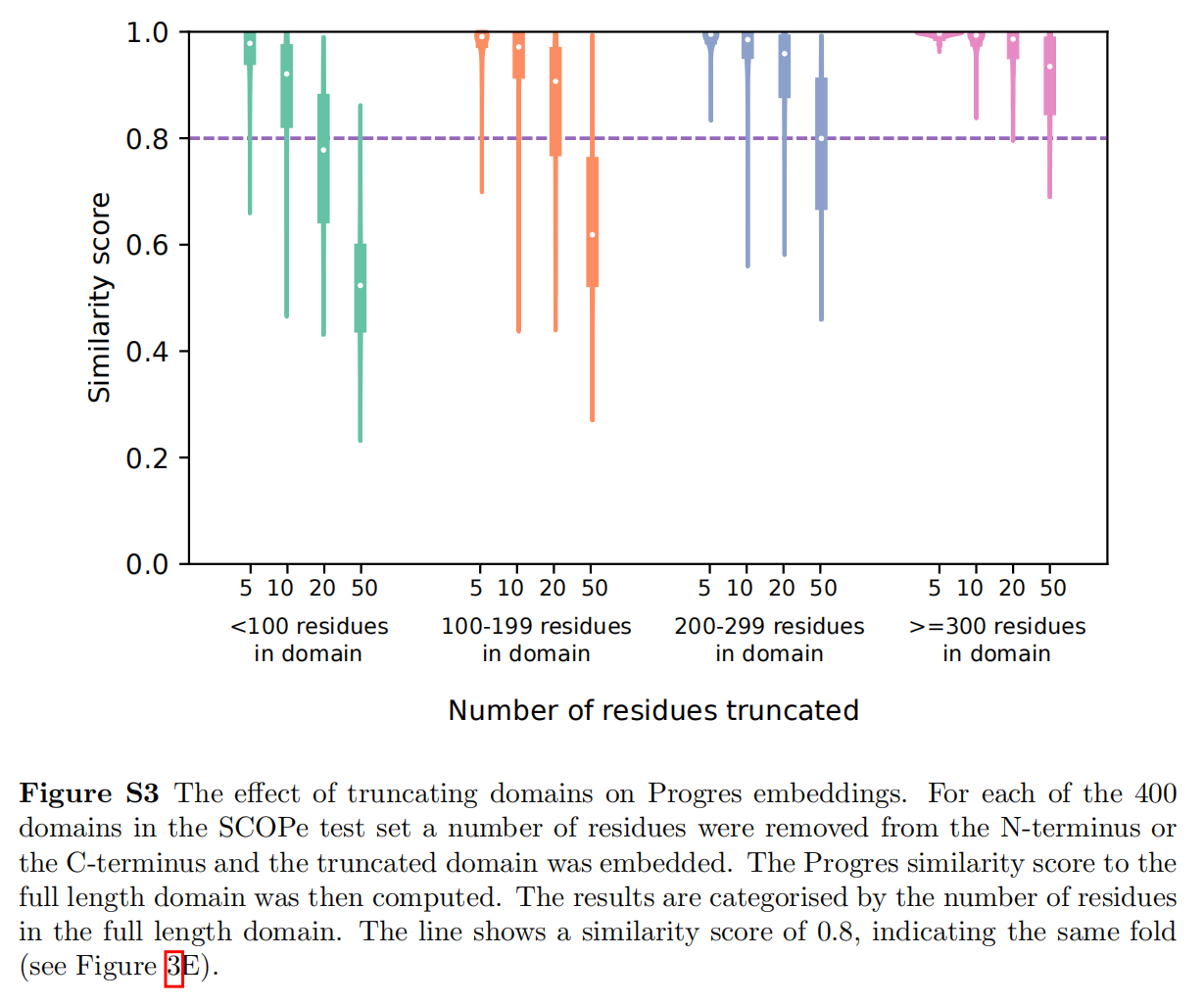
figure S3表示移除多少个N端或C端的氨基酸后将截断的domain embed后进行比较,这表明在domain边界问题上问题不大
Methods
Training
使用Astral 2.08 95% sequence identity set包括不连续的域进行训练,从Astral 2.08 40% sequence identity set随机选了400个domain做测试,还有200个验证。使用MMseqs2删除了这600个结构域中30%或以上的结构域,也删除了残基少于20或超过500的结构域。这使得4,862个famliy中的30,549个领域训练
Cα用10A作为节点并带有以下特征:10A内的Cα原子数除以蛋白质中最大的Cα原子数,Cα原子是否在n端,Cα原子是否在c端,以及对结构域中残基数的64维正弦位置编码
使用的模型是EGNN,边是10A以内,6层,用的sum pooling
对于每个蛋白质家族,随机选择5个其他家族,对于这6个家族,每个家族随机选6个蛋白质,少于6个就复制到6个,将6个label的36个domain输入模型,对比学习的temperature设置为0.1。训练的时候在每个Cα原子的x、y和z坐标上加入方差为1.0A的高斯噪声
使用Adam优化器,5e-5学习率和1e-16的weight decay,训练500 epochs
Testing

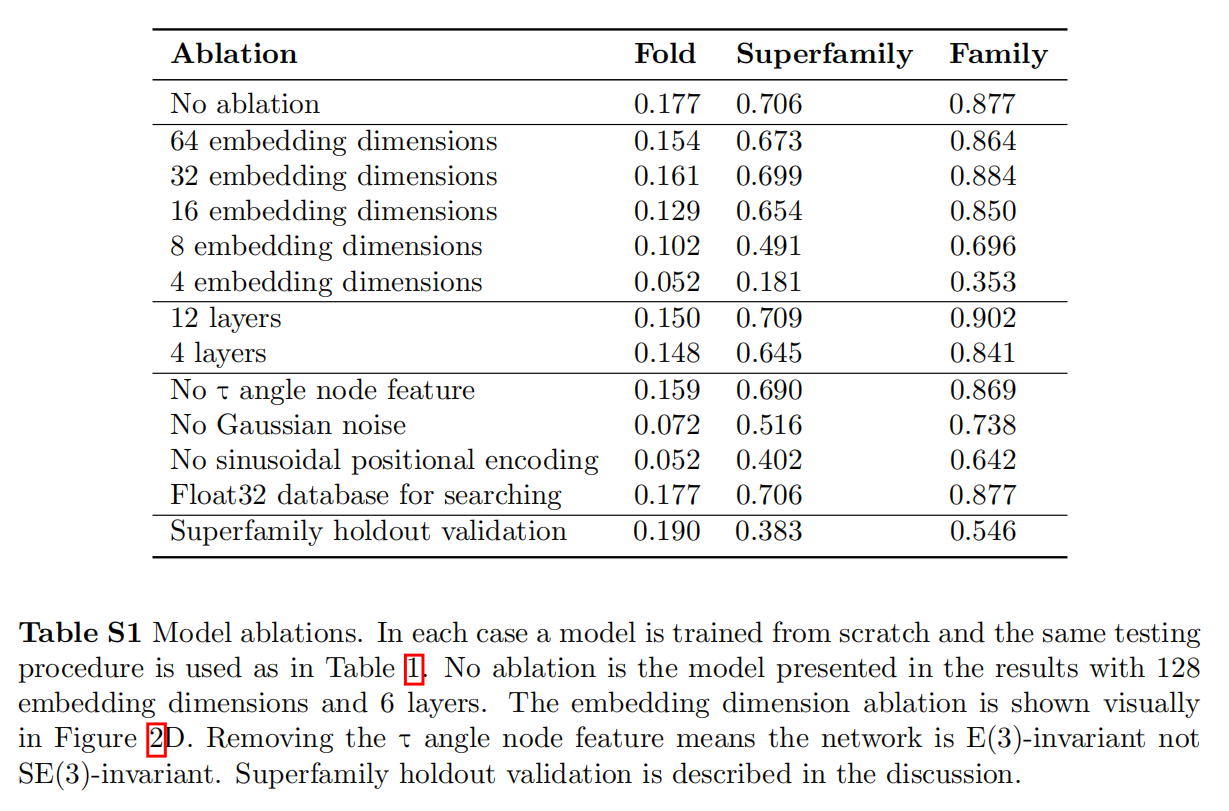
15177个Astral 2.08 40% sequence identity的domain用模型进行嵌入,从table S1可以看到fp16对准确率没什么影响
对于400个domain,计算和 15177 domains的余弦相似度,评估TPs直到第一个错误的fold检测到


