论文地址:PLMSearch: Protein language model powers accurate and fast sequence search for remote homology
论文实现:https://dmiip.sjtu.edu.cn/PLMSearch and https://github.com/maovshao/PLMSearch
PLMSearch:同源家族过滤+蛋白质相似度预测+PLMAlign
Abstract
从序列当中检测结构同源性依旧是挑战,因此提出了PLMSearch,只需要使用序列作为输入。实验显示PLMSearch可以在数秒内搜索数百万的query-target蛋白,并且和最好的结构搜索方媲美,还可以召回出序列不同结构相似的远程同源对
Introduction
基于序列搜索的方法很难检测出远距离的进化依赖,在序列相似度低于0.3的时候,使用sequence profile (MSA)的方法,如HMMER, HHsearch或HHblits更好
因为结构比序列的差异更大,因此结构相似性的搜索更加敏感,主要分为三种:
- contact/distance map-based: Map_align, EigenTHREADER, and DiscoVER
- structural alphabet-based: 3D-BLAST-SW, CLE-SW, Foldseek and Foldseek-TM
- structural alignment-based: CE, Dali, and TM-align
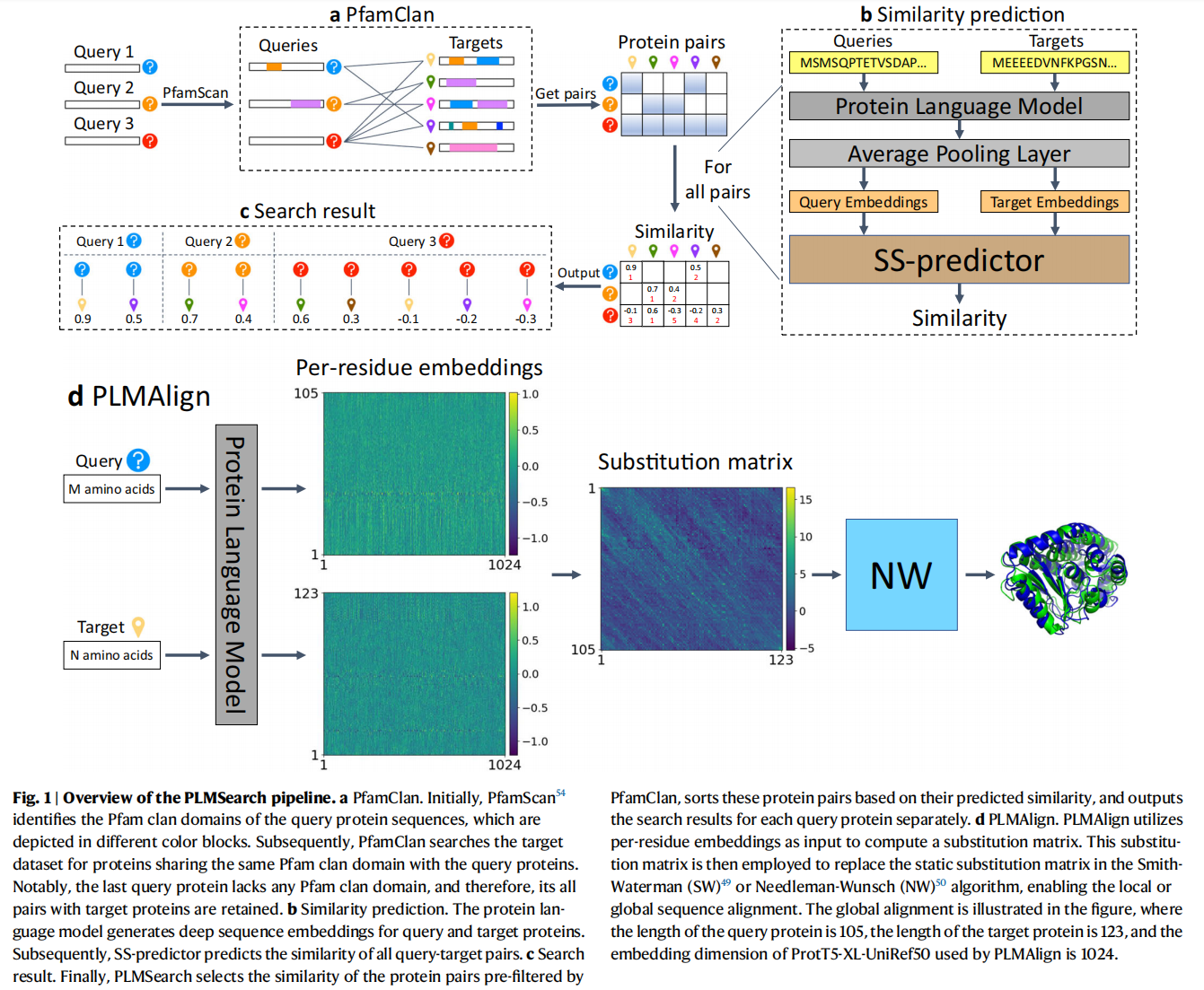
PLMSearch主要包含以下三步:
- PfamClan过滤出具有相同pfam clan domain的蛋白质对
- SS-predictor (structural similarity predictor)预测query-target pairs的相似性,使用TM-score作为ground truth训练
- PLMSearch根据预测的相似度对PfamClan对预过滤的对进行排序,并相应地输出每个查询蛋白的搜索结果。随后,PLMAlign为PLMSearch检索到的顶级蛋白质对提供序列比对和比对得分(图1d)
Results
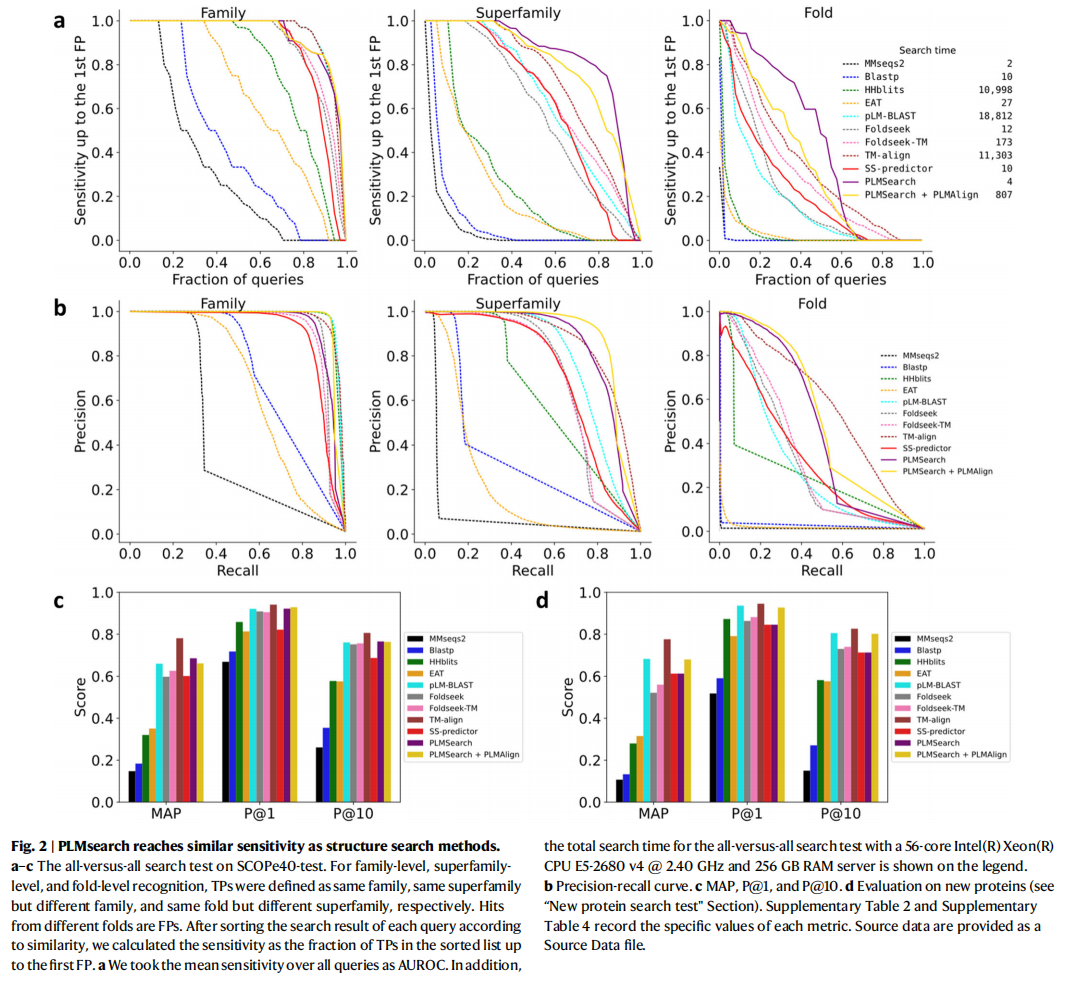
PLMsearch reaches similar sensitivity as structure search methods
检索测试在SCOPe40-test和Swiss-Prot上显示出PLMSearch是达到了准确率和速度的最佳平衡
SCOPe40-test (2207 proteins)合计 4,870,849 query-target pairs,图2a-c,Table S2
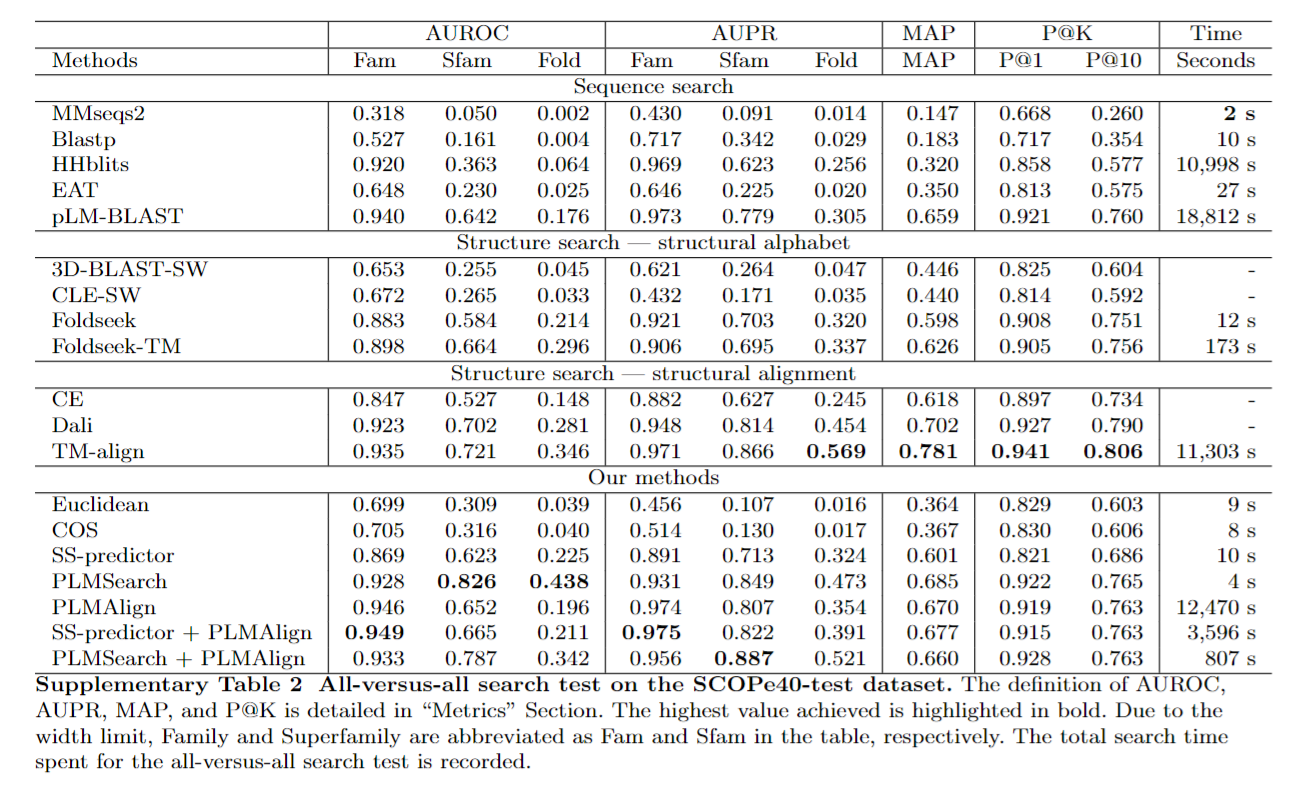
Swiss-Prot search test随机从Swiss-Prots选了50个query,SCOPe40-test中选了50个query,在Swiss-Prot中430,140 target中检索,Fig S2, Table S1
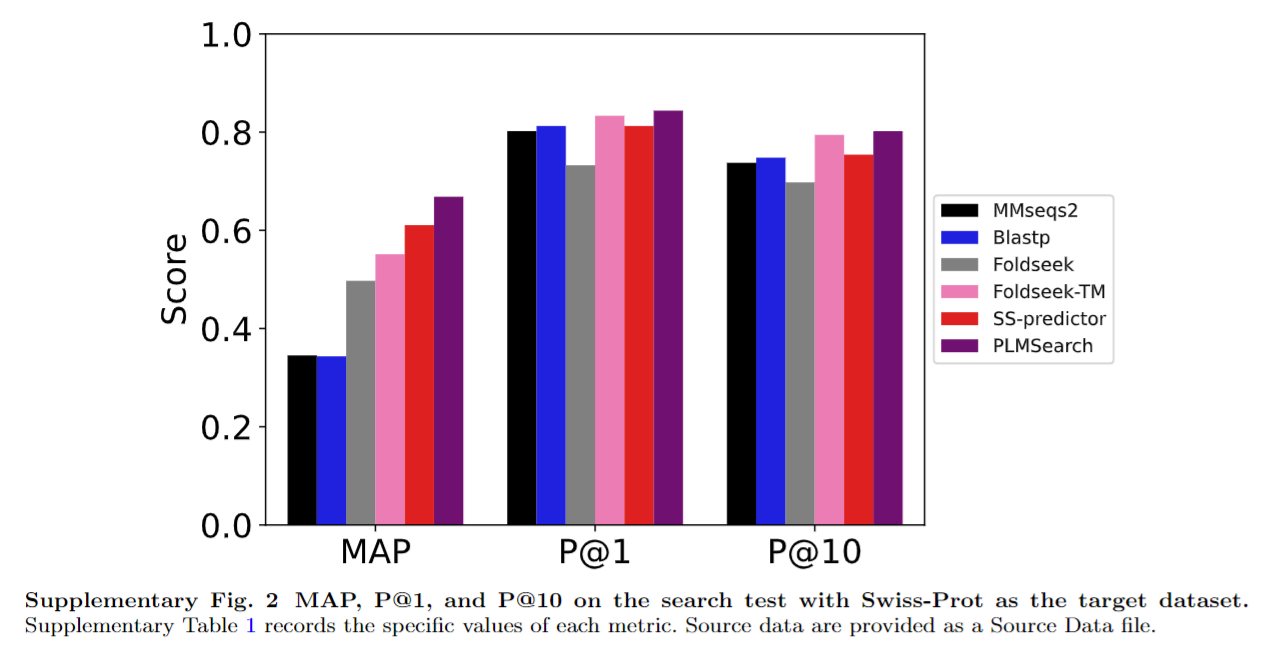
PLMSearch searches millions of query-target pairs in seconds
使用了相同的计算资源(一个56核的Intel ® Xeon ® CPU E5-2680 v4 @ 2.40 GHz和256 GB RAM服务器)
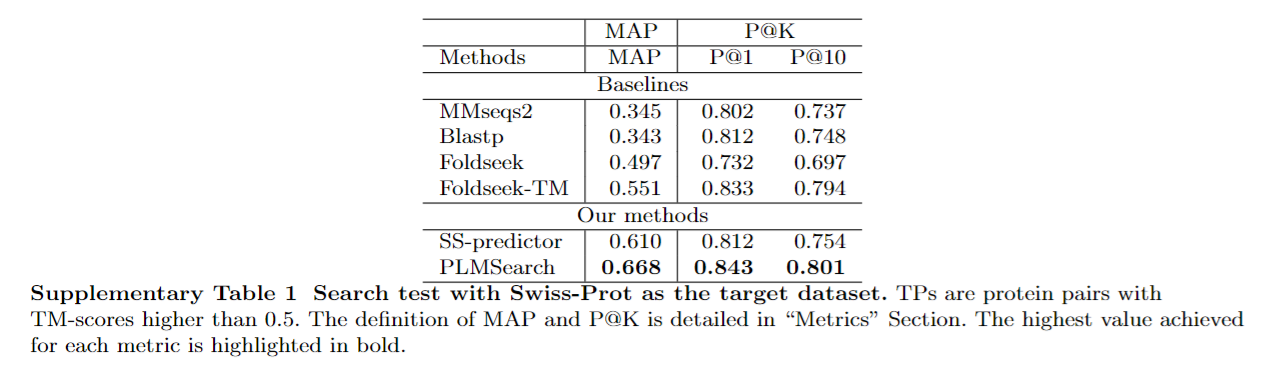
PLMSearch accurately detects remote homology pairs
序列相似度>0.3且TM-score>0.5称为easy pair,序列相似度<0.3且TM-score > 0.5称为remote homology pair
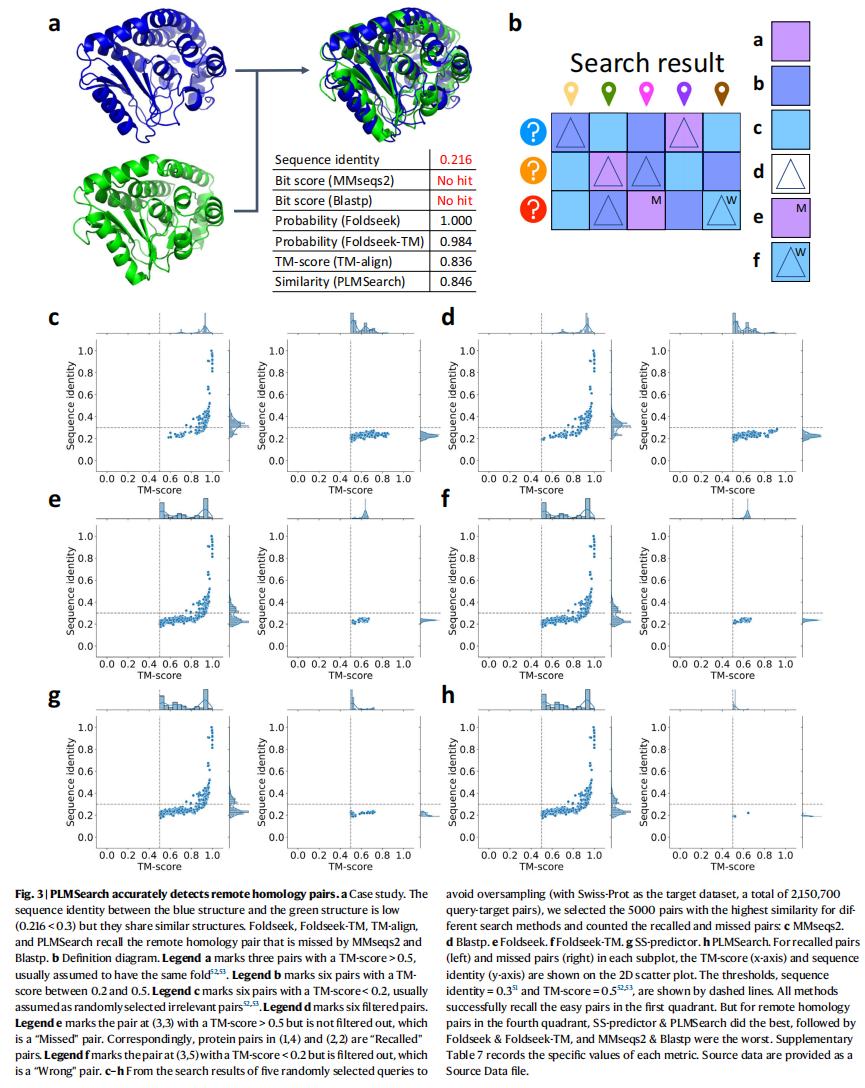
Ablation experiments: PfamClan, SS-predictor, and PLMAlign make PLMSearch more robust
为了评估没有PfamClan组件的PLMSearch,从SCOPe40测试中的2207个查询中总共筛选了110个查询,这些查询没有扫描任何pfam域
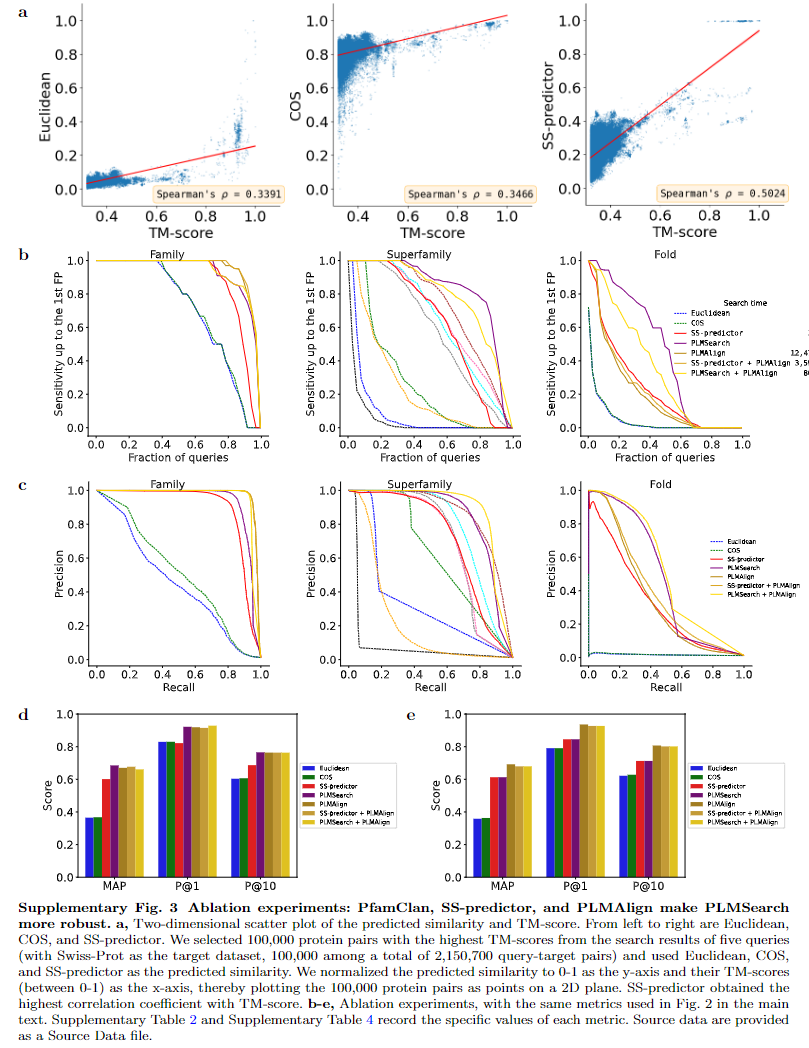

为了评估没有 SS-predictor 组件的 PLMSearch,我们首先基于 PfamClan 对 SCOPe40 测试和 Swiss-Prot 数据集进行聚类
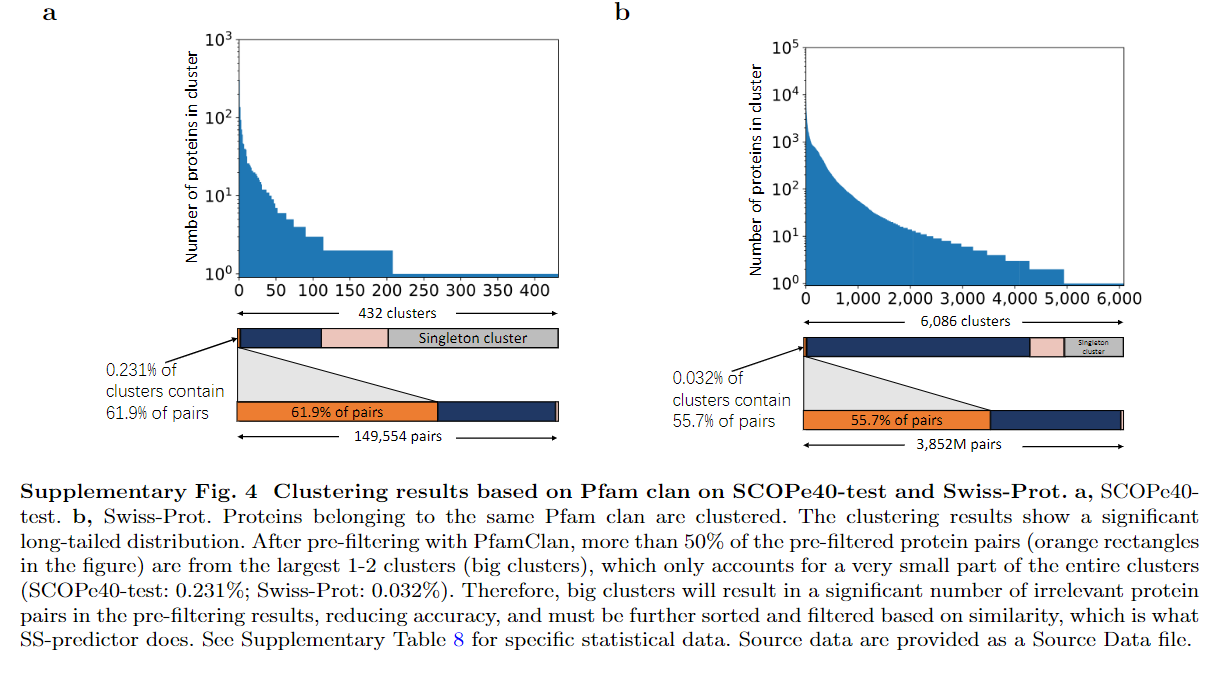
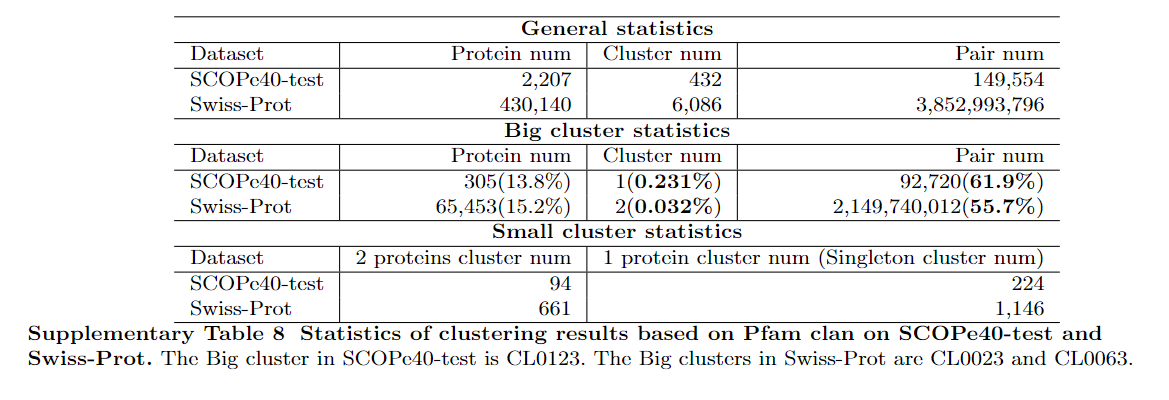
聚类结果显示出很明显的长尾效应,经过PfamClan的预过滤后,超过50%的预过滤的蛋白质对(图中的橙色矩形)来自最大的1-2个簇(大簇),因此,大的聚类会导致预过滤结果中产生大量的不相关的蛋白质对,降低准确性,必须根据相似性进一步进行分类和过滤,这就是ss-predictor所做的。
Methods
PLMSearch pipeline
1.PfamScan检索出Pfam clan domains; 2.相似度预测;3.相似度排序;4.对于高排名的对,PLMAlign生成对比
PfamClan
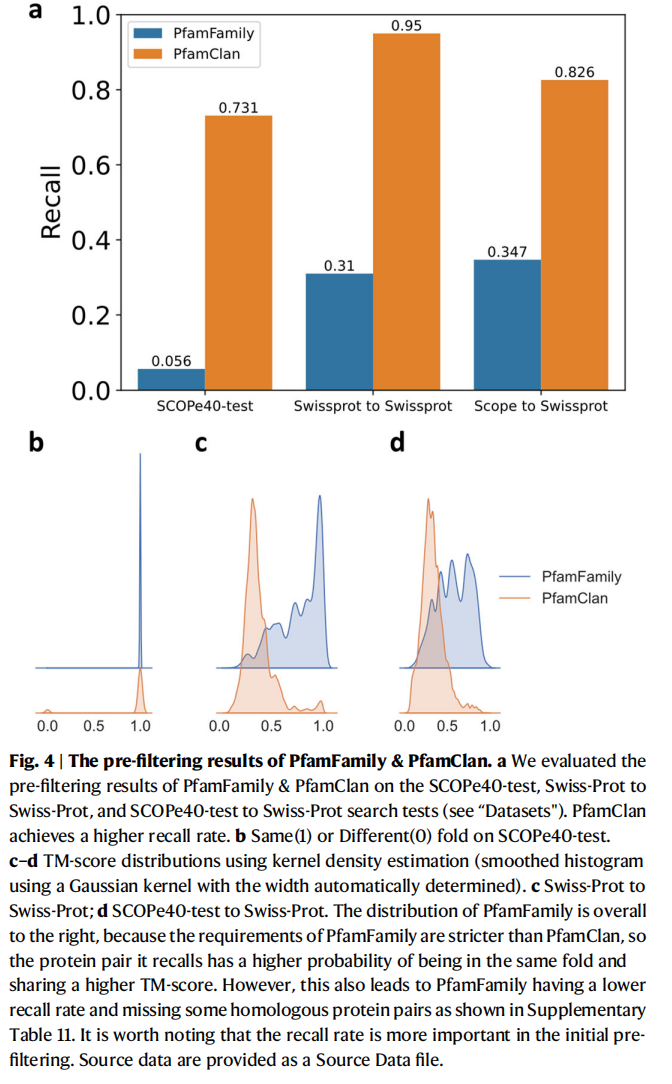
PfamClan过滤出共享相同Pfam clan结构域的蛋白质对(图1a)。值得注意的是,召回率在初始预过滤中更为重要。PfamClan基于一个更宽松的标准,共享相同的pfam clan域,而不是共享相同的pfam fmaliy(pfam家族所做的)。这一特性使得PfamClan在召回率上优于PfamFamily(图4)
Similarity prediction

将蛋白质序列输入ESM-1b,预测TM-score,在SCOPe40-train和CATHS40上进行训练
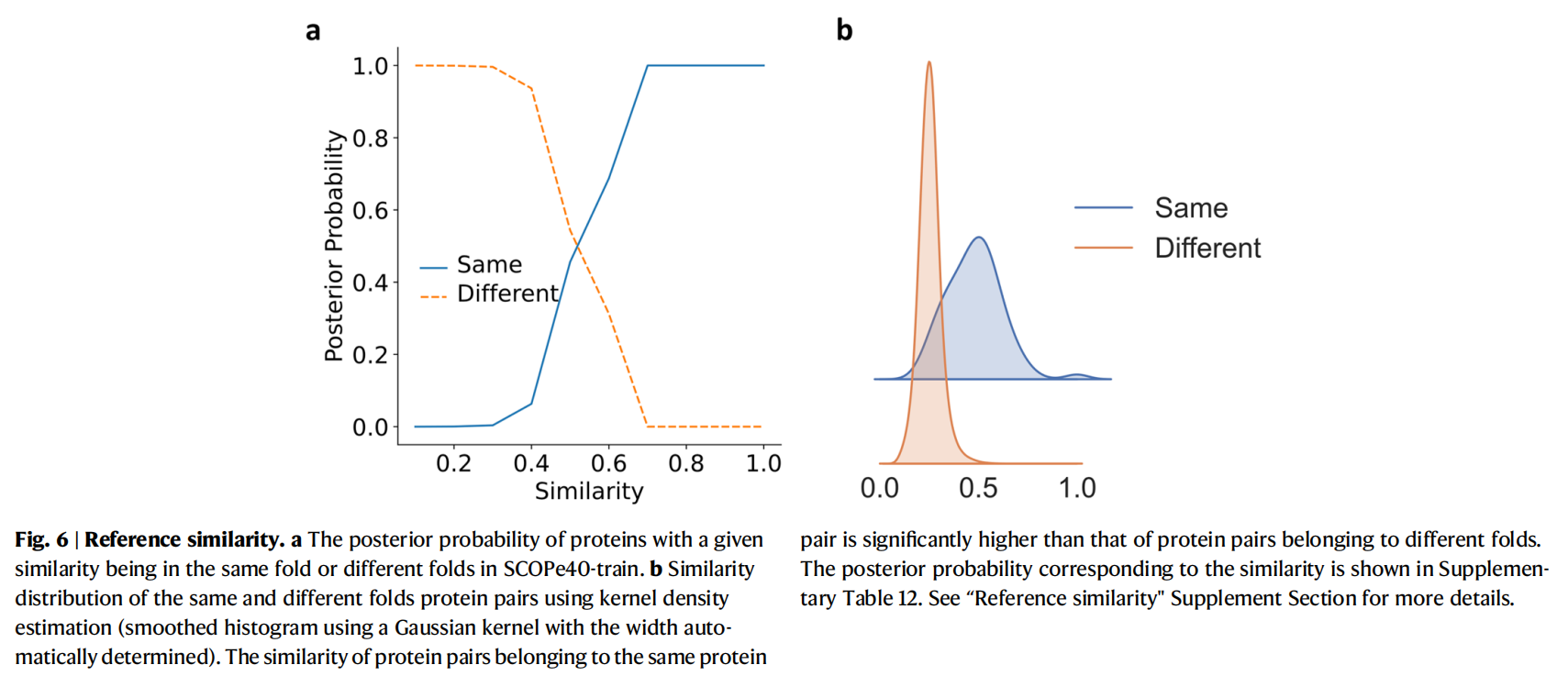
PLMAlign
和PLM-BLAST一样
Datasets
SCOPe40
类似Foldseek,SCOPe40训练/测试集分割为8:2,测试集中没有和训练集一个fold的
New protein search test
引入了一个额外的搜索测试,专门包含无法扫描任何pam域的查询蛋白质,从SCOPe40测试中的2207个查询中总共筛选了110个查询,这些查询没有扫描任何pfam域
Swiss-Prot
开始下载了542,317个蛋白和结构,丢掉pLDDT小于70的得到498,659,丢掉与query蛋白质有40%序列相似度得到430,140
最终是从SCOPe40-test和Swiss-Prot中各选择了50个作为query,测试了43,014,000 query-target pair
CATHS40
丢弃超过300氨基酸长度得到27,270 domains,丢掉和测试集有40%相似度的蛋白得到21,474


