论文地址:Simulating 500 million years of evolution with a language model
ESM3:多模态prompt蛋白质生成模型
Abstract
多模态生成式语言模型涵盖了蛋白质序列,结构和功能。通过prompt ESM3可以实现chain of thought的生成GFP,合成了58% identity的GFP,而实现相似的远端天然GFP需要几百万年的进化
Introduction
ESM3是生成式的掩码语言模型,每个模态在离散的token上进行训练,ALL-to-all的建模离散token使得ESM3可以通过任何模态的组合进行prompt
训练集有2.78B个蛋白质和771B个token,有98B参数,并且发现ESM3对于prompt的响应性很强,能够在复杂的提示组合中找到创造性的解决办法,不同scale的模型可以通过跟随更好的提示实现aligment,并且更大的模型对于alignment有更好的响应
通过生成一种新的GFP进行了验证,命名为esmGFP,与Aequorea victoria GFP有36%的相似度,与最相似的GFP有58%的相似度
ESM3
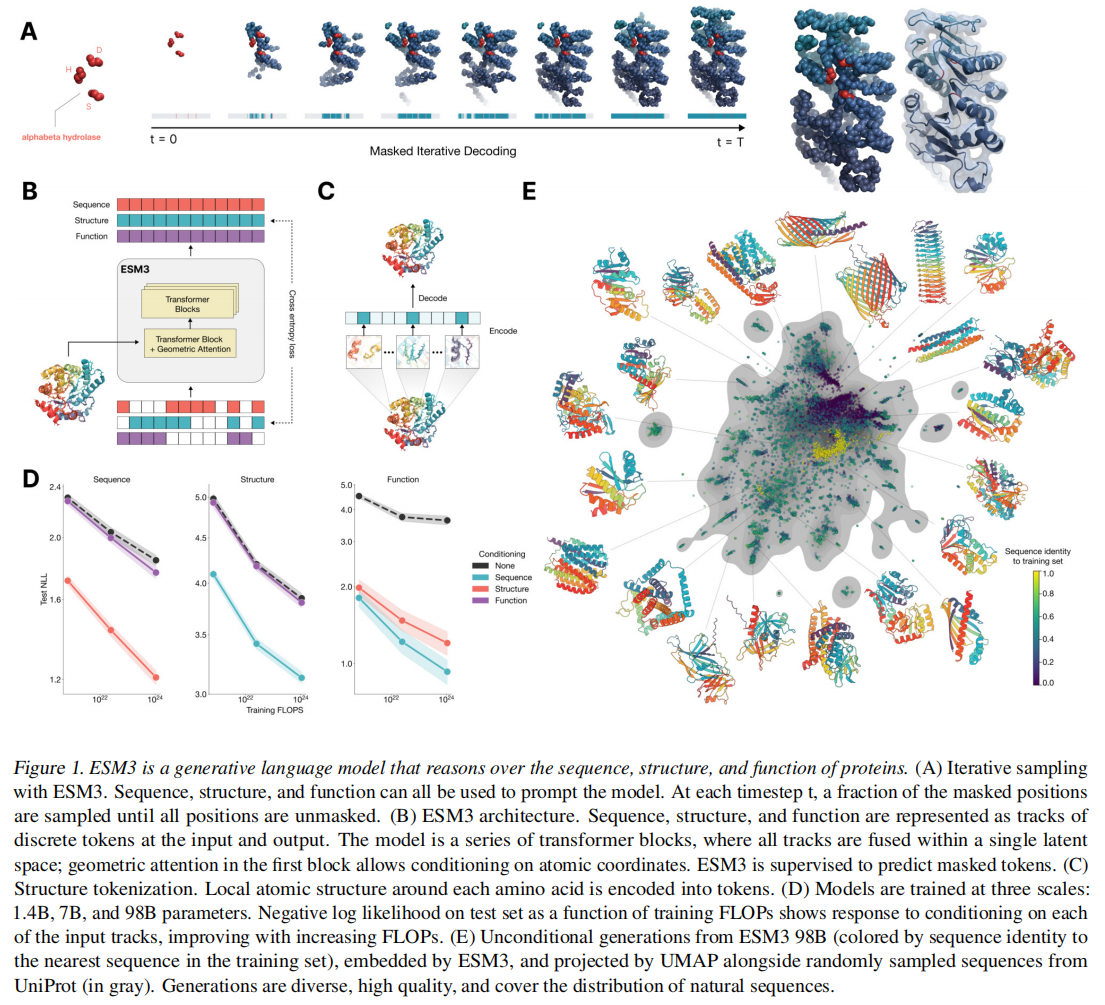
ESM3通过生成式掩码语言模型目标进行训练,而这个mask是从一个noise schedule进行采样的,而不是固定的,这个监督分解了下一个token的所有可能预测的概率分布,确保标记可以从任何起点以任何顺序生成
从所有掩码token的序列开始,token可以一次采样一个,或者以任何顺序并行采样,直到所有token完全解除掩码
mask对于sequence, structure和function track都是独立的,保证任意组合、任意部分或不完整的生成序列
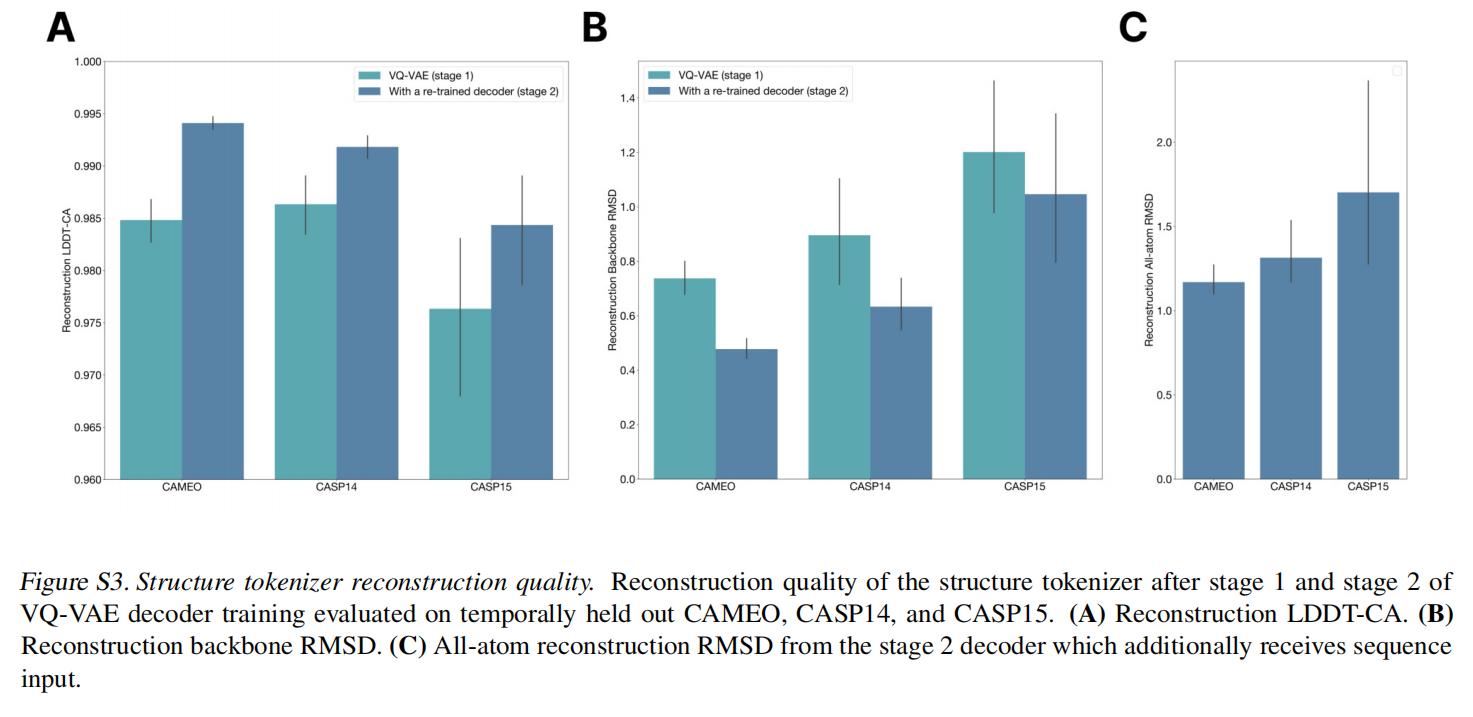
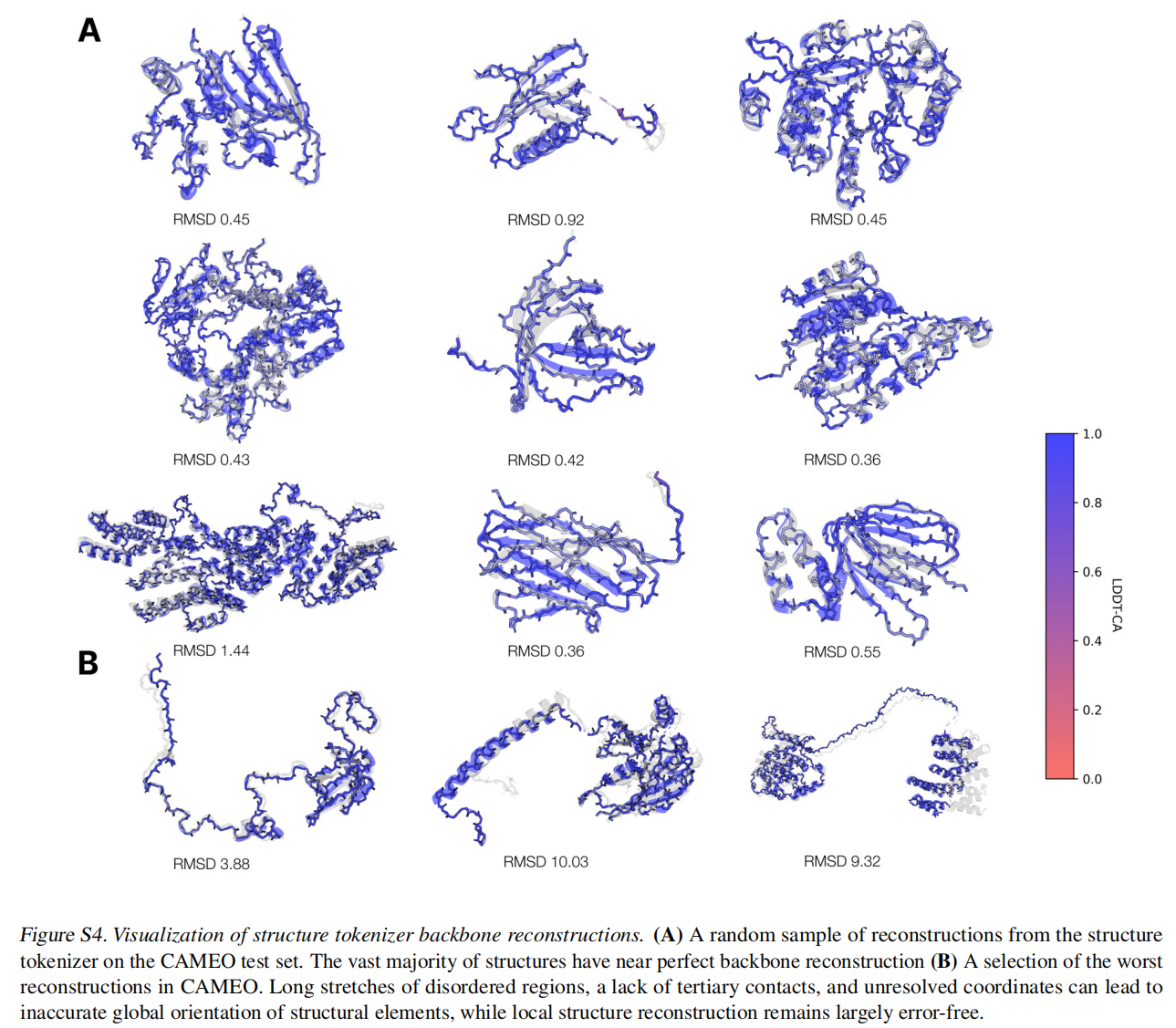
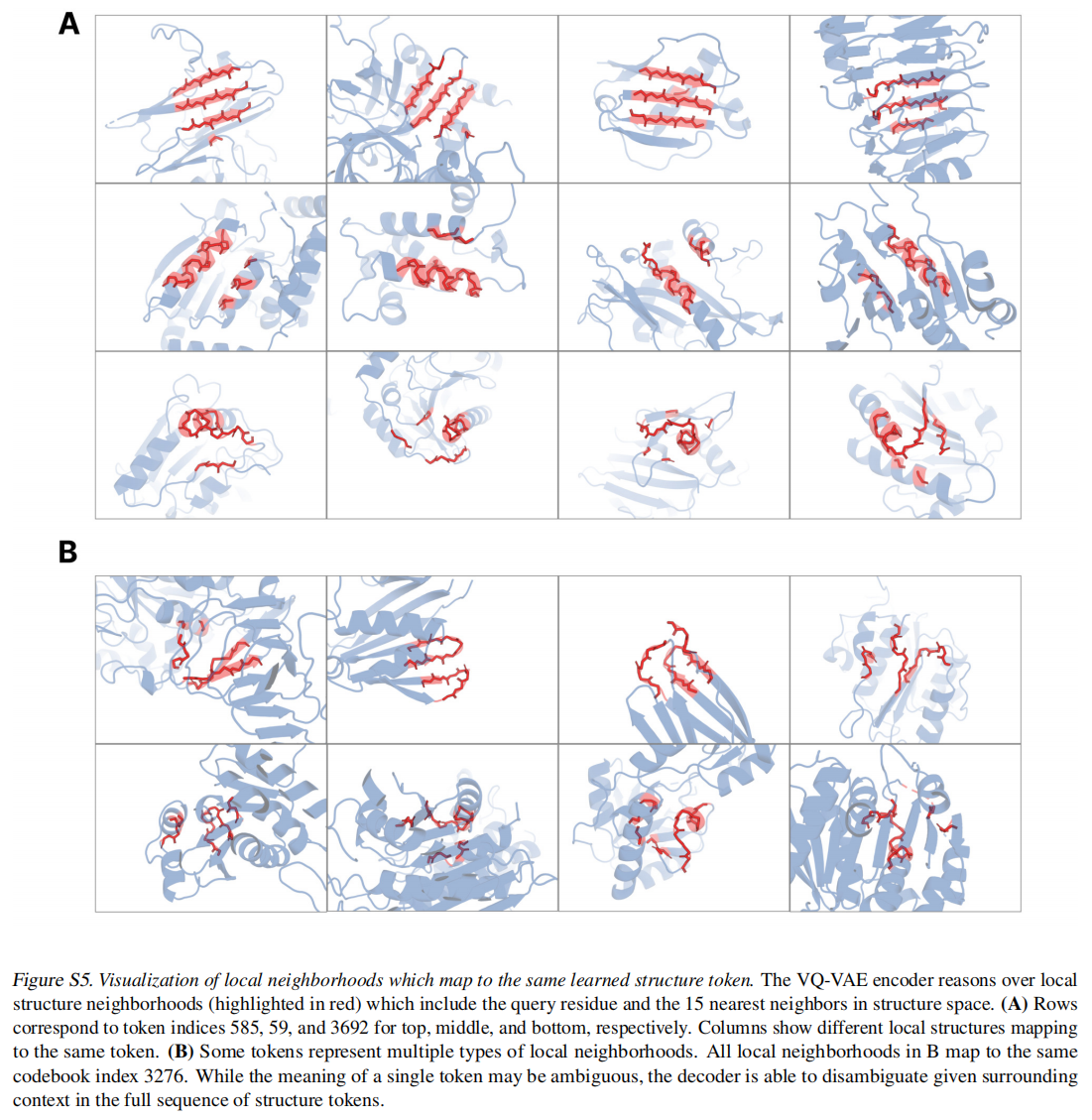
对于结构采用了一个local structure tokenizer将结构嵌入到离散空间中,使用一个不变的几何注意力机制来高效的处理三维结构。ESM3输出的结构token到解码器生成,这种tokenizer已经接近完美重建了蛋白质结构(<0.3A),可以准确表征到原子级别
ESM3可以基于标记化结构和原子坐标中的任何一个或两个条件,还补充了SS8和SASA增强表征,功能以针对序列中的每个位置的tokenized keyword sets表示
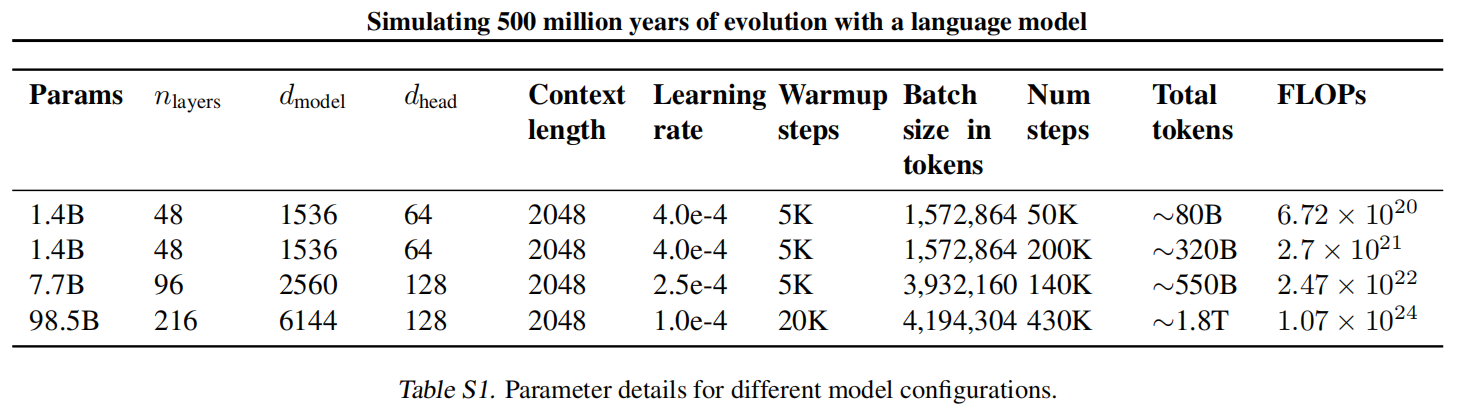
最大的ESM3模型是在来自序列和结构数据库的27.8亿个天然蛋白质上进行训练的,还使用了预测的结构。总的来说,这使训练数据增加到315M个蛋白质序列,236M个蛋白质结构,和539M个具有功能注释的蛋白质,总计771B个独特token。有三个尺度的模型:1.4B,7B和98B,图1D可以看到训练越大,valid loss越小
在单序列结构预测(表S8)中,ESM3 98B得到0.895的平均局部距离差检验(LDDT),超过ESMFold(0.865 LDDT)。无条件生成产生高质量的蛋白质——平均预测LDDT(pLDDT)0.84,预测模板建模评分(pTM)0.52,在序列(平均成对序列标识0.155)和结构(平均成对TM评分0.48)上都存在差异,跨越了已知蛋白质的分布(图
Programmable design with ESM3
ESM3可以通过序列、结构坐标、SS8、SASA和功能关键字进行多种prompt,并为每个track定义了指标:
- constrained site RMSD (cRMSD):生成坐标和提示坐标的RMSD
- SS3 accuracy:生成和提示结构的3 class二级结构比例
- SASA spearman:生成和提示的相关性
- keyword recovery fraction:生成和提示的InterProScan的关键词比例
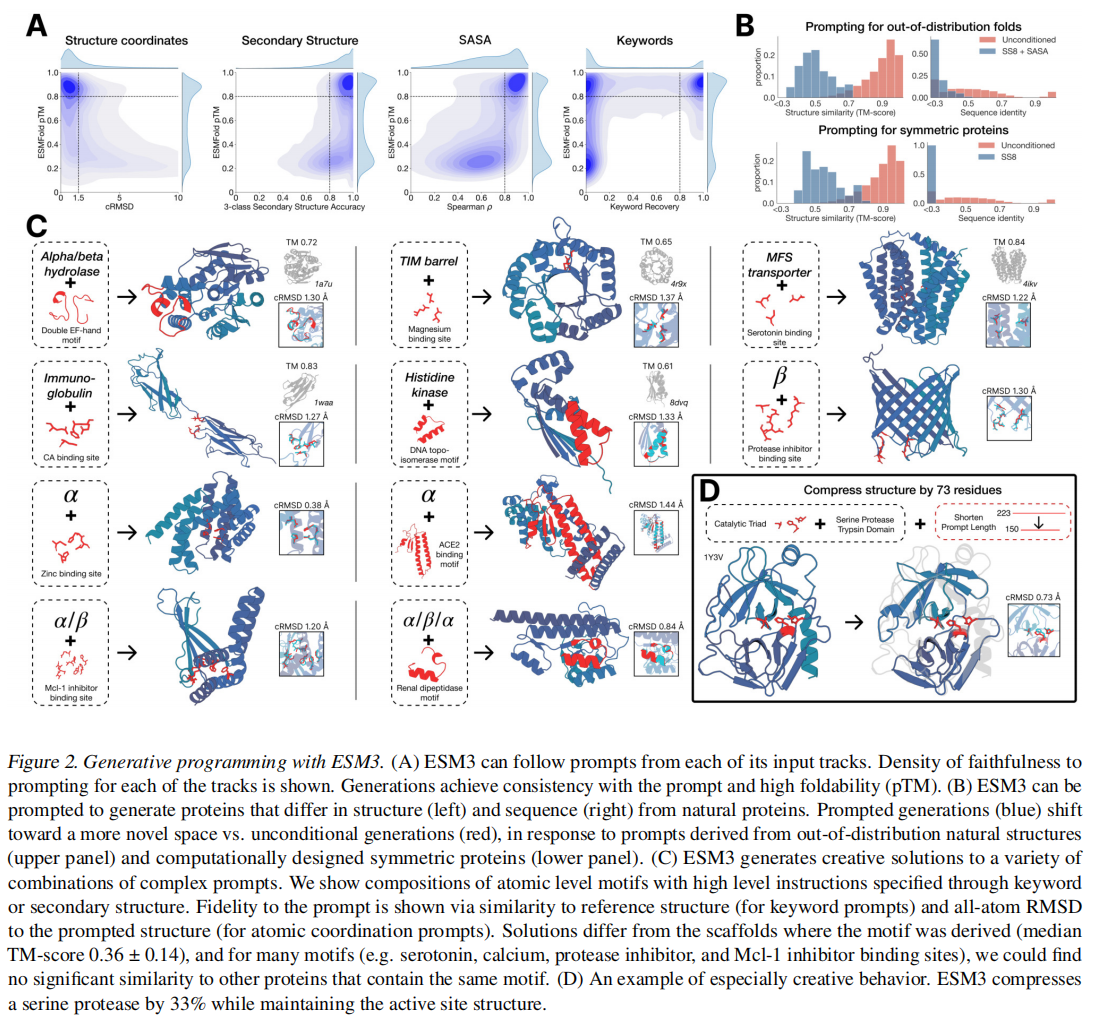
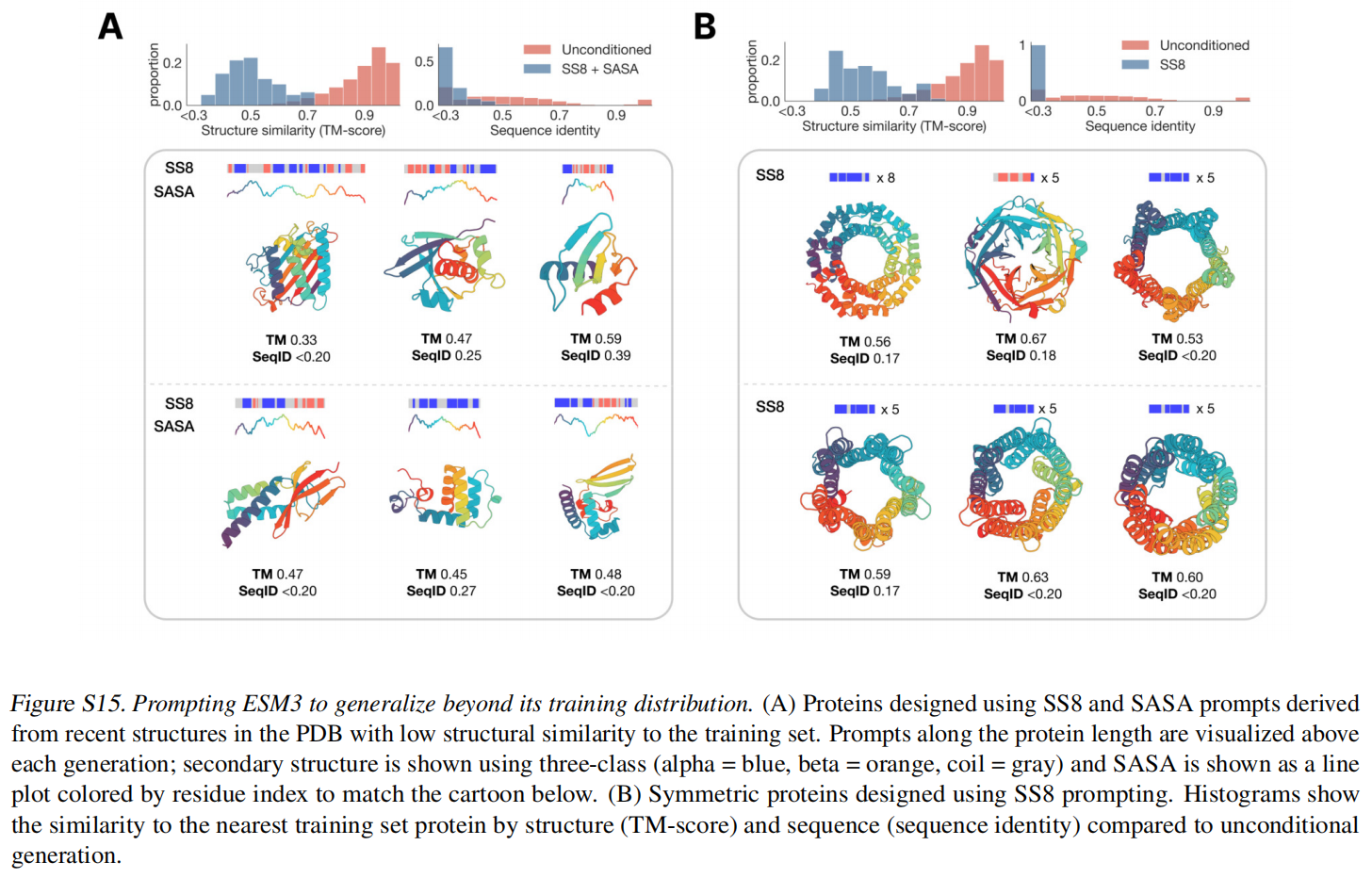
从held out structures (TM < 0.7 to training set)里面进行了一系列的SS8和SASA的prompt组合,尽管生成的序列整体结构依旧合理(mean pTM 0.85),但是和训练集相比还是非常novel的,如图2 B上所示,结构和序列不算相似。还使用了一个人工合成的蛋白质设计数据,用二级结构作为prompt也实现了很好的效果
ESM3还可以遵循复杂的prompt,比如用motif (catalytic centers and ligand binding sites),对于每个独特的motif和scaffold组合,我们生成样本,直到提示满足为止(cRMSD < 1.5˚A为坐标;TM > 0.6为折叠级提示的代表性结构;二级结构提示的SS3精度>80%),具有较高的可信度(pTM > 0.8,pLDDT > 0.8
图2C可以看到,给定prompt的情况下,ESM3生成的蛋白质跟自然界存在的也有一定的相似性,可以看到motife可以嫁接到不同的fold中
图2D展示出ESM3的创造性,从天然胰蛋白酶(PDB 1Y3V)开始,prompt催化三联体的序列和坐标以及描述胰蛋白酶的功能关键字,但将总生成长度减少三分之一(从223到150个残基)。ESM3保持了活性位点(cRMSD 0.73˚A)和整体折叠的协调,具有高可设计性(pTM 0.84,scTM平均0.9d0.97,std0.006),尽管序列长度显著减少,折叠仅由功能关键字提示指定
Biological alignment
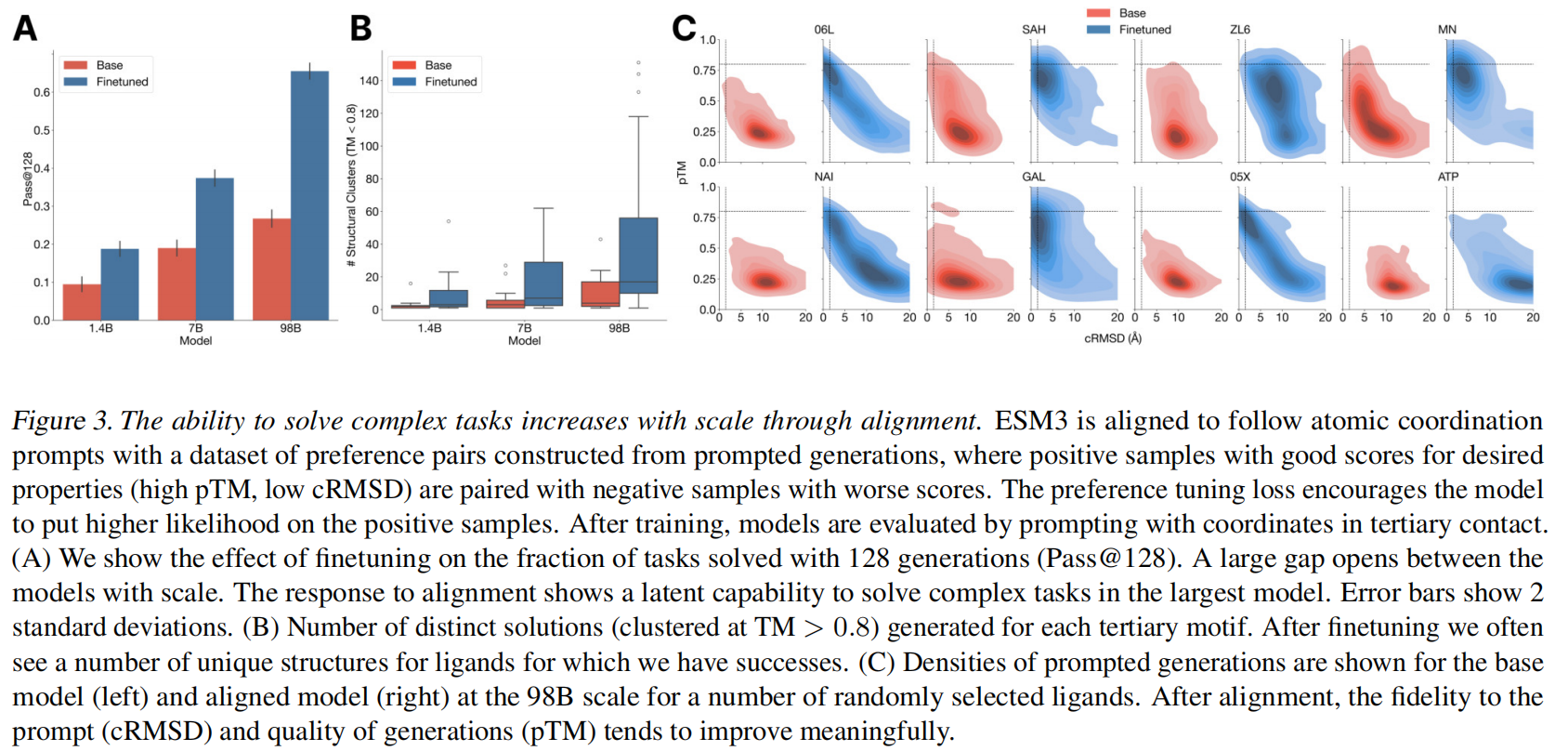
通过微调将模型与任务对齐,能够效果更好
构建一个部分结构提示的数据集,为每个提示生成多个蛋白质序列,然后使用ESM3对每个序列进行折叠和评分,以与提示(cRMSD)和可折叠性(pTM)保持一致性。高质量的样本和低质量的样本成对丢进去进行prompt来构造perference dataset,ESM3来优化在这个perference tuning loss,让模型对高质量的样本生成更高的likelihood,可见Appendix A.4
为了验证使用tertiary motif scaffolding prompts也能生成高质量的骨架,使用了46个从held out数据里来的46个ligand binding motifs,prompt氨基酸身份,原子坐标等,对于每个motif任务,我们通过排列残基的顺序,改变它们在序列中的位置,并改变序列的长度来创建1024个提示,使用128次生成后解决任务的百分比(主干cRMSD < 1.5˚A,pTM>0.8)来评估成功程度
图3A显示经过偏好调整的模型通过率效果提升了两倍,图3B显示更大的模型能够生成更多样的结果(更多的solutions)
Generating a new fluorescent protein
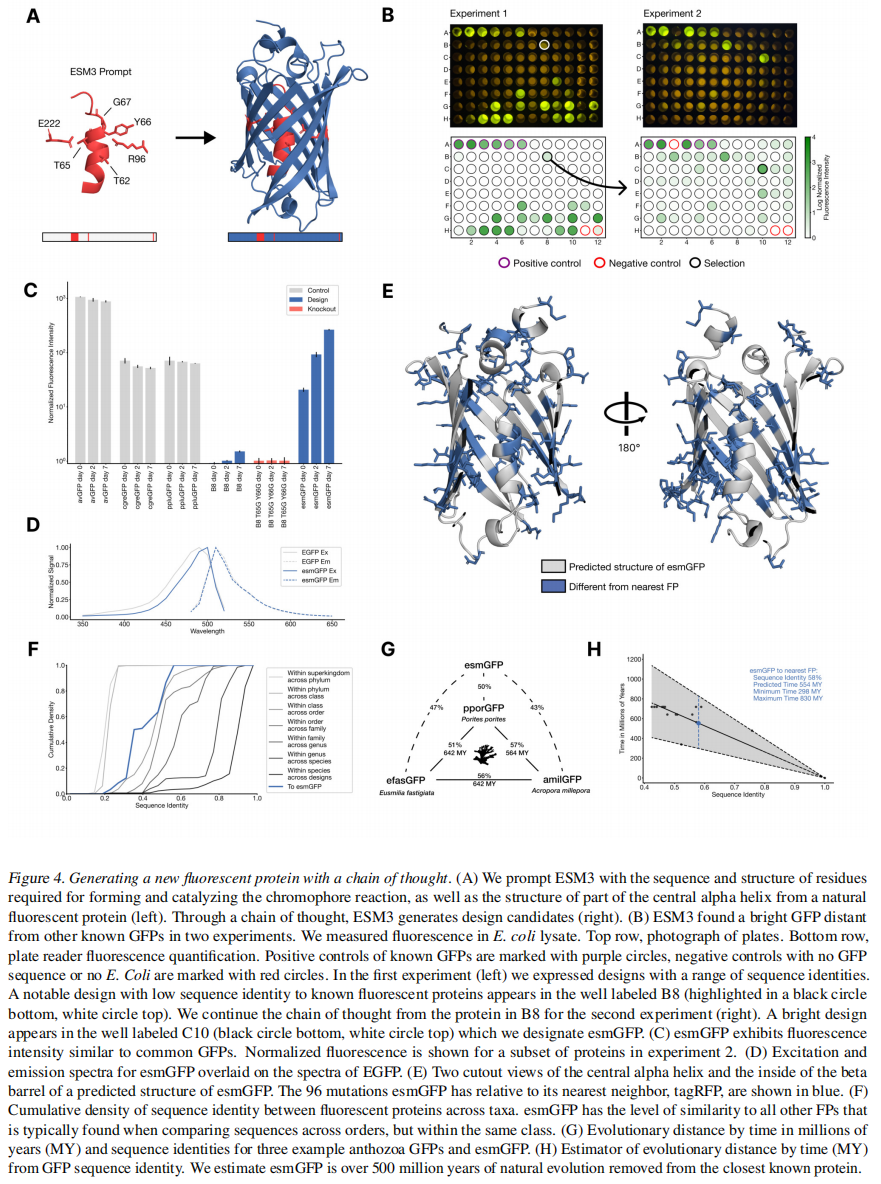
证明base的预训练ESM3已经有足够的生物保真度来生成蛋白质
直接prompt7B参数ESM3生成一个229个残基蛋白,其位置为Thr62、Thr66、Gly67、Arg96、Glu222,这些位置是形成和催化发色团反应的关键残基,还对1QY3实验结构中58到71的结构进行了条件,这些结构对发色团形成的能量有利非常重要,其他位置基本都mask了
使用一个chain of thought的方法来生成GFP,Appendix A5.1
- 先把生成的与1QY3活性中心相似但整体不相似的骨架经过一个filter到下一步2
- 将生成的结构添加到原始提示中,以生成以新提示为条件的序列
- 执行一个迭代的联合优化,交替优化序列和结构
- 并拒绝在chain of thoughts中丢失了活性点位坐标的样例
- 在生成协议的迭代联合优化阶段,从中间点和最终点绘制了10个数千个候选GFP设计的计算池
- 最后通过与已知荧光蛋白的序列相似性进行设计,并使用各种指标进行过滤和排序设计
在96个孔板中对88个设计进行了实验,在B8井中发现了一个设计(用黑色圆圈突出显示),与1QY3序列的序列同源性仅为36%,与最近的现有荧光蛋白tagRFP的序列同源性为57%,但这个设计的比天然的暗50倍
从B8井的设计序列开始,继续思维链,使用上述相同的迭代联合优化和排序程序,生成具有提高亮度的蛋白质。我们创建了第二个96孔板的设计,并使用相同的板阅读器分析,我们发现在这个队列中的几个设计的亮度在自然界中发现的gfp范围内。最好的设计,位于第二个板的C10井(图4B右),我们指定为esmGFP。
Discussion
蛋白质语言模型并没有在进化的物理约束下明确地工作,而是可以隐式地构建一个进化可能遵循的多种潜在路径的模型
Appendix
Architecture
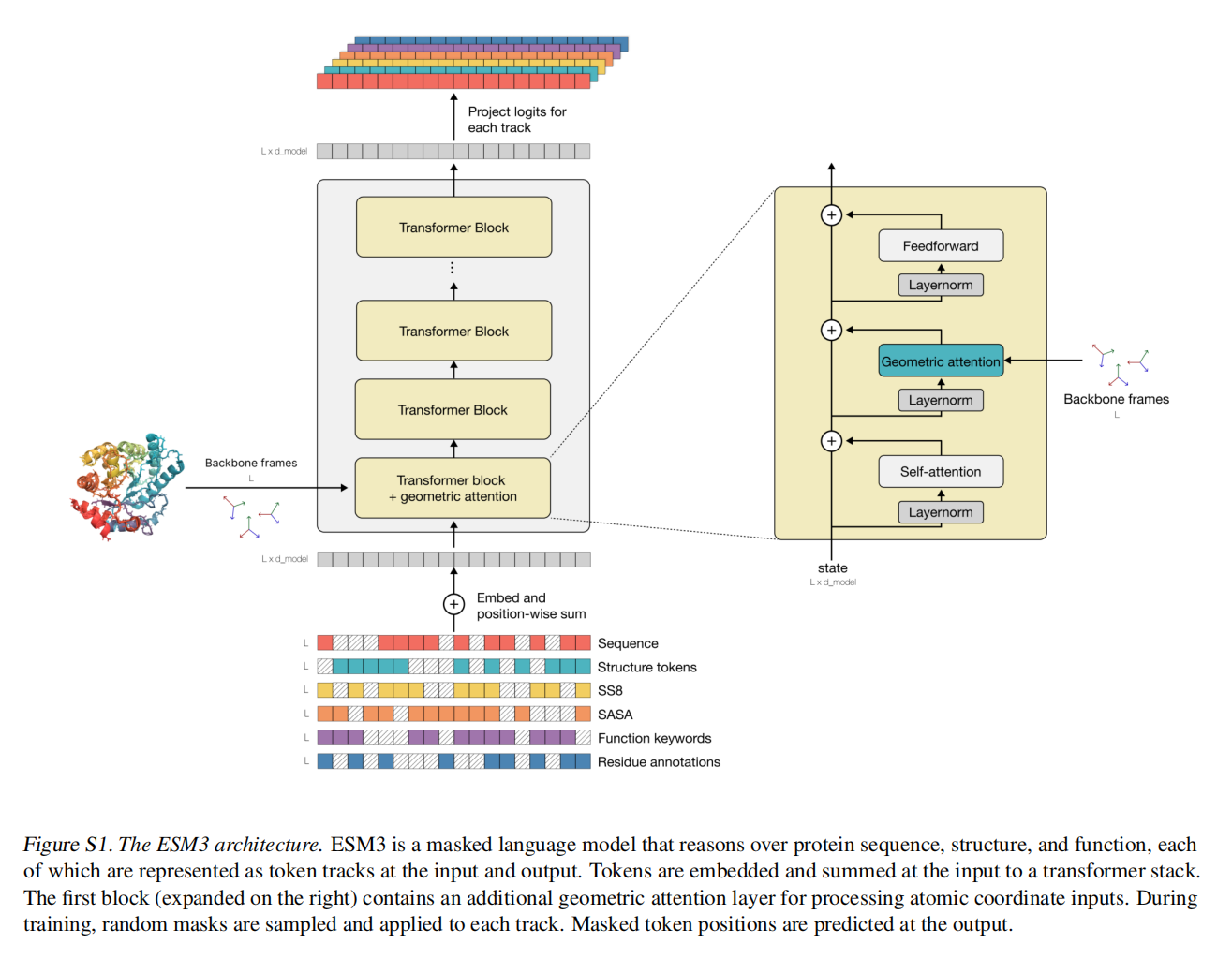
frm_input
(a) sequence, (b) structure tokens, © SS8, (d) quantized SASA, (e) function keyword tokens and (f) residue (InterPro) annotation binary features.
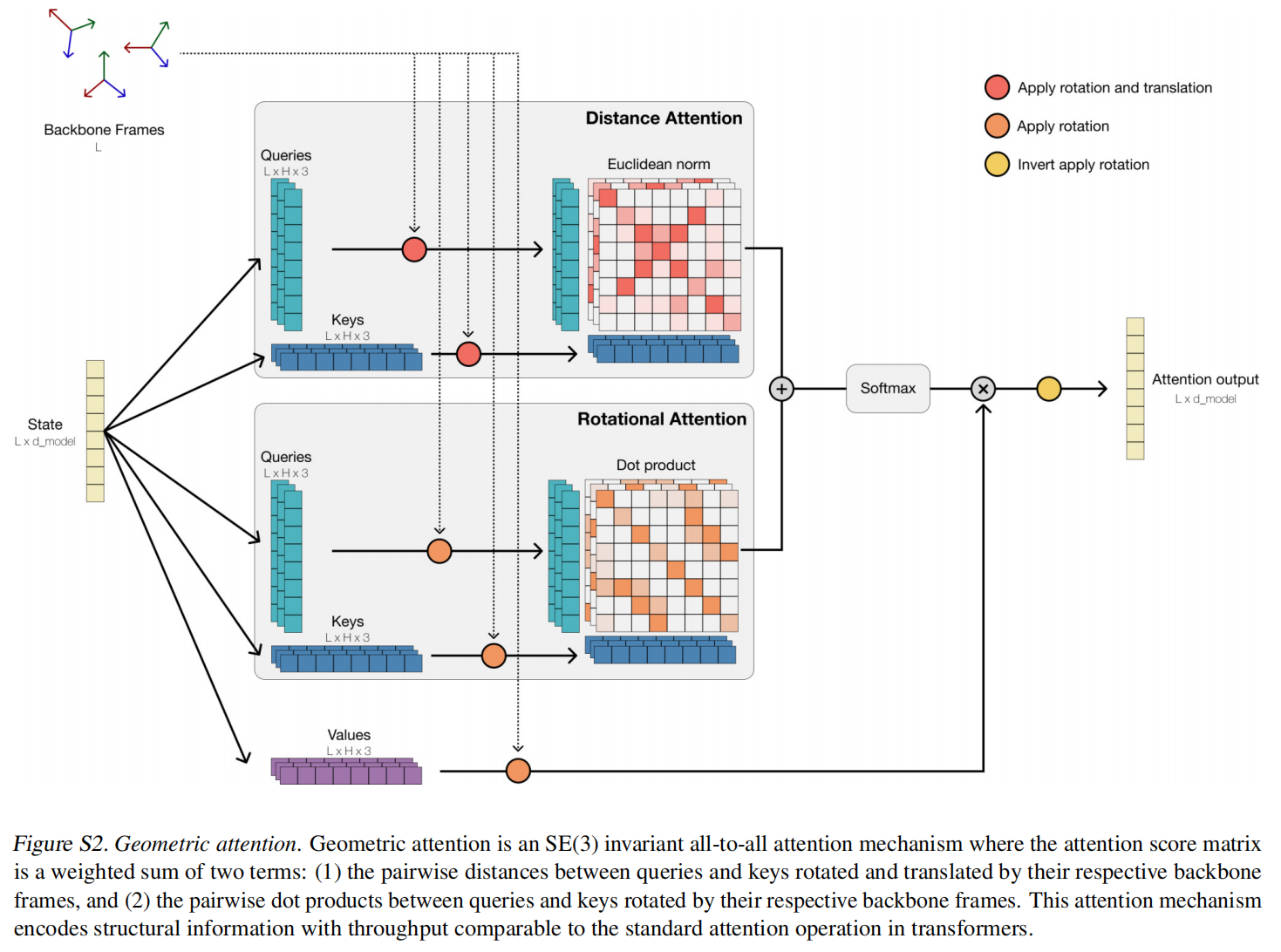
Geometric Attention
Structure Tokenizer
Generation
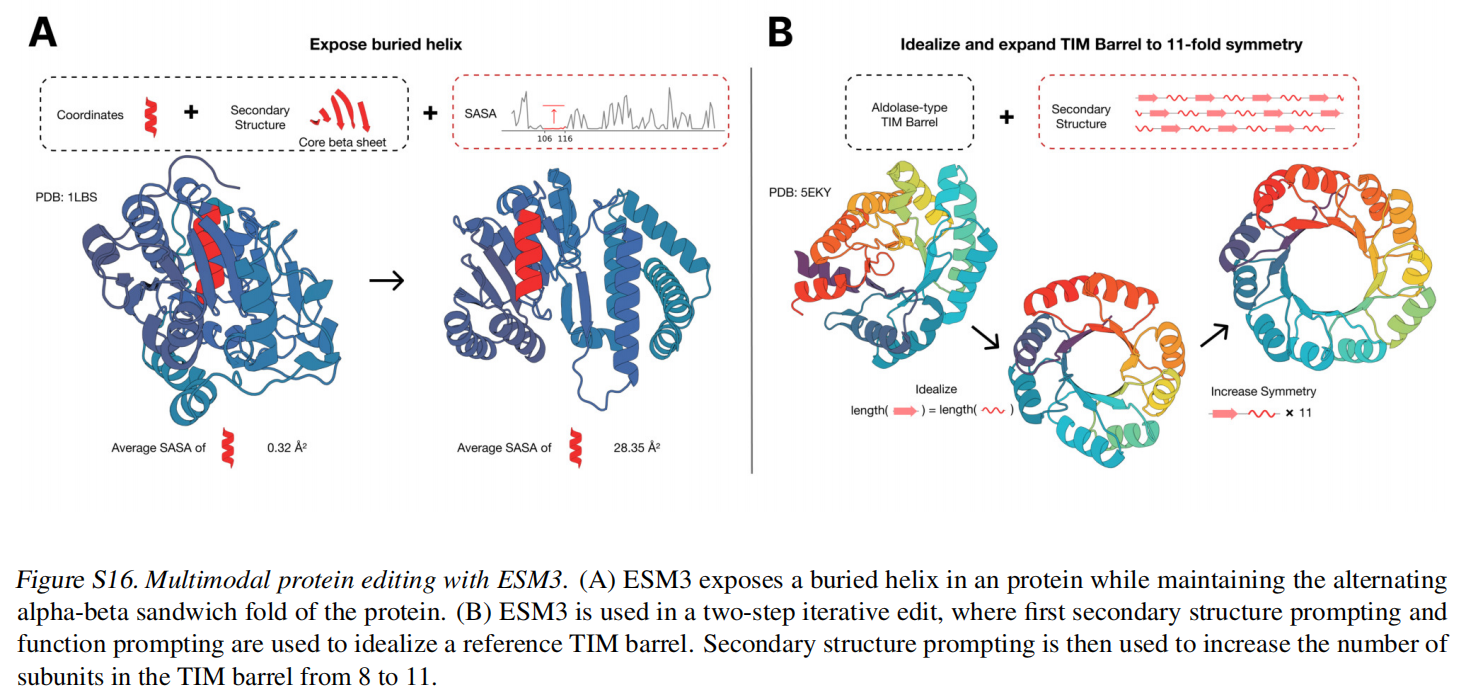
Multimodal protein editing with ESM3