IgDesign:微调ProteinMPNN+LM-Design做CDR设计
Abstract
目前的设计抗体CDR的方法还没有经过体外实验验证,这里作者提出了IgDesign的深度学习方法,并且设计8种治疗性抗原成功结合的binder证明了其模型的稳健性。模型负责设计重链CDR3(HCDR3)或所有三个重链CDRs(HCDR123)。对于这8种抗原,每一种设计了100个HCDr3和100个HCDR123,将它们支架到天然抗体的可变区域,并使用表面等离子体共振(SPR)筛选它们与抗原的结合。作为基线,我们从模型的训练集中筛选了100个HCDR3,并与原生的HCDR1和HCDR2配对。IgDesign是第一个经过实验验证的抗体逆折叠模型。它可以设计多种治疗性抗原的抗体结合物,具有高成功率,在某些情况下,改善临床验证的参考抗体。同时本研究中产生的数据可以作为不同抗体-抗原相互作用的benchmark
Introduction
IgDesign是基于LM-Design的抗体生成式方法,能够同时设计一个参考抗体2的HCDR3和所有三种HCDRs(HCDR123),并保持结合力
基线方法可以看作是一个简单的随机算法(直接从训练数据中抽样,而不考虑目标结构),比较结果证明 IgDesign 比“随机方法”在大多数任务中更强
Methods
为了去了解ProteinMPNN能不能用于抗体设计,先尝试了IgMPNN,是在抗体数据集上进行训练的。IgMPNN 是 IgDesign 中专门用于抗体设计的核心模块,基于 ProteinMPNN 和 AbMPNN 进行了优化。与 AbMPNN 的主要区别在于:1)在训练过程中,IgMPNN 额外提供了抗原序列和抗体框架(FWR)序列作为上下文,使模型能够更好地生成与抗原匹配的抗体序列;2)模型在训练时按顺序解码 CDR 区域(HCDR1 → HCDR2 → HCDR3 → LCDR1 → LCDR2 → LCDR3),但在推断时可以灵活指定生成顺序。同时,在每个 CDR 区域内的氨基酸生成顺序是随机的,以增加生成多样性。这些改进使 IgMPNN 更适用于抗体设计任务
IgDesign中的CDR设计协议是基于LM-Design中提出的结构编码器和序列解码器相结合的组合方法,IgMPNN 提取最终节点嵌入和模型的 logits,通过采样最大似然估计得到一个标记化的序列。该序列被输入到 ESM2-3B 蛋白语言模型中,并提取其最终投影头前的嵌入。接着,使用瓶颈适配器层(BottleNeck Adapter)进行交叉注意力,其中 IgMPNN 的节点嵌入作为 keys,ESM2-3B 的嵌入作为 queries 和 values。生成的新嵌入通过 ESM2-3B 的投影头,得到最终 logits,并与 IgMPNN 的 logits 相加以生成模型的最终输出。
IgMPNN 首先在来自 PDB(分裂为 40% 序列相似性)的通用蛋白质数据集上进行预训练,随后在 SAbDab 的抗体-抗原复合体数据集上进行微调(fine-tuning)。IgDesign 则使用经过预训练和微调的 IgMPNN 作为结构编码器,并在相同的抗体-抗原复合体数据上进一步微调。在数据划分中,采用 40% 序列相似性的抗原排除策略(antigen holdout)来避免数据泄漏,同时从通用 PDB 数据集中移除了所有 SAbDab 的结构以确保独立性。对于每个抗原,都单独训练一组 IgMPNN 和 IgDesign 模型,并确保参考抗体的 HCDR3 序列未出现在训练集中。所有模型均使用 Adam 优化器,学习率设为 10⁻³ 进行训练。
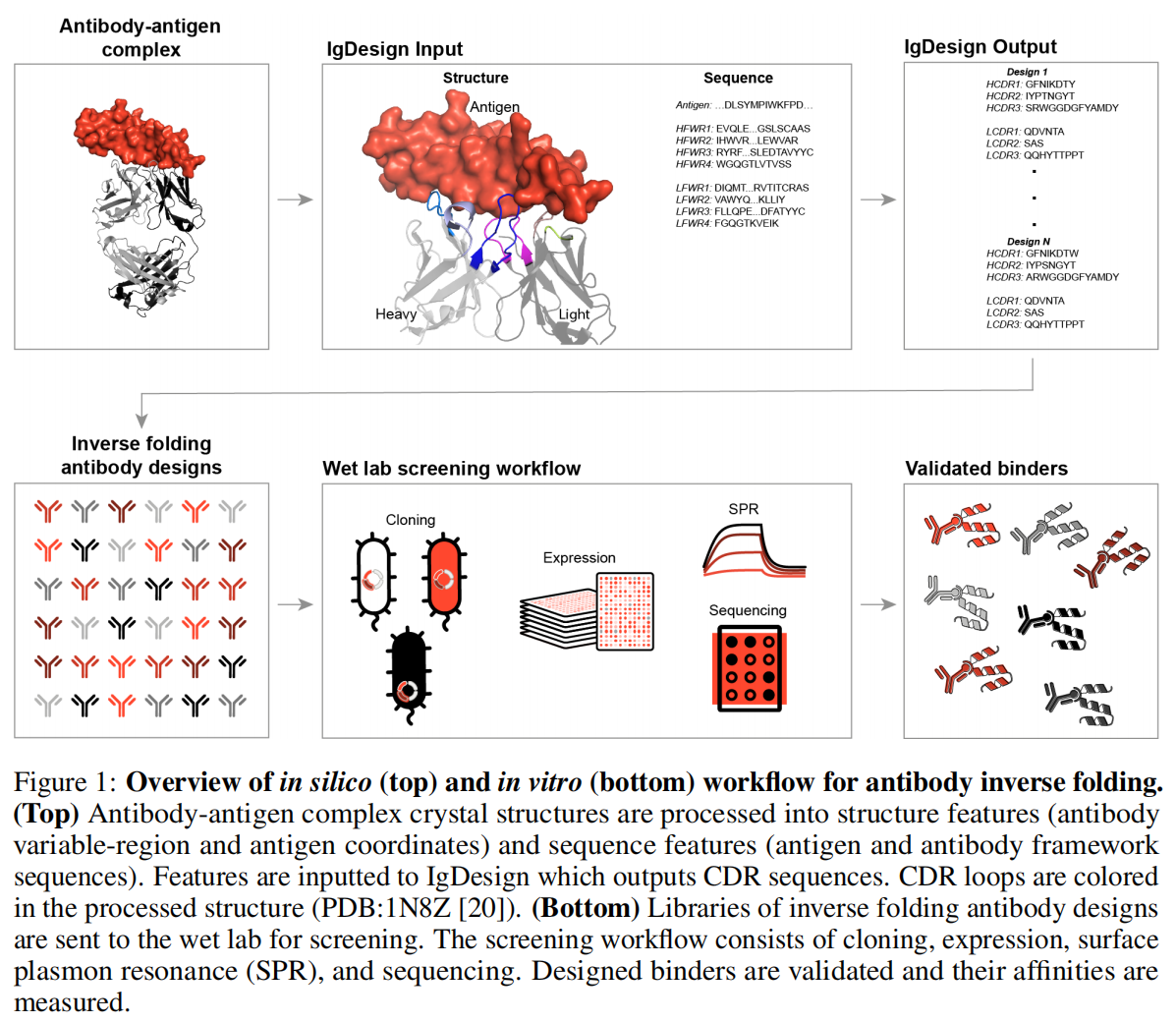
从精心筛选的 SAbDab 数据集中选取了 8 个具有治疗价值的抗原,每个抗原都配有一个参考抗体的结合物(binder)及其抗体-抗原复合体结构。IgDesign 使用抗体-抗原复合体的 3D 骨架坐标、抗原序列和抗体框架(FWR)序列作为输入。在推断时,按以下顺序生成抗体的序列:HCDR3 → HCDR1 → HCDR2 → LCDR1 → LCDR2 → LCDR3。对于每个抗原,IgDesign 生成了 100 万个候选序列,并筛选出交叉熵损失最低的 100 个 HCDR3 序列和 100 个 HCDR123 序列用于体外评估。作为对比基线,研究者从 SAbDab 数据集中为每个抗原随机抽取了 100 个 HCDR3 序列,这些基线序列与模型的训练分布一致,并且与原生 HCDR1 和 HCDR2 配对。最终,研究为每个目标抗原设计了一个抗体库,包括 100 个 IgDesign 的 HCDR3 序列、100 个 IgDesign 的 HCDR123 序列,以及 100 个 SAbDab 的 HCDR3 基线序列。此外,还设置了对照实验以验证 SPR 测试准确区分已知的结合物和非结合物的能力。更详细的信息在附录中说明
Results
Amino acid recovery (AAR)
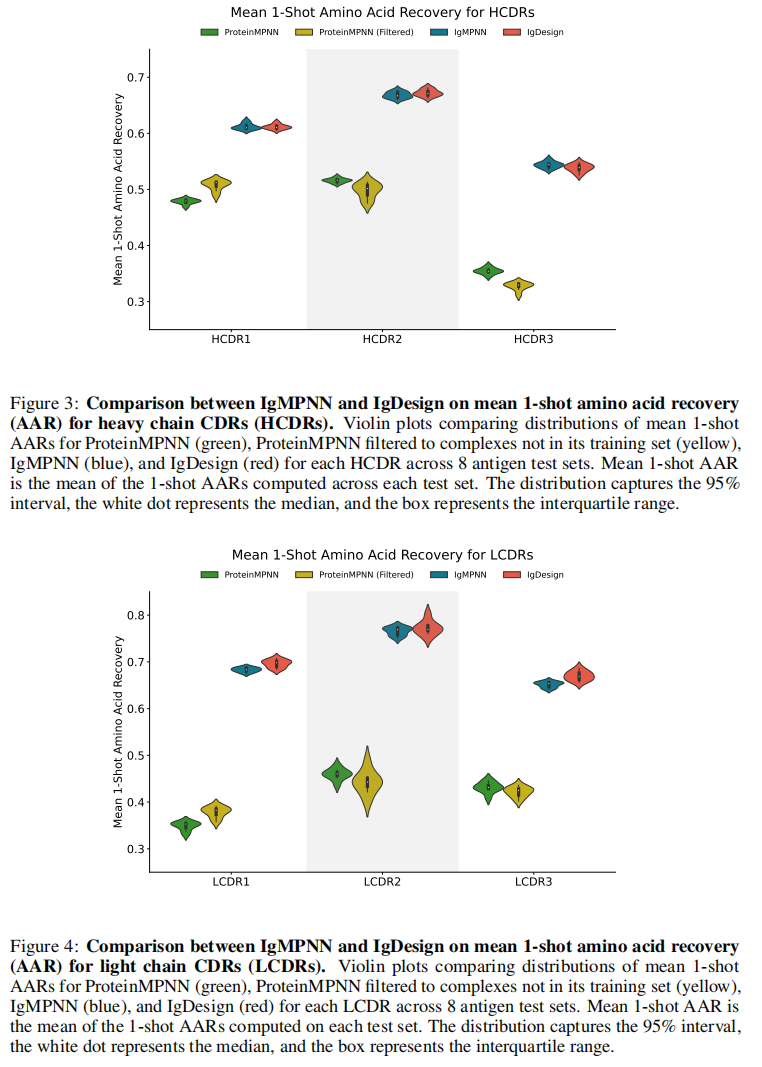
使用 IgMPNN、IgDesign 和 ProteinMPNN(作为基线)来评估 AAR,并对 ProteinMPNN 在训练集中未见过的抗体进行额外评估(称为 “ProteinMPNN (Filtered)”)。对于每个抗原,研究者训练了一个排除该抗原的数据模型,并计算其测试集 AAR,特别关注单样本的 AAR(1-shot AAR 是指在每个模型生成的序列中,随机选择一个样本序列来计算 氨基酸恢复率(Amino Acid Recovery, AAR) 的值)。通过比较每个模型在测试集上的平均 AAR(覆盖 8 种抗原),发现 IgMPNN 和 IgDesign 在所有情况下均显著优于 ProteinMPNN 和其 Filtered 版本(Mann-Whitney U 检验,p < 2e-4)

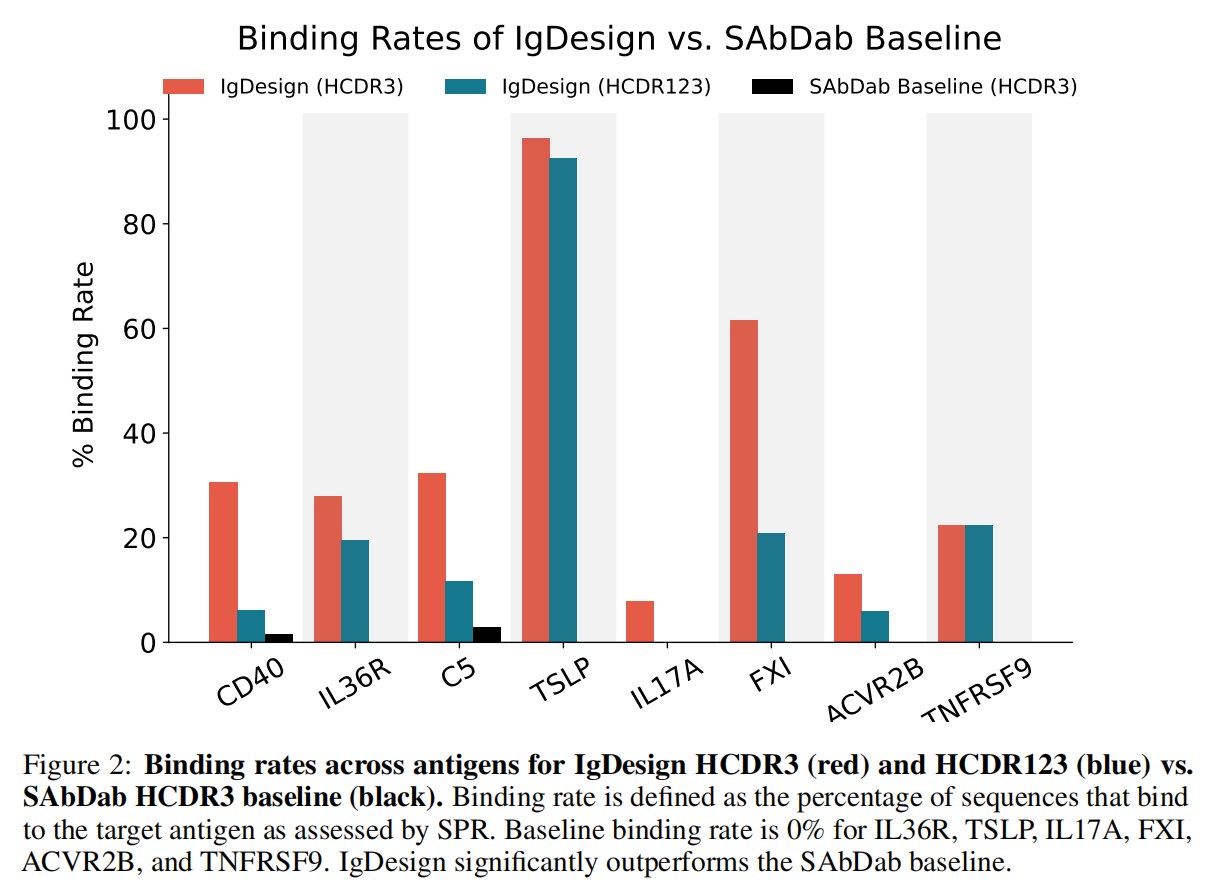
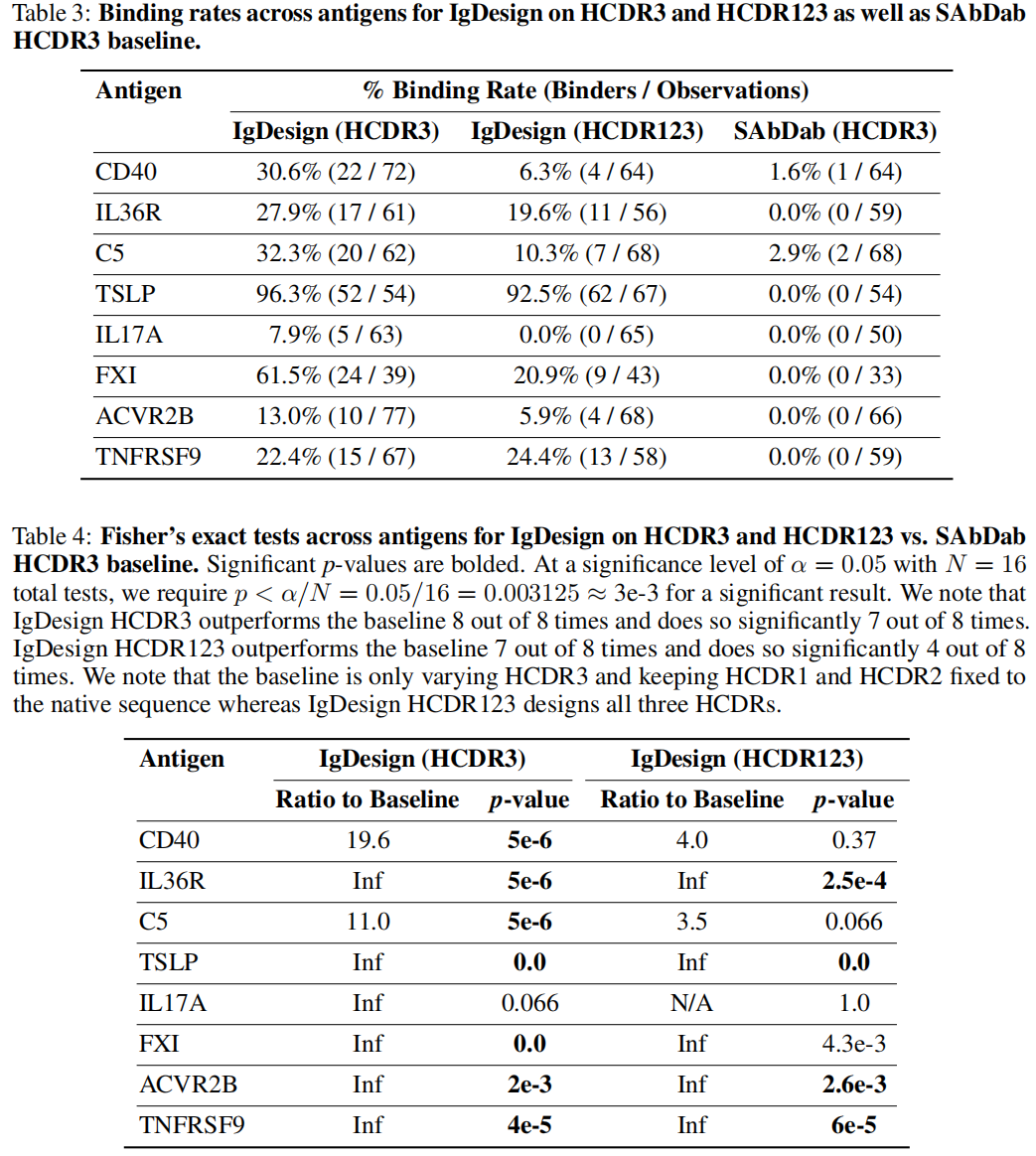
In vitro binding rates and affinities
结合率:
- IgDesign 生成的 HCDR3 序列 在 8 个抗原中的 7 个上表现出显著更高的结合率,与 SAbDab 基线的 HCDR3 相比具有统计学显著性(Fisher 检验,p < 3e-3)。
- IgDesign 生成的 HCDR123 序列 在 8 个抗原中的 4 个上结合率显著高于基线(p < 3e-3)。
- 基线 HCDR3 仅对 2 个抗原显示了结合(CD40 和 C5)。
Self-consistency RMSD (scRMSD)
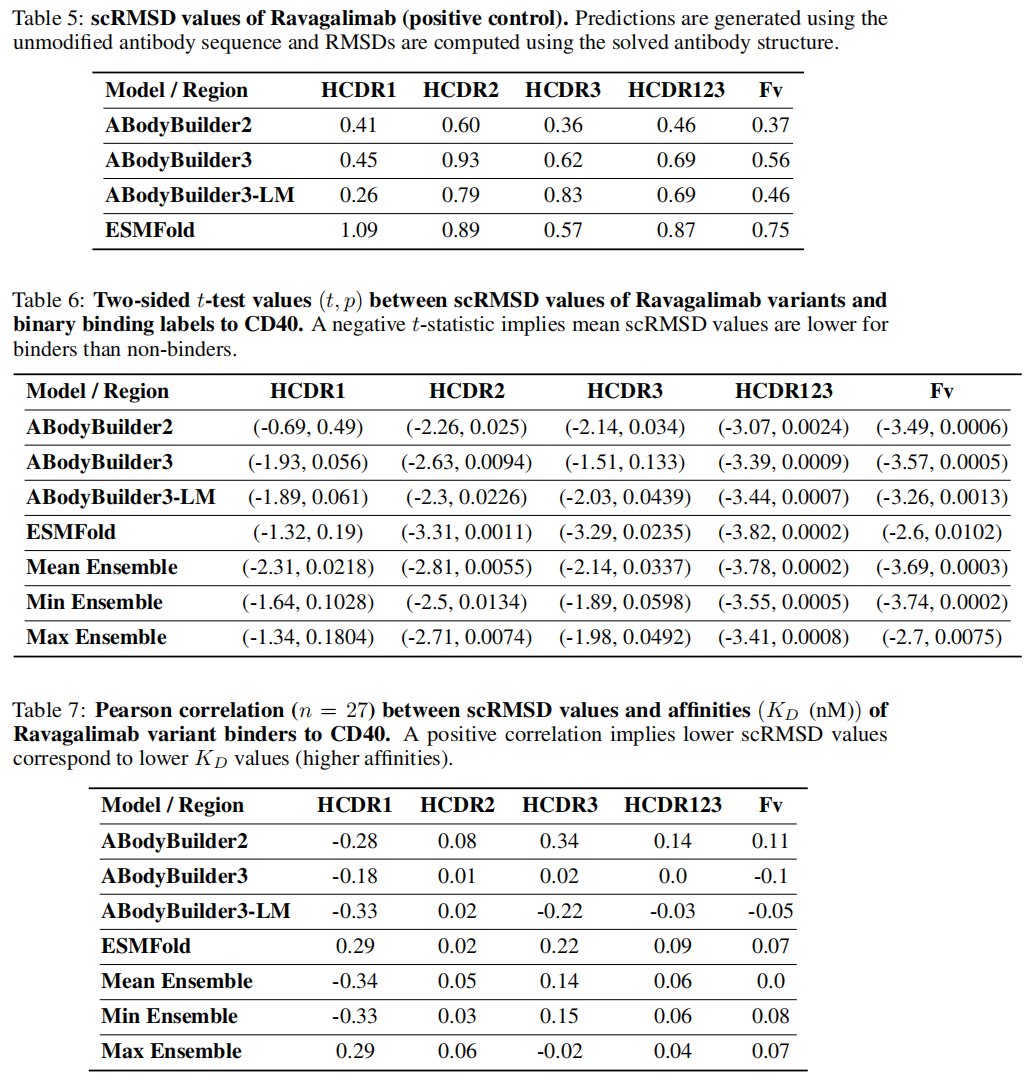
使用 SPR 数据集中关于抗体结合物(binders)和非结合物(non-binders)的亲和力数据,评估了常用的 Self-consistency RMSD(scRMSD)指标在区分结合物和非结合物以及预测亲和力方面的能力。scRMSD 计算覆盖了 HCDR1、HCDR2、HCDR3、HCDR123,以及 Fv 区域(抗体的可变片段)。研究采用 ABodyBuilder2、ABodyBuilder3 和 ESMFold 进行结构预测和 scRMSD 计算。完整的结果,包括 scRMSD 在不同抗原、设计类别和结合标签上的分布可视化以及统计数据,均在附录中展示
研究发现,scRMSD(Self-consistency RMSD) 在区分结合物(binders)和非结合物(non-binders)方面具有一定作用,但效果受数据偏差和其他限制因素的影响:
-
结合物 vs. 非结合物的 scRMSD 值:
-
总体上,结合物的 scRMSD 平均值低于非结合物,有些情况具有统计学显著性(见表 6、9、12 等)。
-
然而,数据集中可能存在以下偏差:
- 数据泄露(data leakage):结构预测模型可能在训练中见过参考抗体序列,导致 scRMSD 偏向参考抗体(见表 5、8、11 等)。
- 基线 HCDR3 的影响:SAbDab 数据集中 HCDR3 的 scRMSD 分布通常高于逆折叠设计的 HCDR3,原因可能是基线 HCDR3 没有基于结构条件化,且在结构预测模型中可能出现过。
-
-
逆折叠设计的 scRMSD 分布:
- 对于仅使用逆折叠设计的抗体,scRMSD 分布更紧致(特别是在 HCDR3 区域)。
- 但在这种情况下,scRMSD 在结合物和非结合物之间的区分能力较低。
-
亲和力(affinity)和 scRMSD 的相关性:
- 通过计算 Pearson 相关系数发现,scRMSD 和亲和力(KD 值)之间的相关性较弱,且方向(正相关或负相关)不一致。
- 唯一始终表现正相关的指标是 Mean Ensemble Fv scRMSD,但其相关性通常很低(如 Ravagalimab 上仅为 0.001)。
- 在 Afasevikumab 数据集(仅 6 个结合物)中,该指标的最大相关性为 0.87,但样本量过小。
Discussion
IgDesign 是一种抗体逆折叠模型,通过整合蛋白质逆折叠模型(如 ProteinMPNN 和 LM-Design)、语言模型(如 ESM2)的思想,以及抗体特定的上下文信息(抗原序列和抗体 FWR 序列)与抗体-抗原复合体的微调训练而开发。研究表明,IgDesign 能够稳定设计出针对多种治疗性抗原的结合物(binders),并通过 SPR 实验验证了其结合能力。虽然 scRMSD 在区分结合物和非结合物以及预测亲和力方面的作用有限,但研究强调需要更明确的评估方法和新指标以改进抗体设计任务。IgDesign 是首个在体外验证抗体逆折叠方法的模型,展示了其在抗体开发中的广泛应用潜力,包括优化抗体变体预测和支持从头设计。研究还公开了相关数据集,以促进社区的基准测试和模型改进。


