RefineGNN:迭代生成序列和结构做CDR设计
Abstract
这篇论文提出了一种新的生成模型,旨在自动设计抗体的互补决定区(CDRs),以增强其结合特异性或中和能力。传统的生成方法通常将蛋白质设计视为一个以已知三维结构为条件的序列生成任务。然而,这种方法在抗体设计中并不适用,因为所需的3D结构通常事先未知。为了解决这一问题,作者将CDR的序列和结构同时作为图进行联合设计。他们提出了一种迭代细化的图神经网络模型,该模型在自回归地生成序列的同时,迭代地细化其预测的全局结构。预测得到的结构又反过来指导后续残基的选择。为了提高计算效率,模型在处理CDR与框架区域之间的条件依赖性时,采用了一种粗粒度的表示方法。实验结果显示,该方法在测试集上取得了更高的对数似然值,并且在设计能够中和SARS-CoV-2病毒的抗体方面,表现优于之前的基线方法
Introduction
抗体通过其互补决定区(CDRs)与病原体结合,决定了其特异性和中和能力,是治疗疾病(如SARS-CoV-2)的重要工具。计算设计抗体的目标是生成具有特定功能(如高结合亲和力、稳定性)的CDR序列,但这在巨大的组合搜索空间中极具挑战性。
现有的基于序列的生成方法忽视了3D结构的影响,可能导致亚优化性能,而基于已知3D结构的生成方法要求预先定义目标结构,但对于抗体设计,这通常是不现实的
研究的核心问题:
- 如何联合建模序列与其对应的3D结构。
- 如何有效建模CDR序列与其上下文(框架区域)之间的条件关系,包括序列和结构交互。
- 如何优化抗体设计以满足多种属性(如结合亲和力、稳定性等),传统的物理计算方法关注于结构能量最小化,但实际上情况更复杂
本文提出的解决方案:
- 将抗体设计问题表述为一个图生成问题,其中图同时编码了序列信息和3D结构信息。
- 提出了一种新的迭代细化图神经网络(RefineGNN),能够在生成过程中动态修改预测的3D结构,以适应新增节点的变化。
- 引入粗粒度的图表示方法,通过将上下文序列划分为节点块,显著提升了模型的计算效率。
开发一种能够联合设计CDR序列和3D结构的生成模型,能够优化抗体设计以实现特定属性(如SARS-CoV-2的中和能力),这种迭代细化模型还与分子构象预测的评分匹配方法和基于点云的扩散方法有关
Antibody Sequence and Structure Co-Design

Overview
抗体通过与抗原(例如病毒)结合,呈递给免疫系统并激发免疫反应,中和抗体不仅能与抗原结合,还能抑制其活性
抗体由重链和轻链组成,每条链包含一个可变区(VH/VL)和若干恒定区。可变区进一步分为框架区和三个互补决定区(CDRs),CDR是抗体中最具多样性和功能性的部分,决定了抗体的结合和中和能力
抗体设计的建模:
- 抗体设计被建模为一个CDR生成任务,生成CDR的序列,同时基于其框架区的上下文信息。
- 作者使用图的表示方法来同时编码抗体的序列和3D结构信息
RefineGNN模型的核心思想:
- 提出了一种新的图生成方法——RefineGNN,用于联合生成CDR序列和3D结构。
- 模型通过迭代细化全局抗体图的方式生成序列,同时动态调整预测的结构。
- 在生成过程中,CDR的序列和3D结构共同演化,使生成过程更加精确
为简化任务,模型的研究重点是重链的CDR区域(特别是CDR-H3)的生成。但提出的方法可以扩展至轻链的CDR设计
Notations
氨基酸序列表示为s,还有<mask>表示未知氨基酸type,x表示骨架坐标
Graph Representation
节点 $v_i$ 特征表示为抗体的三种主链二面角,边特征由四部分组成, $O_i$ 是残基i的局部坐标系,图中的边被限制为 KKK-近邻图(KNN),每个节点只与最近的 K=8 个节点相连,以减少计算复杂度
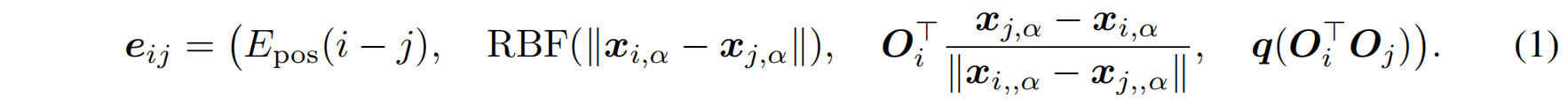
Iterative Refinement Graph Neural Network (RefineGNN)
输入是初始抗体图 $G^{(0)}$ ,其中所有的残基序列未知,用特殊符号<mask>初始化,每条边的初始距离设置为3|i-j|,方向和旋转特征初始化为0
输出是一个完整的抗体图 $G(s)$ ,包含所有节点的序列和3D结构信息
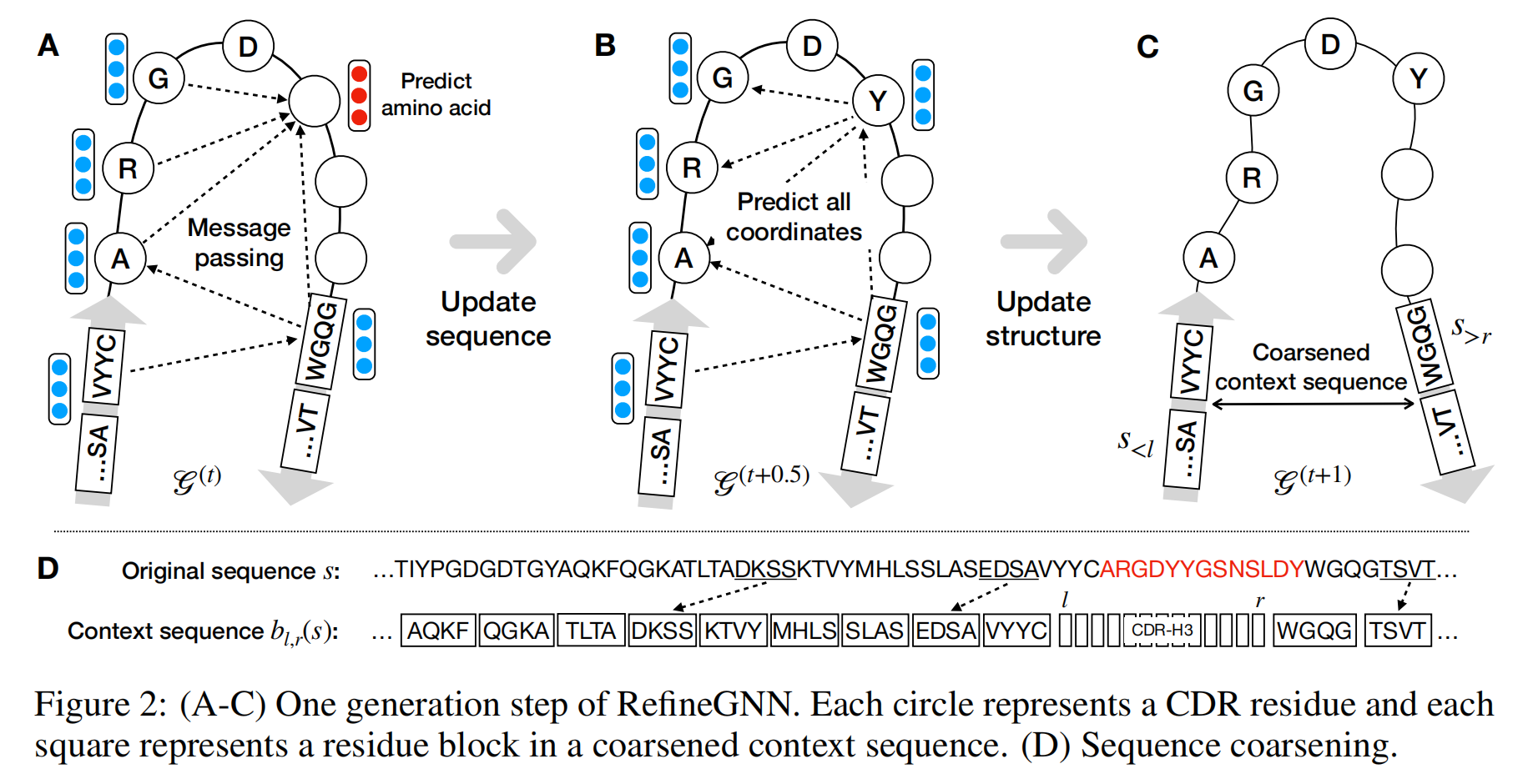
在每个生成步骤t中,主要分为以下三个过程:
-
图编码:
-
使用消息传递网络(Message Passing Network, MPN)编码当前图得到每个残基表示\
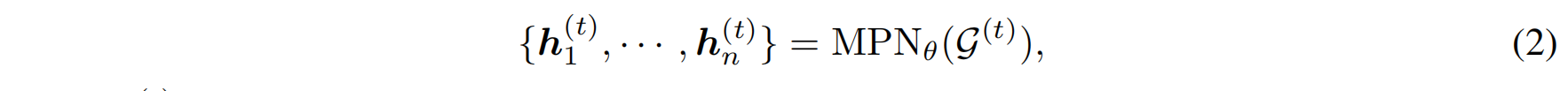
-
MPN使用多层消息传递更新节点
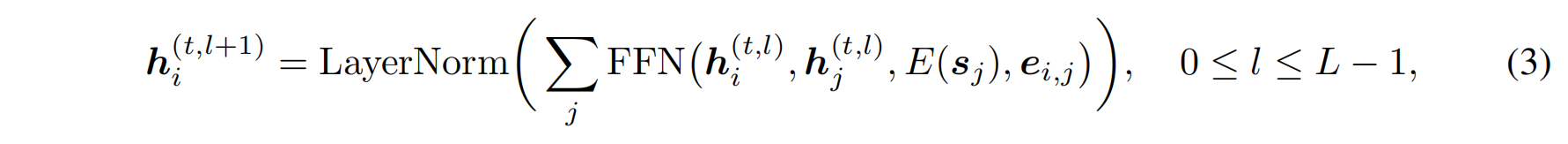
-
-
序列预测:
-
预测下一个残基(节点)的氨基酸类型,根据图编码器输出 $h_{t+1}^{(t)}$ 通过softmax进行预测,图2A
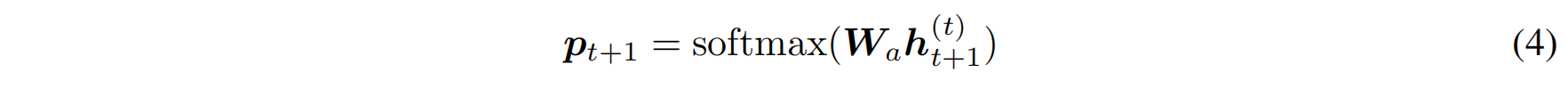
-
得到新的抗体图 $G^{(t+0.5)}$ ,其中新增残基的标签被更新,图2B
-
-
结构更新:
-
根据当前的抗体图,预测所有节点的 3D 坐标,图2C
-
使用一个独立的结构预测网络(另一个MPN参数为 $\tilde\theta$ ),对图进行编码并预测所有节点的坐标
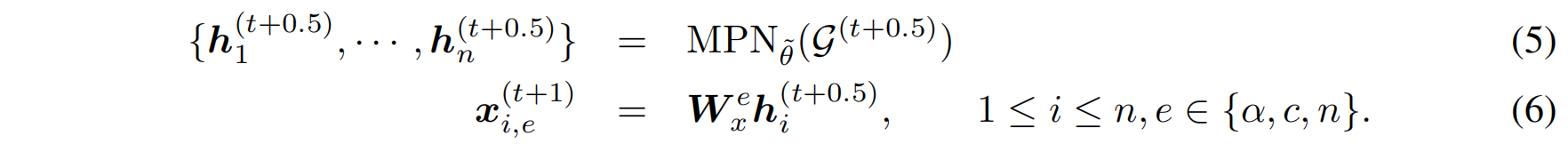
-
生成的新抗体图 $G^{(t+1)}$ ,其节点标签和边特征已更新
-
Training
- 序列预测:使用 teacher forcing 方法,将前 ttt 个残基设置为真实的氨基酸类型,其余节点设置为
。 - 结构预测:完全迭代细化,无需 teacher forcing,即每次更新结构时使用上一步预测的图作为输入。
Loss Function
总的来说包含序列和结构两部分损失 $L=L_\mathrm{seq}+L_\mathrm{struct}$
- $L_\mathrm{seq}$ 是交叉熵损失,衡量氨基酸序列的预测准确性
- $L_\mathrm{struct}$ 包含三部分:
* **距离损失**:预测与真实 Cα 原子间距离的 Huber 损失
<div align="center" style="float:center"><img src="https://blog-img-1259433191.cos.ap-shanghai.myqcloud.com/RefineGNN/frm7.png" alt="avatar" style="zoom:50%;" /></div>
* **二面角损失**:预测与真实二面角的均方误差。
<div align="center" style="float:center"><img src="https://blog-img-1259433191.cos.ap-shanghai.myqcloud.com/RefineGNN/frm8.png" alt="avatar" style="zoom:50%;" /></div>
* **Cα 角度损失**:预测与真实 Cα 原子之间角度和二面角的均方误差。
<div align="center" style="float:center"><img src="https://blog-img-1259433191.cos.ap-shanghai.myqcloud.com/RefineGNN/frm9.png" alt="avatar" style="zoom:50%;" /></div>
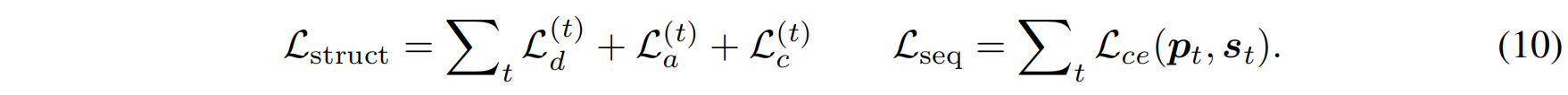
独立网络设计:序列预测和结构预测使用不同的 MPN 网络,使其专注于不同的任务
旋转和平移不变性:通过使用基于距离和角度的损失函数,保证模型对抗体图的几何变换不敏感
Conditional Generation Given the Framework Region
抗体设计任务的实际需求:在实际中,抗体的框架区通常是固定的,只需要设计CDR序列。因此,模型需要在已知框架区(上下文序列)的条件下生成CDR序列
目标:学习条件分布 $P(s^{\prime}|s_{<l},s_{>r})$ ,其中 $s’$ 是要生成的CDR区域,其他是CDR左侧和右侧上下文
为了利用框架区域的信息,作者引入了注意力机制,将框架区的信息传播到CDR生成的过程中
Conditioning via attention
上下文构造,直接拼接: $\tilde{s}=s_{<l}\oplus\mathrm{
用GRU对上下文进行编码,然后逐步预测序列、更新结构
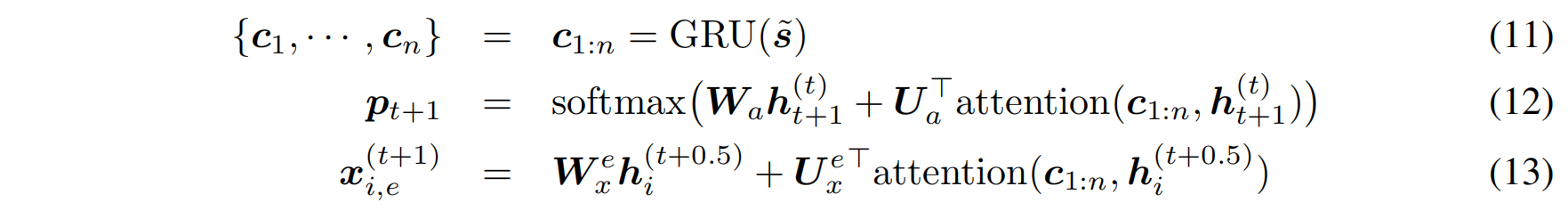
Multi-Resolution Modeling
注意力机制能够捕捉框架区域的序列信息,但无法建模框架区和CDR之间的空间交互。为了解决这个问题,作者引入了多分辨率建模,分为以下步骤:
粗粒度上下文表示:
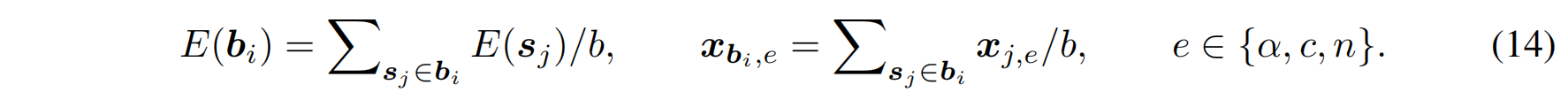
- 为了降低计算复杂度,将上下文序列按每b个残基聚合成一个残基块
- 每个残基块的嵌入和坐标通过其包含的残基平均值计算
基于残基块构造粗粒度上下文图的边由残基块的平均坐标计算得到,在生成过程中,RefineGNN 可以同时细化粗粒度图和CDR的局部精细图

条件生成的具体过程在 Algorithm 1 中展示
Property-Guided Sequence Optimization
生成新抗体序列,使其具有特定的目标属性(如抗原中和能力)。这一任务可以形式化为一个优化问题
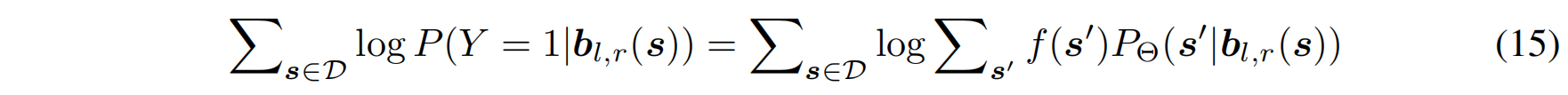
D是训练集抗体,Y是目标属性(例如中和能力)的二进制指标变量, $b_{l,r}(s)$ 固定的框架区上下文序列, $P(Y=1|b_{l,r}(s))$ 是条件生成模型的预测概率
- 在真实抗体结构数据上预训练生成模型,以学习CDR序列和结构的先验分布
- 随机从训练集中采样一个抗体序列 s,从中移除CDR序列,仅保留框架区上下文序列,利用生成模型生成M个新的候选序列
- 使用预训练的目标属性预测器 $f(s’)$ 来评估每个候选序列 $s’$ 的目标属性和分数,若 $f(s_i^{\prime})>\max(f(s),\mathrm{threshold})$ 则将该候选序列加入优化集 Q
- 从优化集采样一批新的抗体序列作为训练目标,最小化器生成损失 $L_\mathrm{seq}$
Experiments
Setup
-
语言建模与3D结构预测任务:
- 测量模型在生成新抗体序列时的困惑度(Perplexity, PPL)。
- 比较生成的 CDR 结构与真实结构之间的误差,使用 RMSD(Root Mean Square Deviation)评估。
-
抗原结合抗体设计任务:
- 在一个已建立的抗体设计基准数据集上评估 RefineGNN 的表现。
- 目标是生成能够与给定抗原结合的 CDR-H3 序列。
-
抗体优化任务:
- 从 SARS-CoV-2 数据集中重新设计抗体的 CDR-H3 区域,以增强其中和能力。
- 这种任务通常固定框架区,仅优化 CDR-H3,这是一种抗体工程中的常见实践
Baselines
-
LSTM(基于序列的模型):
- 不利用3D结构信息,仅使用序列信息进行抗体生成。
- 包括一个编码上下文序列的编码器、生成CDR序列的解码器以及一个连接二者的注意力层。
-
AR-GNN(自回归图生成模型):
- 使用图神经网络按残基逐步生成抗体图。
- 无法在生成过程中修改部分生成的3D结构。
-
RAbD(Rosetta Antibody Design):
- 基于物理的抗体设计方法。
- 包括 CDR 片段的选取、结构的能量最小化以及多轮序列设计。
- 仅用于抗原结合设计任务,不适用于 SARS-CoV-2 优化任务,因为该任务需要预定义的3D结构。
hidden dimension为256,块大小为b = 4
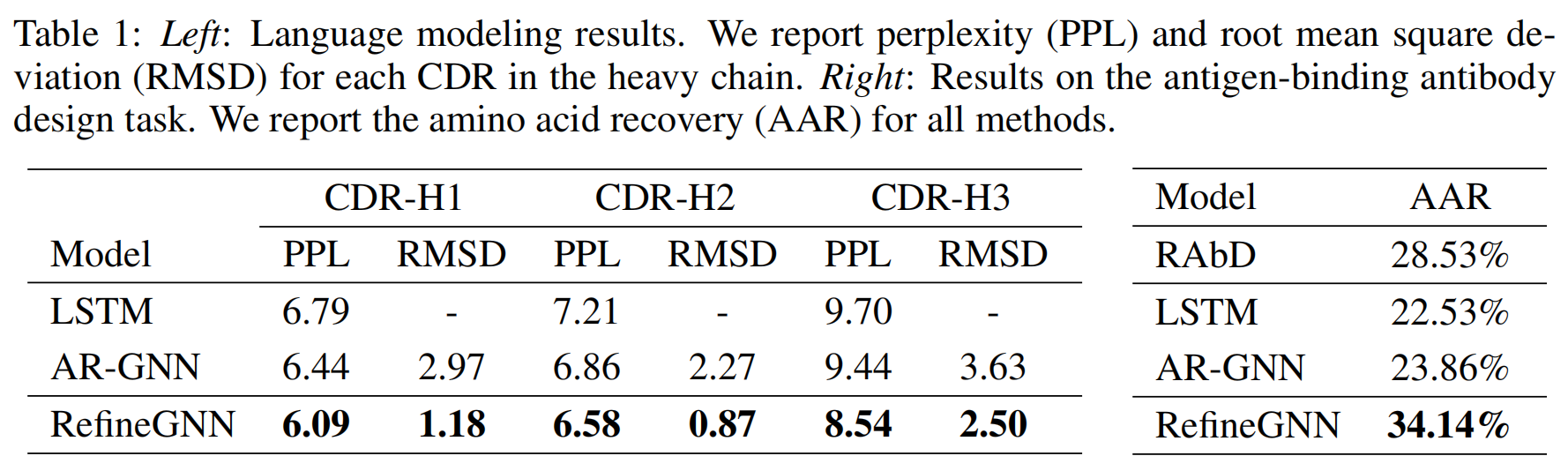
Language Modeling and 3D Structure Prediction
数据
- 使用 SAbDab(Structural Antibody Database)数据集,有4994抗体结构。
- 数据集被分为训练、验证和测试集,基于 CDR 的序列簇划分(例如 CDR-H3)。
评估指标
-
困惑度(PPL):
- 衡量模型生成序列的合理性。
- PPL 越低,表示模型生成的序列越贴近真实数据分布。
-
RMSD(Root Mean Square Deviation):
- 比较生成结构与真实结构的偏差。
- RMSD 越低,表示生成结构越精
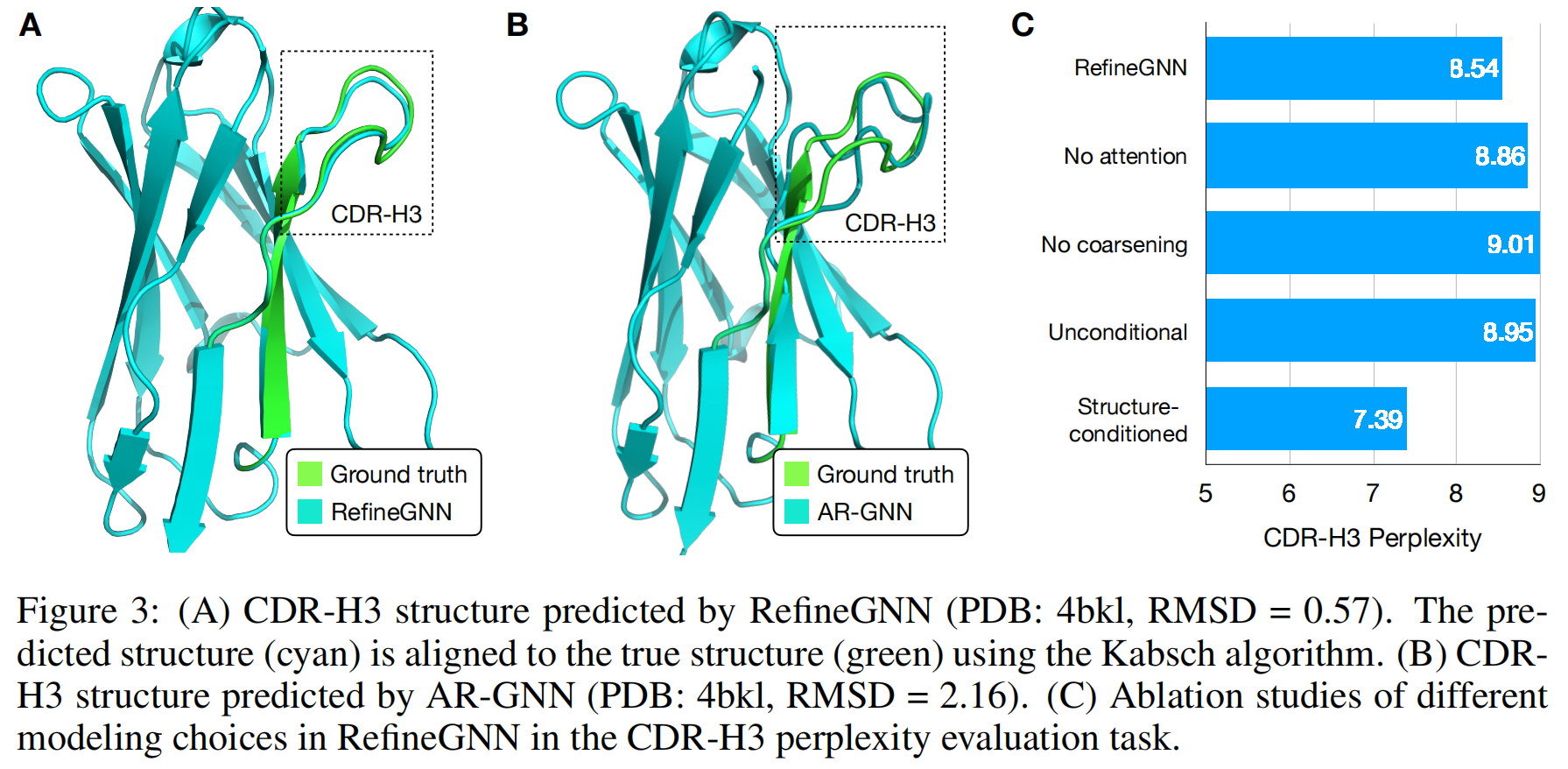
可视化展示了 RefineGNN 的预测结果如何更准确地匹配真实抗体结构
Antigen-Binding Antibody Design
数据
- 使用了 Adolf-Bryfogle et al. (2018) 提出的抗体设计基准数据集。
- 数据集包含 60 个抗体-抗原复合体,用于评估抗体设计任务。
- 给定抗体的框架结构(framework region),目标是设计其 CDR-H3 片段,使其能够与对应的抗原结合。
- 简化条件:所有方法在生成 CDR-H3 时未使用抗原的结构信息。抗原条件生成方法被留作未来工作
RefineGNN 的 AAR 得分为 34.14%,显著高于所有基线模型(提升约 7%),表1
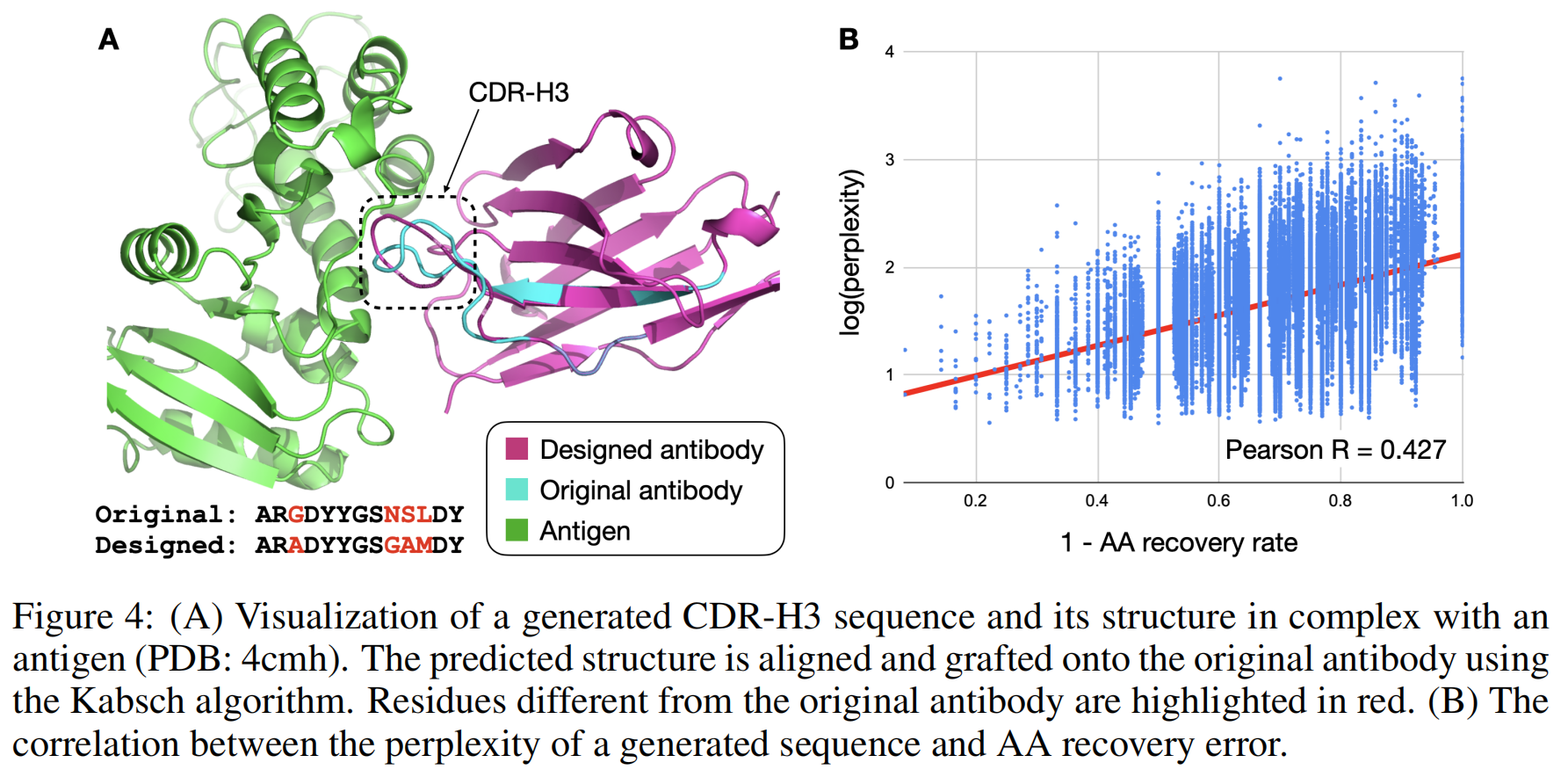
- 图 4A 展示了生成的 CDR-H3 序列与抗原复合体结构的示例。
- 通过 Kabsch 算法对预测的结构进行对齐和嫁接,并用红色高亮显示与原始抗体序列不同的残基。
- 研究发现,具有较低困惑度的序列往往具有较低的氨基酸恢复误差(Pearson 相关系数 R=0.427)。
- 困惑度可以作为抗体设计任务中候选序列排序的有效标准
SARS-CoV-2 Neutralization Optimization
数据 CoVAbDab(Coronavirus Antibody Database):
- 包含 2411 个抗体数据,每个抗体都与多个二元标签关联,指示其是否在特定表位中和冠状病毒(包括 SARS-CoV-1 和 SARS-CoV-2)。
- 数据集按照 CDR-H3 的簇(cluster)划分为 训练集、验证集 和 测试集,比例为 8:1:1。
中和能力预测器
-
目标:评估抗体是否具有 SARS-CoV-2 的中和能力。
-
输入:抗体的重链序列(VH sequence)。
-
模型结构:
- 每个残基被嵌入到一个 64 维的向量中。
- 嵌入向量经过一个 SRU 编码器(简单递归单元,Simple Recurrent Unit)。
- 使用平均池化(average-pooling)层后,连接一个两层的前馈神经网络。
- 输出两个概率值 p1 和 p2,分别代表抗体中和 SARS-CoV-1 和 SARS-CoV-2 的能力。
- 得分函数:选择中和 SARS-CoV-2 的概率 f(s)=p2 作为中和分数。
CDR 序列约束
为确保生成的抗体序列可用于治疗性抗体设计,引入以下约束条件:
-
净电荷:
- 序列的净电荷必须在 -2.0 至 2.0 范围内。
- 净电荷的定义基于残基类型(例如正电荷的 R、K 和负电荷的 D、E)。
-
避免糖基化:
- 序列中不得包含易于糖基化的 N-X-S/T 模式(其中 X 表示任意氨基酸)。
-
氨基酸重复:
- 序列中任意氨基酸不能连续重复超过 5 次(如 “SSSSS” 不合法)。
-
困惑度约束:
- 生成序列的困惑度(Perplexity, PPL)必须低于 10。
- PPL 由 LSTM、AR-GNN 和 RefineGNN 模型分别计算,确保比较公平。
评估指标
-
中和分数:
-
对于测试集中每个抗体,生成 100 条新的 CDR-H3 序列。
-
将这些序列与抗体上下文序列拼接形成完整的 VH 序列。
-
使用中和能力预测器 f 评估生成抗体的中和分数:
- 若生成序列满足所有约束条件,则直接使用预测的分数 f(s′)。
- 否则,使用原始抗体的分数作为对比。
-
-
困惑度(PPL):
-
使用训练于 SAbDab 数据的预训练模型,评估 CoVAbDab 测试集上的困惑度
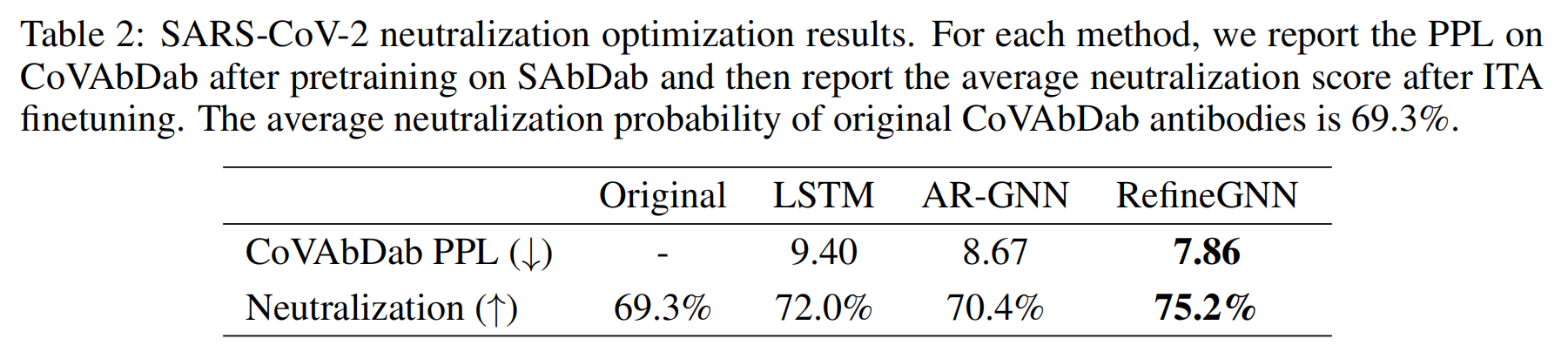
模型在 SAbDab 数据上预训练后,使用 ITA 算法(Iterative Target Augmentation)在 CoVAbDab 上微调。


