论文地址:FlowDesign:Improved Design of Antibody CDRs Through Flow Matching and Better Prior Distributions
FlowDesign:DiffAb+FlowMatching做co-design
Abstract
介绍了一种名为 FlowDesign 的新方法,用于 抗体互补决定区(CDR) 的 序列-结构联合设计。FlowDesign 主要基于 Flow Matching 技术,相较于传统的 扩散模型(diffusion models),它具有以下优势:
- 灵活的先验分布选择:可以集成不同的先验知识,如 蛋白质语言模型(PLM) 和 数据驱动的结构模型,从而提高设计质量。
- 直接匹配离散分布:避免了在氨基酸类型上应用连续噪声导致的不稳定性。
- 高计算效率:相比扩散模型,FlowDesign 在 大规模抗体设计 任务中 计算成本更低。
研究中,FlowDesign 在 氨基酸恢复率(AAR)、均方根偏差(RMSD)和 Rosetta 能量 方面均优于现有方法。此外,研究团队还利用 FlowDesign 重新设计了 HIV-1 抗体 Ibalizumab 的 HCDR3 序列,并通过 生物层干涉(BLI) 和 伪病毒中和实验 证明其改进版本在多种 HIV 突变株上表现更优。最终,FlowDesign 在 抗体设计和蛋白质工程 领域展示了 良好的应用潜力。
Introduction
抗体是免疫系统中至关重要的成分,能够通过与特定抗原结合中和病原体。设计能够特异性地靶向特定抗原的抗体,对药物研发和生物学研究具有重要意义。抗体的互补决定区(CDR)是决定抗体与抗原结合的关键区域,直接影响抗体的亲和力和特异性。因此,抗体设计的核心任务之一就是设计具有优异亲和力和特异性的CDR序列。
然而,由于CDR的组合空间过于庞大,传统的实验方法,如体外筛选,往往受到高成本和时间限制。因此,计算方法显得尤为重要。通过模拟和机器学习模型,可以有效缩小实验验证的搜索范围并提高设计效率。传统的计算方法通常通过模拟来预测抗体与抗原的结合,但这些方法往往依赖于经验能量函数,且计算消耗大,容易陷入局部最优解。
现有方法的缺陷:
-
传统的 序列生成模型 基于序列的稳定性进行优化,但 抗体和抗原的结合亲和力 依赖于 抗体-抗原复合物的三维结构,而现有的序列模型往往缺乏结构建模能力。
-
扩散模型:尽管扩散模型在抗体设计中取得了一定进展,但仍然面临以下挑战:
- 先验分布问题:扩散模型常用标准高斯分布作为先验,但这无法充分反映抗体分子的物理可行性。
- 离散氨基酸的处理问题:扩散模型的连续性与氨基酸类型的离散性不兼容,导致模型在生成过程中出现不平滑的转换。
- 计算代价高:扩散模型需要多次迭代去噪,这使得大规模采样变得计算成本过高。
近年来,深度学习模型,尤其是蛋白质语言模型(PLM),被广泛应用于抗体设计。这些模型能够从大量已知蛋白质数据中学习序列的“语法”,从而生成具有潜力的抗体序列。然而,现有的序列生成模型主要侧重于序列稳定性的提升,往往忽视了抗体与抗原之间的结构性关系。抗体的亲和力不仅仅依赖于其序列,还涉及到抗体与抗原的三维结构,而现有的序列模型缺乏对抗体结构的建模能力。
扩散模型作为近年来的一种新兴方法,虽然在抗体设计中取得了一定的进展,但仍然存在一些显著的缺陷。首先,扩散模型通常使用标准的高斯分布作为先验,这无法反映抗体分子的物理可行性。其次**,扩散模型的连续性与氨基酸类型的离散性不兼容**,容易导致模型在生成过程中出现不平滑的过渡,影响结果的准确性。最后,扩散模型需要多次迭代去噪,这导致其在大规模采样时计算代价过高。
为了克服这些问题,本文提出了一种名为FlowDesign的新方法,基于Flow Matching技术来进行抗体的序列-结构联合设计。FlowDesign的创新之处在于,它能够灵活选择先验分布,结合不同类型的先验知识,如基于蛋白质语言模型或数据驱动的结构模型。同时,FlowDesign通过直接匹配氨基酸类型的离散分布,避免了扩散模型中不平滑的生成过程,能够更有效地生成抗体序列。与传统扩散模型相比,FlowDesign在计算效率上具有显著优势,适用于大规模的抗体设计任务。将 CDR 设计视为 传输映射问题(Transport Mapping Problem),通过学习将任意初始分布直接映射到目标分布。与常规的多步“扩散”模型不同,FlowDesign 采用了一步式映射,因此具有以下优点:首先,它在选择先验分布时非常灵活,能够集成多种不同的先验知识;其次,FlowDesign 能够直接匹配离散分布,避免了在氨基酸类型上的不平滑生成过程;最后,它的高计算效率使得大规模采样变得可行,并且计算成本较低。
通过大量的实验评估,首先评估了不同形式的先验知识对抗体设计的影响,这些先验知识包括从 蛋白质语言模型、物理化学函数 以及 结构数据驱动模型 中提取的分布
FlowDesign在多个指标上,如氨基酸恢复率(AAR)、均方根偏差(RMSD)和Rosetta能量,均优于现有的其他方法。该方法不仅成功地优化了HIV-1抗体Ibalizumab的设计,还展示了在多种HIV变种上的显著改善,尤其是在结合亲和力和中和效力方面。
Results
Overview of FlowDesign
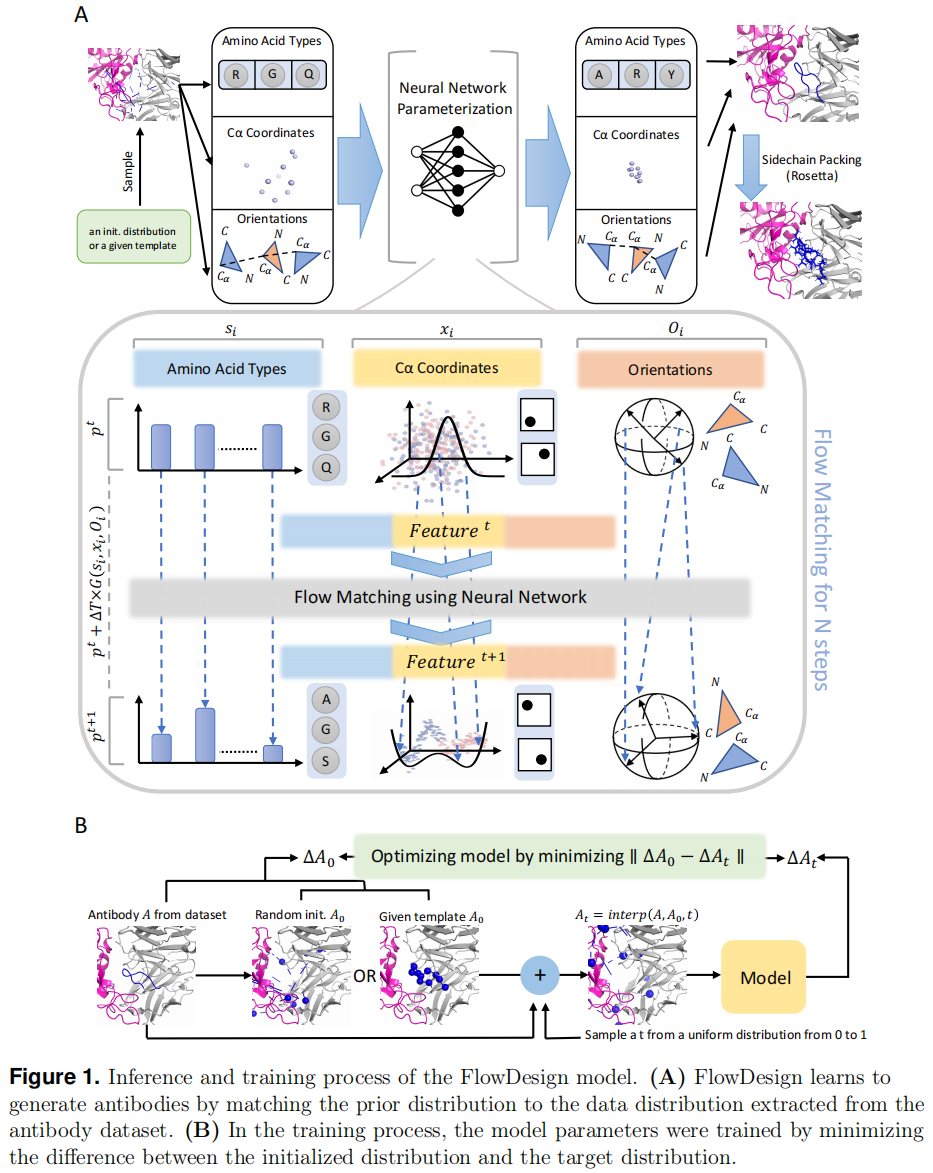
FlowDesign 采用了序列-结构联合设计(sequence-structure co-design)方法,用于优化抗体 CDR。抗体被表示为 {si, xi, Oi},分别对应氨基酸类型、Cα 原子坐标以及主链取向。在设计过程中,FlowDesign 以抗体-抗原复合物为输入,并对待优化的 CDR 进行初始化。CDR 的初始状态来自于某种先验分布,而该分布可以灵活选择。接下来,FlowDesign 通过预测漂移力(drift forces),将 CDR 从初始分布映射到目标分布(图 1A)。
在建模过程中,FlowDesign 允许两种工作模式:
- CDR 设计模式:给定抗体框架和抗原复合物,仅优化 CDR 的序列和结构。
- 抗体-抗原复合物优化模式:固定抗体框架的氨基酸类型和结构,并结合 dyMEAN 和 AlphaFold3 进行复合物预测,然后优化 CDR。
在推理阶段,FlowDesign 结合 Rosetta 进行侧链构建和结构优化,去除原子碰撞并提升模型预测的合理性。同时,在训练过程中,FlowDesign 直接匹配初始分布和目标分布,而非像扩散模型那样进行多步去噪。因此,它具备更快的计算速度和更好的生成稳定性(图 1B)。
Performance for different prior distributions
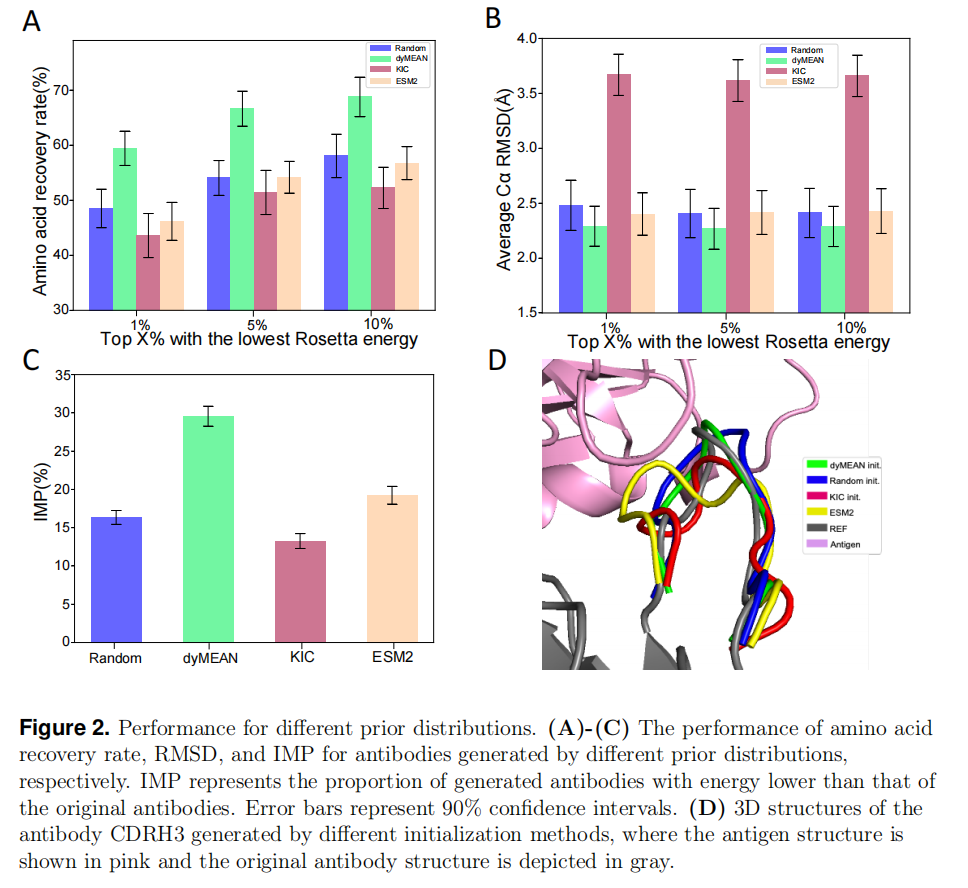
FlowDesign 允许灵活选择先验分布,以确保更优的抗体序列和结构。研究对比了四种不同的先验分布:
- 随机分布(Random Distribution)
- dyMEAN 生成的序列-结构联合分布
- 基于 KIC(Kinematic Closure)的结构分布
- 基于蛋白质语言模型(PLM,如 ESM2)的序列分布
实验针对 CDRH3 进行了训练和评估,因为 CDRH3 是抗体结合抗原的关键区域,且多样性最高。结果显示:
- 采用 dyMEAN 先验分布 的模型表现最佳,在 氨基酸恢复率(AAR) 上比随机初始化提高 约 15%(图 2A)。
- dyMEAN 初始化 生成的抗体结构的 RMSD 最低(平均 2.291Å),说明它生成的 CDR 结构最接近真实结构(图 2B)。
- KIC 方法 由于计算成本较高,生成的样本数量有限,因此效果不如 dyMEAN。
- PLM(ESM2) 方法的序列多样性较大,但由于缺乏结构信息,导致其恢复率较低,无法为 CDR 结构提供良好的初始约束。
综上,dyMEAN 作为先验分布最有效,因为它同时提供了抗体的序列和结构信息,使得 FlowDesign 能够更准确地优化 CDR 设计。
Performance for Sequence-Structure Co-design
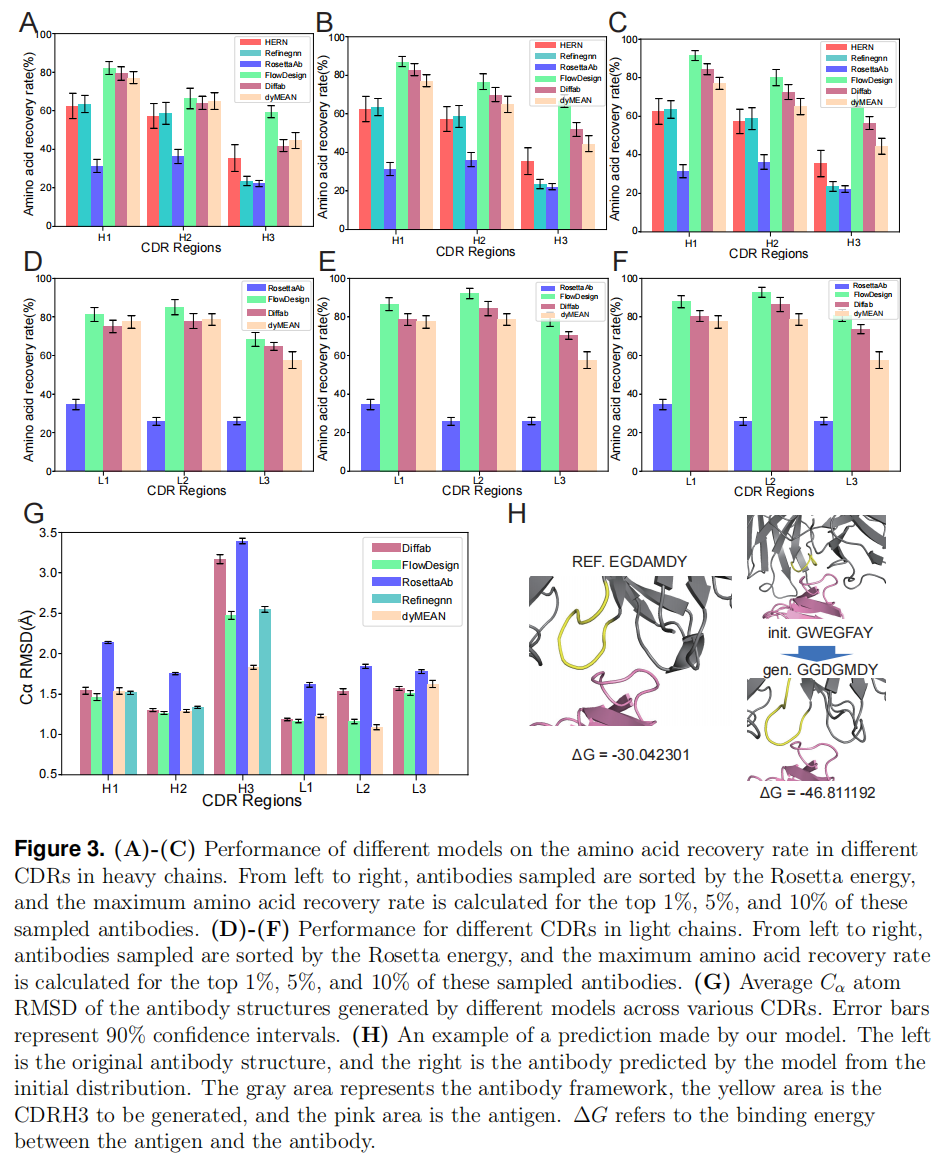
FlowDesign 还与多个抗体设计基准方法进行了对比,包括:
- HERN(仅生成序列),RefineGNN(仅优化结构),RosettaAb(基于经验能量的优化),Diffab(基于扩散模型的联合设计),dyMEAN(基于全原子建模的优化)
评估实验针对 CDRH1、CDRH2、CDRH3、CDRL1、CDRL2、CDRL3 进行,主要指标包括:
- 氨基酸恢复率(AAR)(图 3A-3F)
- 均方根偏差(RMSD)(图 3G)
- Rosetta 能量(图 3H)
实验发现:
- FlowDesign 在 CDRH3 设计方面优势显著,其氨基酸恢复率超过 60%,远高于其他方法(图 3A)。
- RMSD 方面,FlowDesign 生成的抗体结构比大多数基准方法更接近真实结构,仅次于 dyMEAN(图 3G)。
- 能量优化 方面,FlowDesign 生成的抗体具有更低的 Rosetta 能量,表明它们在结合稳定性和亲和力方面更优(图 3H)。
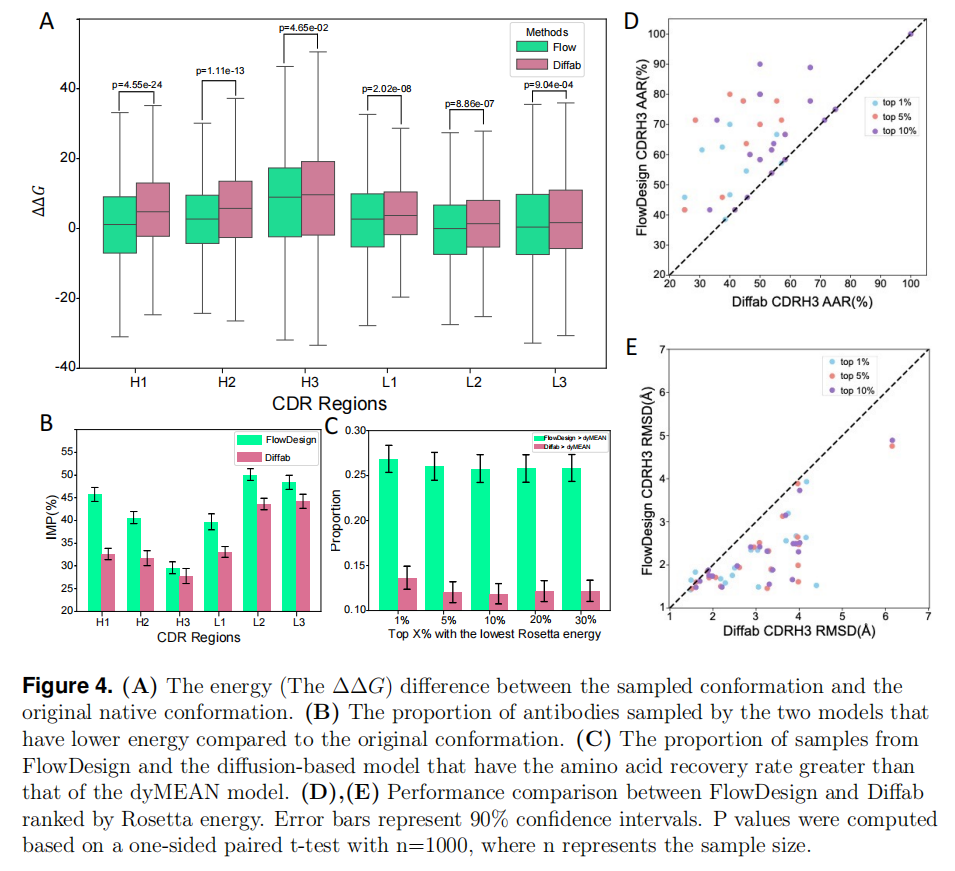
FlowDesign 在与 Diffab 等其他生成模型进行对比时,展示了其在抗体设计中的优势,特别是在 氨基酸恢复率 和 结构优化 方面。实验结果表明,FlowDesign 的表现超越了 Diffab 等传统方法,尤其在 能量优化 和 抗体多样性 上表现更好。以下是详细结果:
- 能量差异:FlowDesign 生成的抗体相比 Diffab 具有更低的 ΔΔG(即能量变化)。在图 4A 中,FlowDesign 生成的抗体有更多的候选抗体能量低于原始抗体,特别是在 CDRH1 区域,改进幅度达到 13.11%。
- 氨基酸恢复率:在能量筛选后,FlowDesign 生成的抗体表现出比 dyMEAN 生成的抗体更高的 氨基酸恢复率,表明 FlowDesign 更好地整合了先验知识,并且学会了更加精确的 序列-结构联合分布(图 4B)。
- 多样性与稳定性:FlowDesign 在 大规模采样 下展示了 更高的多样性,即使在样本数量有限的情况下,它仍能提供更多高质量的候选抗体。图 4D 和 4E 展示了 FlowDesign 在不同的 能量筛选比例 下的表现,与 Diffab 进行对比,结果表明 FlowDesign 在 结构恢复 和 能量优化 上都具有明显优势。
Performance for designing CDRs based solely on sequences
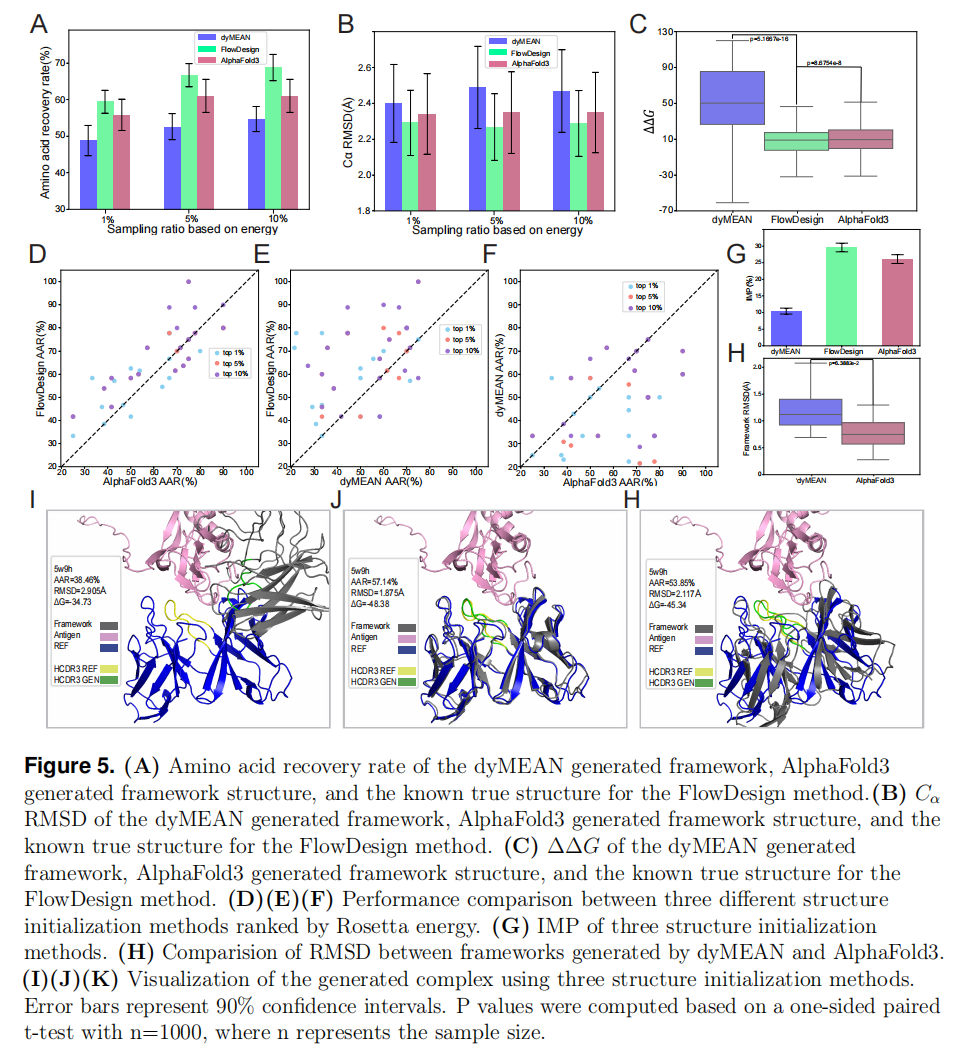
在现实应用中,抗体的三维结构可能未知,仅有序列信息可用。因此,研究进一步评估了 FlowDesign 在仅依赖 抗体框架序列 进行 CDR 设计的能力。
研究使用了两种方法预测抗体框架结构:
- dyMEAN 生成框架结构
- AlphaFold3 预测框架结构
之后,FlowDesign 设计了 CDRs,并与真实结构的设计进行了对比。以下是关键结果:
- 氨基酸恢复率(AAR):在图 5A 中,使用 AlphaFold3 生成框架结构 的 FlowDesign 在 氨基酸恢复率 上表现优异,接近真实结构的设计方法。相比之下,使用 dyMEAN 生成框架结构 的 FlowDesign 表现较差,显示出 AlphaFold3 提供的框架结构更适合进行 CDR 设计。
- RMSD 和能量:在图 5B 和 5C 中,AlphaFold3 生成的框架结构 所设计的 CDR 在 RMSD 和 ΔΔG (能量差异)方面表现最好,表明它生成的 CDR 结构和能量最接近实际的结构,并且具有较高的稳定性。
- 框架生成质量:图 5H 显示了三种不同框架生成方法的比较:dyMEAN、AlphaFold3 和真实结构。结果显示,AlphaFold3 预测的框架 更加接近真实结构,生成的抗体抗原复合物与真实结构的重合度更高,表现出较低的 RMSD 和较低的 能量(图 5G、5J、5K)。
这些实验结果表明,FlowDesign 能够在缺乏真实结构的情况下,通过 AlphaFold3 预测框架的结构来设计高质量的 CDR,从而展示了其在实际应用中的 强大灵活性 和 鲁棒性。
Performance for HIV antibody design
FlowDesign 还被应用于 HIV-1 抗体设计,特别是优化 Ibalizumab 抗体的 CDRH3。实验过程中:
-
生成 50,000 种不同的 CDRH3 变体,并使用 Rosetta 能量评分筛选出 12,000 个高质量候选抗体。
-
构建酵母展示文库,并进行 三轮 FACS(荧光激活细胞分选) 以筛选结合 CD4 受体能力最强的抗体。
-
选出结合能力最强的 10 种抗体,并进一步选择 3 种最低能量的候选抗体(imab-mut-1、imab-mut-2、imab-mut-3)。
-
BLI(生物层干涉)实验 评估抗体结合能力,结果显示新设计的抗体与 CD4 结合能力比原始 Ibalizumab 更强。
-
伪病毒中和实验 评估新抗体对 11 种 HIV-1 亚型(A、B、C、CRF01、CRF07) 的中和能力:
- imab-mut-2 在 6 种 HIV-1 亚型上的中和能力优于 Ibalizumab。
- 其 IC50 值比 Ibalizumab 低 4 倍,表明它能以更低浓度有效中和病毒。
这些实验结果表明,FlowDesign 能够成功优化 HIV-1 抗体,提高其结合亲和力和中和能力,展示了该方法在 生物制药 领域的广阔应用前景。
Performance for HIV antibody design
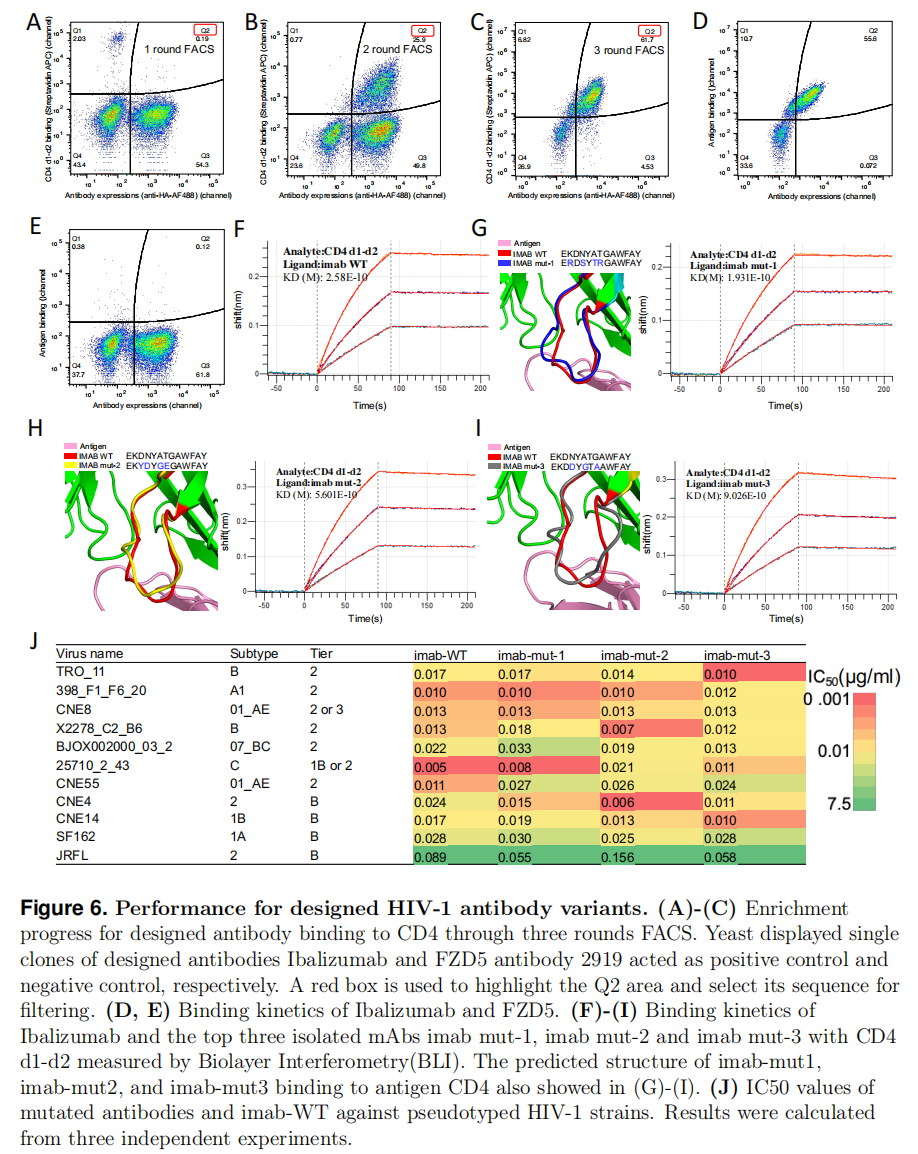
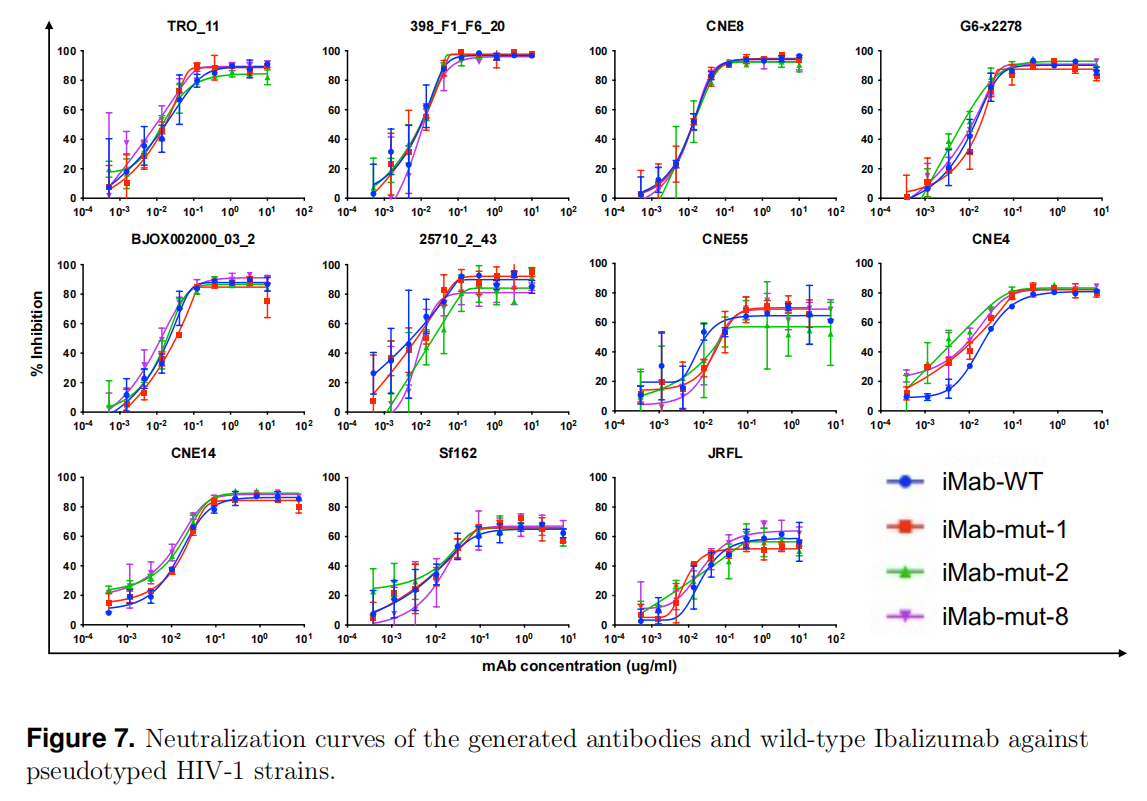
FlowDesign 还被应用于 HIV-1 抗体设计,特别是优化 Ibalizumab 抗体的 CDRH3。实验过程中:
-
生成 50,000 种不同的 CDRH3 变体,并使用 Rosetta 能量评分筛选出 12,000 个高质量候选抗体。
-
构建酵母展示文库,并进行 三轮 FACS(荧光激活细胞分选) 以筛选结合 CD4 受体能力最强的抗体(图 6A-C)。
-
选出结合能力最强的 10 种抗体,并进一步选择 3 种最低能量的候选抗体(imab-mut-1、imab-mut-2、imab-mut-3)。
-
BLI(生物层干涉)实验 评估抗体结合能力,结果显示新设计的抗体与 CD4 结合能力比原始 Ibalizumab 更强(图 6F-6I)。
-
伪病毒中和实验 评估新抗体对 11 种 HIV-1 亚型(A、B、C、CRF01、CRF07) 的中和能力:
- imab-mut-2 在 6 种 HIV-1 亚型上的中和能力优于 Ibalizumab(图 7)。
- 其 IC50 值比 Ibalizumab 低 4 倍,表明它能以更低浓度有效中和病毒。
这些实验结果表明,FlowDesign 能够成功优化 HIV-1 抗体,提高其结合亲和力和中和能力,展示了该方法在 生物制药 领域的广阔应用前景。
Methods
Baselines
在评估 FlowDesign 的性能时,研究团队选择了多个 抗体设计领域的基线方法 进行比较:
- Diffab(扩散模型,约 400 万参数):可以同时生成大规模 CDR 结构和序列。
- RefineGNN(基于图神经网络,约 600 万参数):通过迭代优化抗体序列和结构。
- dyMEAN(全原子模型,约 200 万参数):基于等变编码器更新抗体序列和结构。
- HERN(分层等变优化,约 700 万参数):专门用于 CDR 序列优化。
为了确保公平对比,研究团队重新划分了数据集,并重新训练了所有基线模型。在测试 Diffab 这类生成模型时,研究团队采样了 相同数量的抗体 进行评估,而对于 非生成模型(如 RefineGNN),则直接测试其输出结果。
此外,抗体的结合能计算由 PyRosetta 的 InterfaceAnalyzerMover 进行计算,使用 REF2015 作为能量权重设置。
Model Input
FlowDesign 采用 抗体-抗原复合物 作为输入,表示为:
$$
X = {s_i, x_i, O_i}
$$
- $s_i$:第 $i$ 个残基的氨基酸类型(20 维 one-hot 编码)。
- $x_i$:Cα 原子的三维坐标(3D 坐标)。
- $O_i$:骨架方向(旋转矩阵表示 SO(3))。
此外,CDR 片段的 初始状态 记为:
$$
T = {T s_i, T x_i, T O_i}
$$
其中 $T$ 由 先验分布 采样得到(如 dyMEAN 生成的结构)。
FlowDesign 的优化目标
FlowDesign 通过 学习一个映射:
$$
T(X) \to X_{\text{target}}
$$
- $T(X)$ 是模型生成的抗体 CDR 片段
- $X_{\text{target}}$ 是数据集中的真实抗体 CDR 片段
该映射由 流匹配(Flow Matching)网络 进行建模。
Dataset
(1) 数据来源
FlowDesign 在 SAbDab(Structural Antibody Database) 上训练:
- 包含 13,279 个抗体-抗原复合物。
- 仅选择 蛋白抗原(去掉非蛋白抗原的数据)。
- 过滤掉分辨率 低于 4Å 的结构。
- 数据划分:训练集:80%,验证集:5%,测试集:15%
- 抗体 CDR 片段长度:
- CDRH3:12.3(训练集),11.3(测试集)
- 其他 CDR 片段长度范围 5.8~12.5
- 抗体 CDR 片段长度:
(2) 先验分布
FlowDesign 允许选择不同的 先验分布 进行初始化:随机分布(standard Gaussian),dyMEAN 生成的结构(数据驱动先验),KIC 运动学闭合法,ESM2 蛋白语言模型
Loss Function
FlowDesign 的损失函数由 四部分组成,分别优化:氨基酸类型(Amino Acid Type),Cα 原子坐标(Cα Coordinate),骨架方向(Backbone Orientation),整体流匹配损失
(1) 氨基酸类型损失
使用 均方误差(MSE):
$$
L_{\text{seq}} = \mathbb{E} \left[ \frac{1}{m} \sum_{k=1}^{m} \text{MSE}(s_k - T s_k, F(R^t, C)) \right]
$$
- $s_k$ 是真实的氨基酸类型(one-hot),$T s_k$ 是初始先验分布,$F(R^t, C)$ 是模型预测的氨基酸类型变换
(2) Cα 原子坐标损失
使用 均方误差(MSE):
$$
L_{\text{pos}} = \mathbb{E} \left[ \frac{1}{m} \sum_{k=1}^{m} \text{MSE}(x_k - T x_k, G(R^t, C)) \right]
$$
- $x_k$ 是目标坐标,$T x_k$ 是初始分布的坐标,$G(R^t, C)$ 是模型预测的坐标变换
(3) 骨架方向损失
使用 四元数(quaternion) 表示方向,并计算误差:
$$
L_{\text{qua}} = \mathbb{E} \left[ \frac{1}{m} \sum_{k=1}^{m} \text{MSE}(q_k \wedge T q_k^*, H(R^t, C)) \right]
$$
- $q_k$ 是 SO(3) 旋转矩阵转换成的四元数,$T q_k$ 是初始状态的四元数,$H(R^t, C)$ 是模型预测的方向变换
此外,还有 SO(3) 旋转矩阵损失:
$$
L_{\text{ori}} = \mathbb{E} \left[ \frac{1}{m} \sum_{k=1}^{m} | O_k^T (T O_k Q(H(R^t, C))) - I | \right]
$$
(4) 最终损失函数
将以上损失函数加权求和:
$$
L = \mathbb{E}{t \sim U(0,1)} \left[ L{\text{seq}} + L_{\text{pos}} + \alpha L_{\text{qua}} + \beta L_{\text{ori}} \right]
$$
- $\alpha, \betaα$ 为权重超参数,训练时动态调整:
- 前 200K 轮:$\alpha = 1, \beta = 0$(先优化四元数损失)
- 后 5K 轮:$\alpha = 0, \beta = 1$(优化 SO(3) 损失)


