论文地址:IgGM:A Generative Model for Functional Antibody and Nanobody Design
IgGM:两阶段PLM+Diffusion生成抗体抗原结构
Abstract
免疫球蛋白(Immunoglobulins)是由免疫系统产生的关键蛋白,能够识别并结合外来物质,在防御感染和疾病方面发挥重要作用。特异性抗体的设计为疾病治疗开辟了新的途径。随着深度学习的发展,AI 驱动的药物设计成为可能,并推动了多种抗体设计方法的出现。然而,许多现有方法依赖额外的实验条件,与实际应用场景存在差距,使得这些方法难以直接融入现有的抗体设计流程。为了解决这一问题,我们提出了 IgGM,一个用于 免疫球蛋白 de novo 设计 的生成模型,能够针对特定抗原同时生成抗体序列和三维结构。IgGM 由 三大核心模块 组成:预训练语言模型(用于提取序列特征)、特征学习模块(用于识别关键特征)、预测模块(用于生成抗体序列并预测完整的抗体-抗原复合体结构)。IgGM 不仅在结构预测任务中表现出色,还能设计具有新颖性的抗体和纳米抗体,这使其在抗体设计的多种实际应用场景中具有很高的实用价值。
Introduction
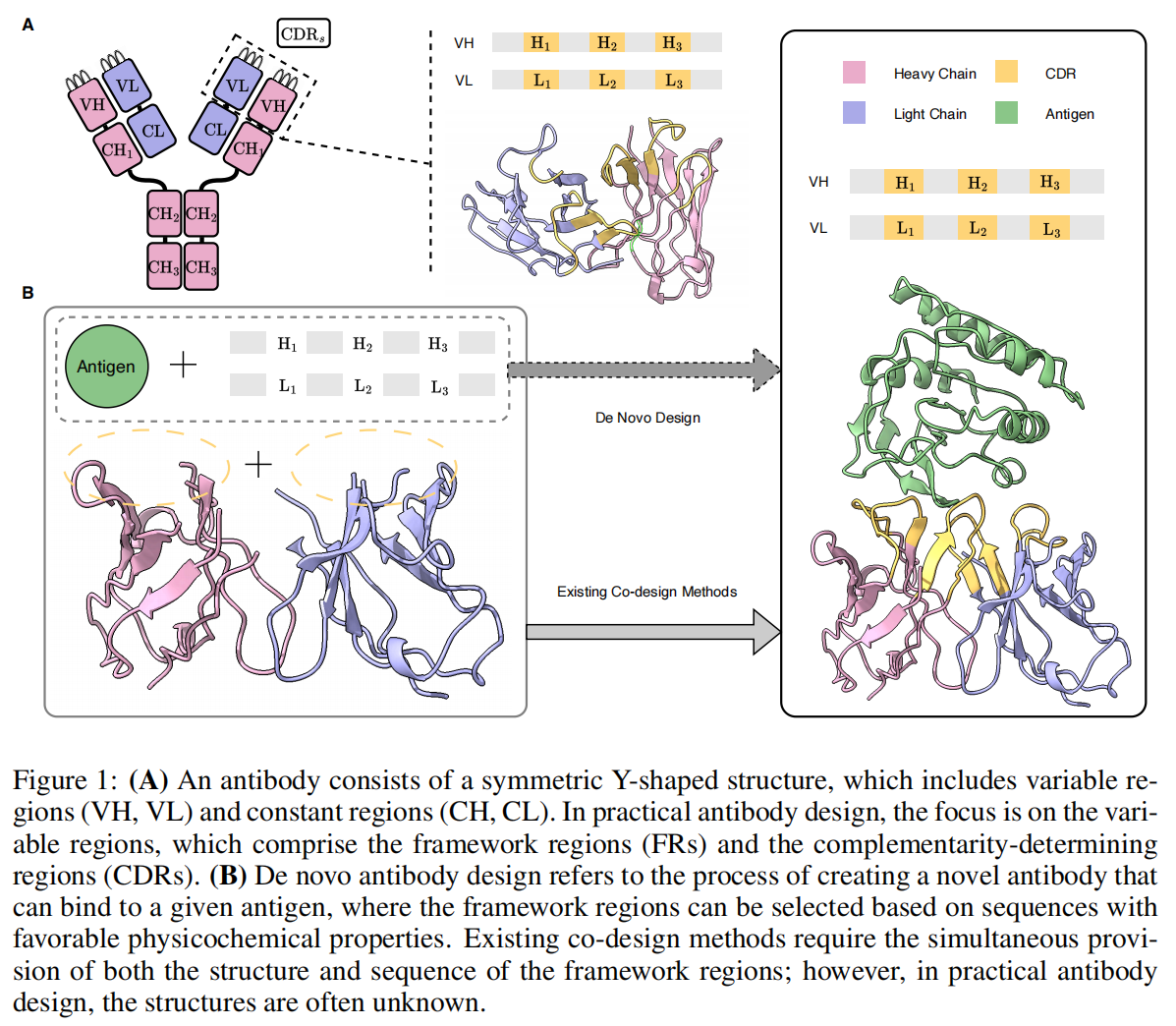
抗体(Antibodies),也称为免疫球蛋白(Ig),是由 B 淋巴细胞分泌的 Y 形蛋白,主要存在于血液和淋巴液中(Silverthorn, 2015; Akkaya et al., 2020)。如 Figure 1(A) 所示,抗体由 两条重链(heavy chains)和两条轻链(light chains) 组成,每条链包含 可变区(VH 或 VL) 和 恒定区(CH 或 CL)。可变区包括 三个互补决定区(CDRs),这些 CDR 负责抗原结合,并决定抗体的特异性。此外,可变区还包含 四个框架区(FRs),其主要作用是提供结构支持,并且其序列变化较小。抗体在免疫系统中的核心功能是 识别并结合特定外来物质(如细菌、病毒、真菌和寄生虫),并通过免疫反应清除这些外来物质(Schroeder Jr & Cavacini, 2010; Litman et al., 1993)。抗体在医学、科学研究和生物技术领域具有重要价值,被广泛应用于 疾病治疗、个性化医疗、疫苗开发和新药研发(Nelson et al., 2010; Weiner, 2015; Sliwkowski & Mellman, 2013)。
然而,尽管抗体在各个领域具有广泛的应用价值,传统的抗体生产方法仍然面临诸多挑战,例如 生产周期长(Georgiou et al., 2014)、批次间变异性大(Bradbury et al., 2018),以及为了降低免疫原性而进行人源化改造的需求(Safdari et al., 2013)。这些问题限制了抗体在临床和工业中的广泛应用,并影响了其治疗效果。为了克服这些限制,研究者们开始探索 人工智能(AI)驱动的抗体设计。早期的方法多基于 能量计算(energy-based computational methods),如 基于统计能量函数的计算方法(Li et al., 2014; Adolf-Bryfogle et al., 2018),但其表达能力有限,难以满足复杂的抗体设计需求。随后,基于序列的语言模型(Liu et al., 2020; Saka et al., 2021; Akbar et al., 2022; Shin et al., 2021; Jing et al., 2020; Cao et al., 2021)开始应用于抗体设计,但由于 缺乏结构信息,其性能仍然不理想。
最近,共设计方法(co-design methods) 逐渐兴起,这些方法能够同时设计蛋白质的序列和结构(Anishchenko et al., 2021; Wang et al., 2022; Anand & Achim, 2022; Shi et al., 2023),为 AI 驱动的抗体设计提供了新的可能性(Jin et al., 2021; 2022; Luo et al., 2022; Kong et al., 2023a; b; Wu & Li, 2024)。然而,现有共设计方法仍然面临重大挑战,主要包括:
- 依赖已知抗体-抗原复合体的实验结构,即许多方法需要已测定的抗体-抗原结构进行修饰或优化,而 在新抗原场景下,这些数据往往不可用。
- 需要已知的抗体框架区(FRs)结构或来自数据库的模板,然而在 de novo 设计(从头设计)时,框架区的结构通常是未知的,这限制了方法的通用性。
为了解决上述问题,我们提出了 IgGM,一个用于 同时设计抗体序列和结构 的生成模型。IgGM 采用多级网络架构,首先利用 预训练蛋白语言模型 提取抗体序列的进化特征,然后通过 特征编码器 学习抗原-抗体的相互作用,最后由 预测模块 生成抗体的序列和完整三维结构。IgGM 的关键特性是它能够 利用序列和结构的相互作用,优化抗体设计,即使框架区(FRs)仅部分已知,也能生成合理的抗体结构。这种能力符合实际应用场景,并为抗体设计提供了新的可能性。
IgGM 具有强大的适应性,支持多种抗体设计任务,包括:
- 预测抗原-抗体复合体结构。
- 设计 CDR H3 关键结合区域,以及 同时设计多个 CDR 片段(CDR H1-H3 & CDR L1-L3)。
- 无需重新训练即可适应不同应用需求,例如 de novo 设计完整抗体或纳米抗体(nanobodies)。
- 适用于纳米抗体(VHH)设计,纳米抗体是一种小型单域抗体,具有强抗原结合能力和高稳定性(Cai et al., 2020)。
实验结果表明,IgGM 在多个抗体设计任务中表现出色:
- 在 抗原-抗体复合体结构预测任务 上,IgGM 的性能可与现有最先进的方法(如 AlphaFold3)媲美。
- 在 抗体 de novo 设计任务 上,IgGM 优于现有方法(如 dyMEAN、DiffAb),并在 序列恢复率(AAR) 和 DockQ 结构精准度 等关键指标上表现更优。
综上,IgGM 通过结合 序列建模、结构预测和生成建模,突破了传统抗体设计方法的局限性,使其 更符合真实生物制药需求,并为抗体和纳米抗体的计算机辅助设计提供了强大的工具。
Background
Preliminaries
由于纳米抗体(nanobody)可以被视为仅由抗体重链组成的单链抗体,因此在接下来的讨论中,我们将主要以抗体(antibody)为例进行说明。蛋白质由 20 种不同的氨基酸 组成,对于长度为 NNN 的蛋白质序列,可以表示为 $S = {s_i}{i=1}^{N}$,其中每个 sis_isi 代表一个氨基酸残基。蛋白质的三维结构可以用骨架原子(backbone atoms)的 三维坐标 来表示,定义为 $X = {x{i, \omega}}{i=1}^{N}$,其中每个坐标 $x{i, \omega} \in \mathbb{R}^3$,$\omega \in {C_{\alpha}, N, C, O}$ 代表蛋白质骨架中的不同原子。
抗体是一类特殊的蛋白质,由两条不同的链组成,而 纳米抗体 仅包含 一条重链。每条抗体链由 四个框架区(FRs) 和 三个互补决定区(CDRs) 组成。CDRs 可进一步细分为:
- CDR H1, H2, H3(位于重链)
- CDR L1, L2, L3(位于轻链)
抗原-抗体复合体可以表示为:
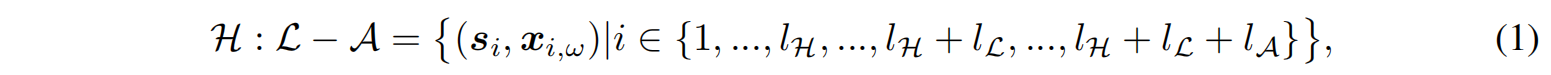
其中,$H : L - A$ 代表抗体的重链(H)、轻链(L)和抗原(A),而 $l_H$、$l_L$、$l_A$ 分别表示 重链、轻链和抗原的序列长度。对于 纳米抗体-抗原复合体,由于纳米抗体 不包含轻链(L),因此相应的复合体表示中不包括轻链部分。
过去的研究通常将 抗体设计问题 定义为:在已知框架区(FRs)序列的情况下,设计 CDRs 以生成能够结合特定抗原的抗体(Shin et al., 2021; Akbar et al., 2022)。由于框架区对抗原-抗体相互作用的影响相对较小,早期研究主要关注 CDR 设计,通常假设 框架区的结构是固定不变的。然而,在面对 全新的抗原 时,所设计抗体的整体结构(包括框架区)是未知的,无法提前确定。IgGM 允许在 抗原结合过程中考虑框架区的结构变化,使其能够在 无实验结构 的情况下进行完整抗体的设计。此外,由于框架区主要提供结构支持,并且某些已知框架区序列在物理化学性质上具有较好的稳定性(Bennett et al., 2024; Vincke et al., 2009),因此在设计时 没有必要完全重新生成框架区序列,而是主要 针对 CDR 进行设计。
Problem Formulation
在实际的抗体设计任务中,现有研究通常使用已知的 框架区序列(FRs) 来引导抗体设计(Bennett et al., 2024)。然而,在没有 互补决定区(CDRs) 的情况下,抗体的结构并不是固定的。因此,我们的研究聚焦于更贴近真实应用场景的问题,即 在给定框架区序列的情况下,设计能够结合特定抗原表位(epitope)的位置的抗体序列和结构。
为了更清晰地表示设计任务,我们将 CDRs 残基集 与 框架区残基集 进行区分:
-
CDRs 残基集:
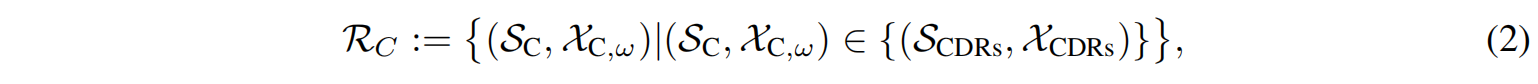
-
框架区(FRs)残基集:
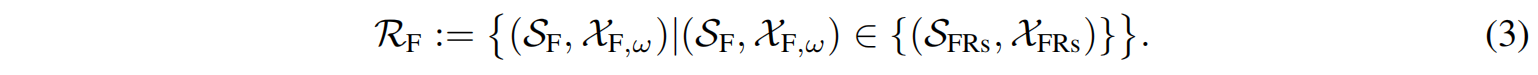
最终,抗体设计问题可以简化为 在已知抗原结构 $R_A = (S_A, X_A, \omega)$ 和已知抗体框架区序列($S_F$)的情况下,设计 CDR 序列 SCS_CSC 以及完整抗体的结构 $(X_C, X_F)$,以确保抗体能够有效结合抗原。
Methods
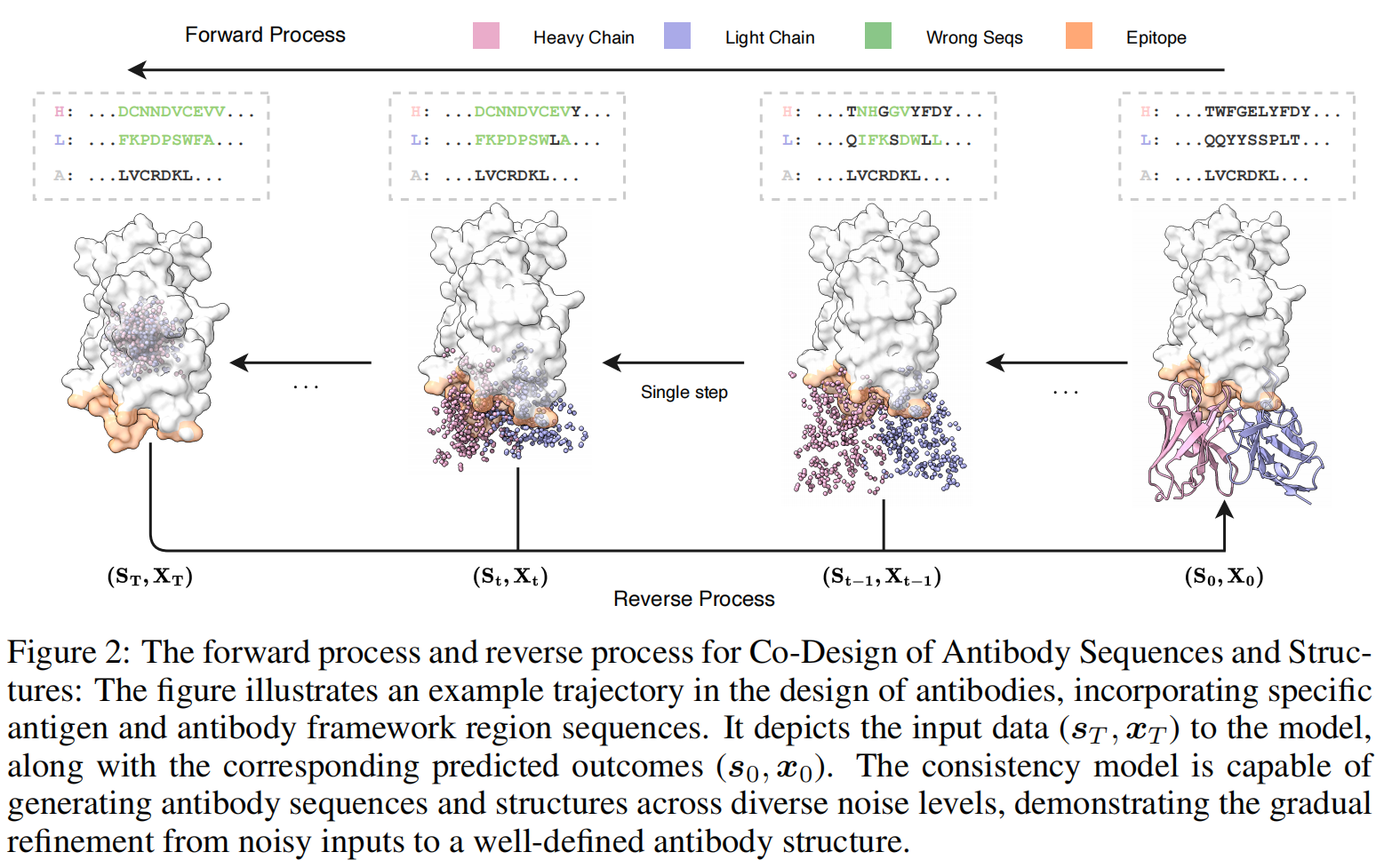
Denoising Network Architecture
IgGM 是一个灵活的抗体设计模型,能够生成能够结合特定抗原的抗体。该模型支持多种抗体设计任务,包括 抗体序列和结构的联合设计。在本节中,我们首先介绍 IgGM 的去噪网络架构(denoising network architecture),然后描述其训练方法和目标,最后介绍 IgGM 采样策略。
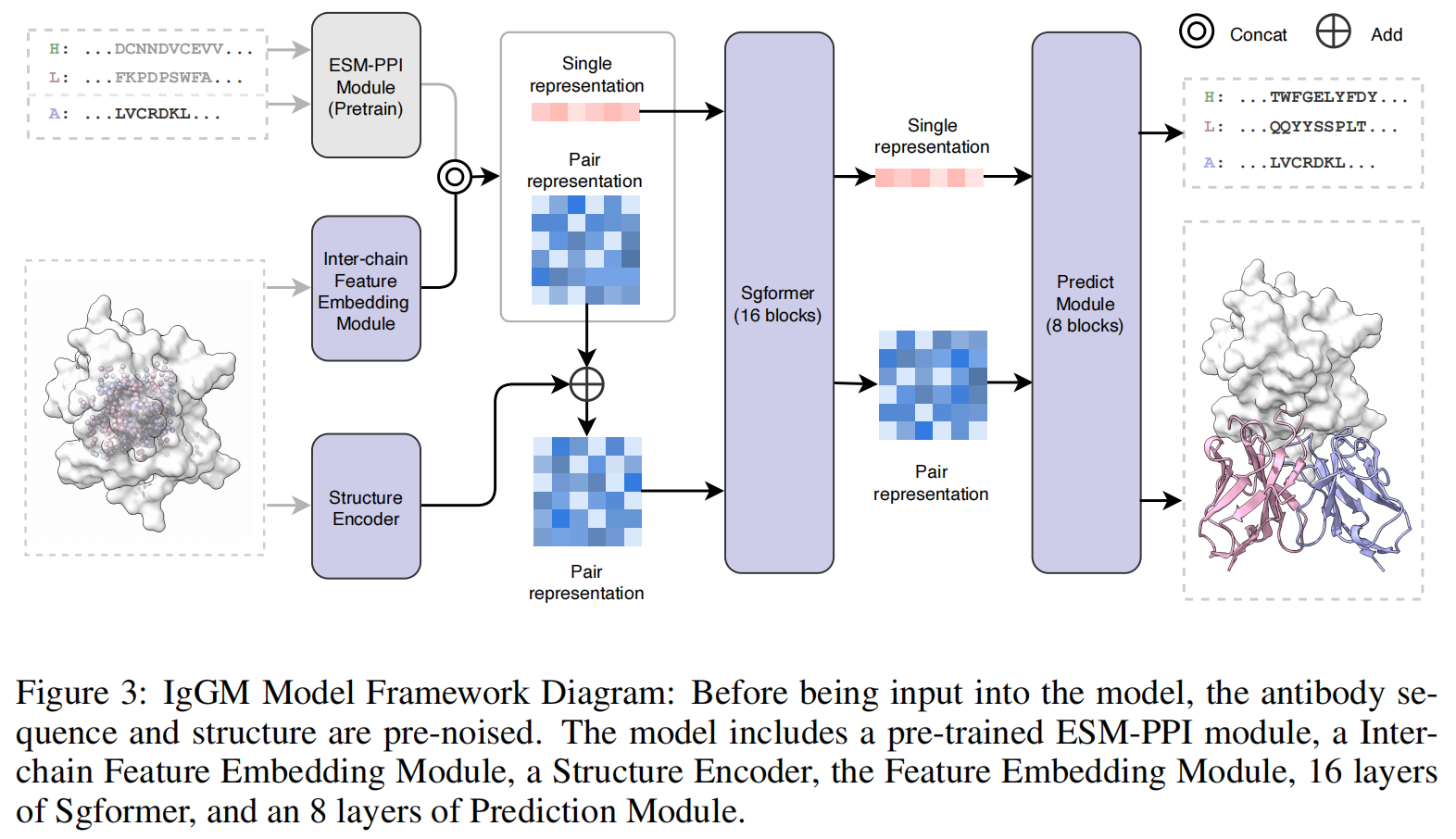
IgGM 的整体网络架构如 Figure 3 所示,包括 预训练蛋白语言模型、特征学习模块和序列-结构设计模块。在输入抗原结构和初始化的抗体序列后,预训练的蛋白质语言模型首先提取序列特征,随后特征编码器(Sgformer)融合这些特征,最终由 序列-结构设计模块(预测模块) 生成抗体序列和结构,并确保其能有效结合抗原。
Feature Extraction from Protein Language Models
受到自然语言处理领域预训练语言模型成功的启发,我们采用 预训练蛋白语言模型(PLM) 作为特征提取器(Lin et al., 2022; Chen et al., 2023; Hayes et al., 2024)。我们选择 ESM-PPI(Wu et al., 2024)作为序列特征提取器,因为它能 有效建模蛋白-蛋白相互作用(PPI)。ESM-PPI 是 ESM2(Lin et al., 2022) 的扩展版本,针对多链蛋白复合体的结构和功能特征进行了优化。抗原和扰动后的抗体数据首先通过 PLM 进行处理,提取最终层的特征,并将其输入 特征编码器。在训练过程中,我们 冻结 PLM 的参数 以保持特征的完整性并减少计算成本。
Multi-level Feature Encoder
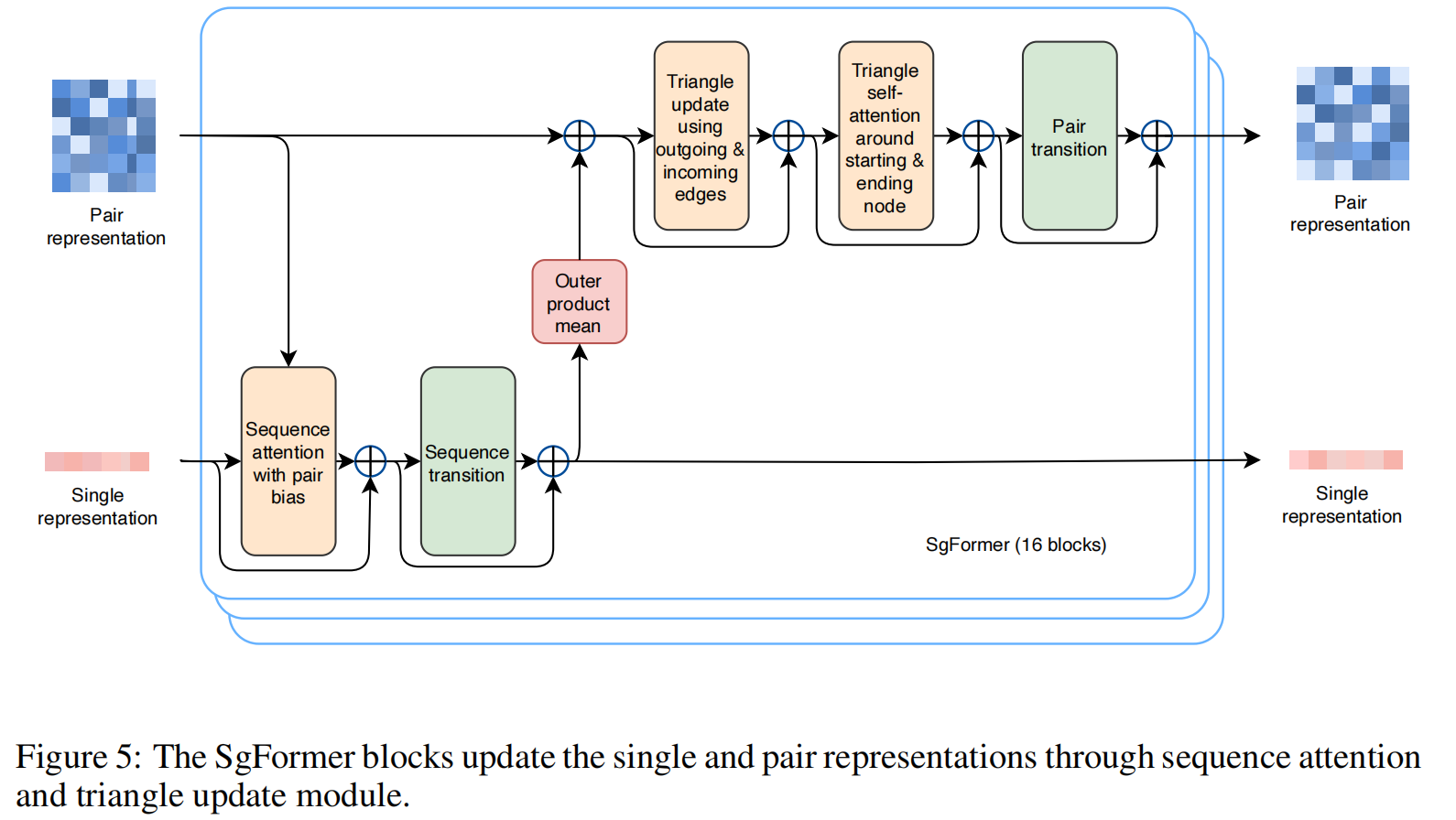
为了充分利用不同特征之间的相互作用,我们设计了 多级特征编码器(multi-level feature encoder),如 Figure 3 所示。该编码器能够建模抗体结构的 不同链之间的相互作用,并通过 在 PLM 输出特征中引入链特定表示(chain-specific representations) 来增强模型的表达能力。此外,我们特别考虑了 抗原表位(epitope) 信息,在抗原特征中增加了专门的表示,以强化抗体-抗原相互作用的学习。该编码器还包含一个 结构编码器(structure encoder),用于捕捉氨基酸的空间位置。这些特征随后被输入 16 层 Sgformer 进行进一步融合和编码,提取出的序列特征对于抗体的去噪恢复至关重要,而 配对(pair-wise)表示 则用于建模抗原-抗体复合体的复杂折叠结构。
Sequence and Structure Design Module
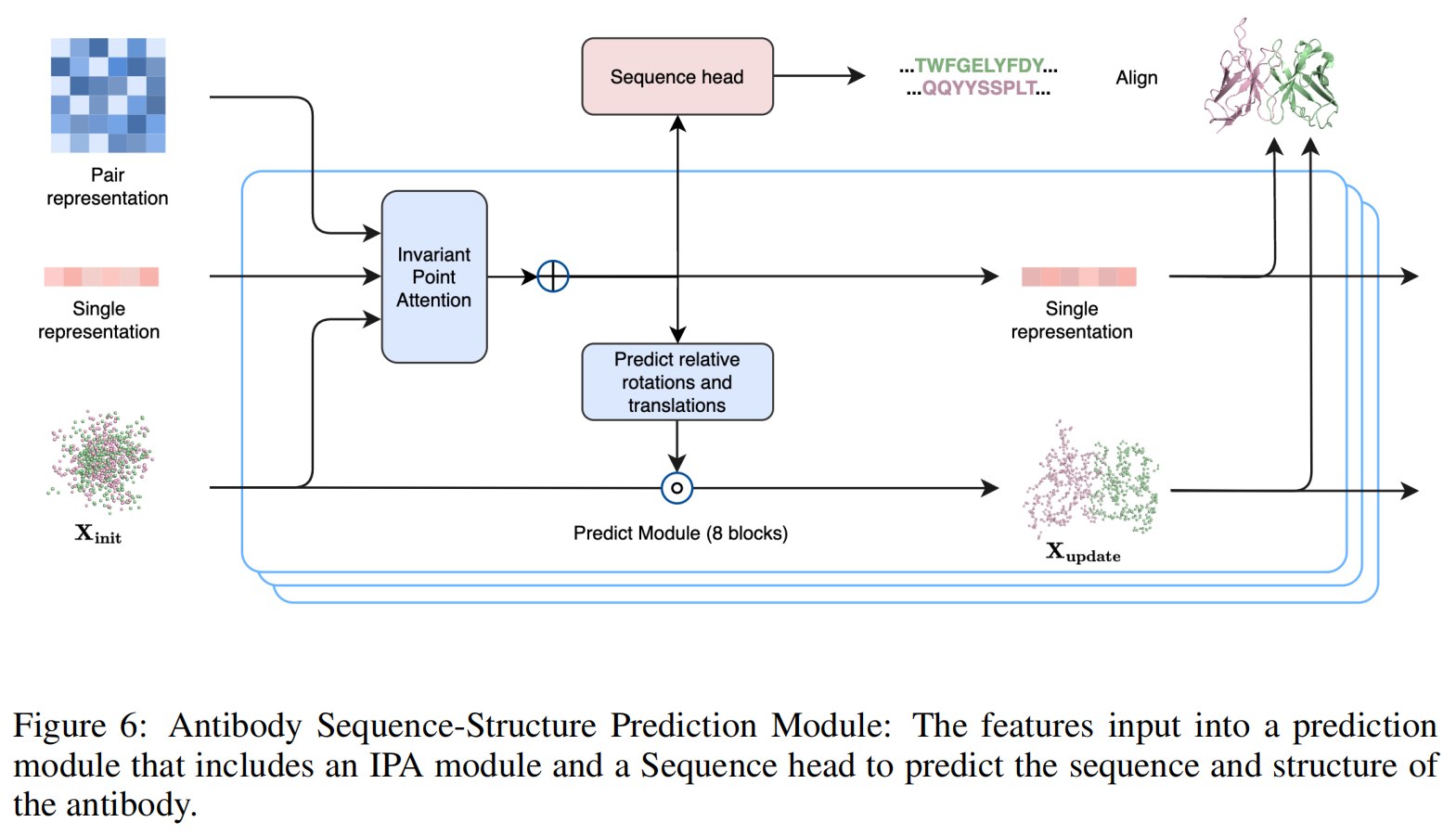
IgGM 使用 8 层预测模块(Predict Modules) 进行结构优化和序列生成(见 Figure 6)。该模块采用 不变点注意力(Invariant Point Attention, IPA) 进行结构优化,并同时输出设计的抗体序列。由于 IPA 具有旋转和平移不变性,因此能够确保抗体结构在不同空间位置和取向下的一致性。预测模块利用 Sgformer 学习的序列和配对特征,同时结合从初始采样生成的抗体结构信息,迭代优化氨基酸坐标,最终得到完整的 抗体三维结构。
Inter-chain Feature Embedding Module and Structure Encoder
IgGM 采用两个核心组件来建模不同抗体链的特征及其与抗原的相互作用,如 Figure 3 所示:
- Inter-chain Feature Embedding Module:通过融合氨基酸的 位置信息 和 跨链信息,捕获抗体不同链的相对位置关系,并提取 链特定特征(chain-specific features)。
- Structure Encoder:编码蛋白质结构信息,利用距离特征计算氨基酸之间的空间关系,并通过 离散化处理 生成可输入的特征。为了充分利用 抗原表位信息,我们将其编码为 单体表示(Single Representation) 和 配对表示(Pair Representation),以增强对 表位附近结构的生成能力。
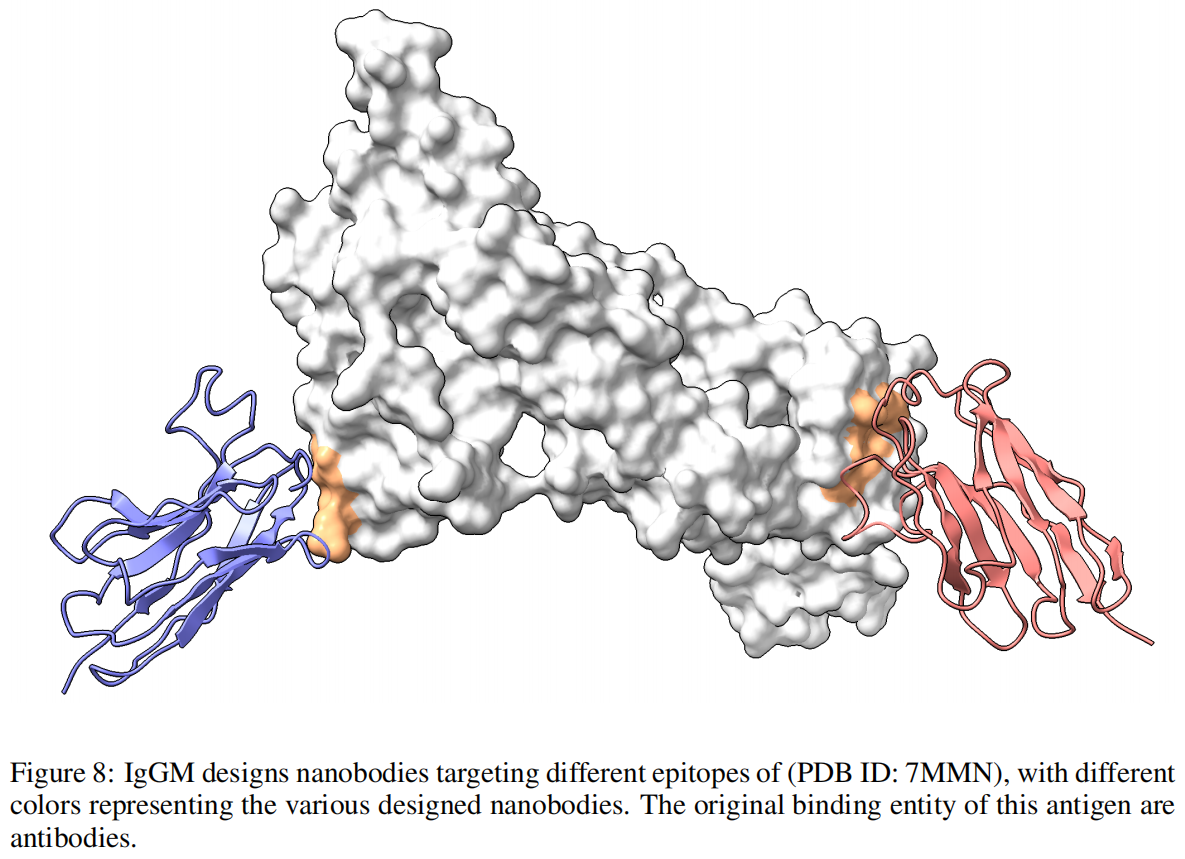
如 Figure 8 所示,IgGM 可生成能够特异性结合不同表位的抗体。
Training Details
我们采用 蒸馏(distillation)方法 训练 一致性模型(consistency model),该训练分为 两个阶段。首先,我们预训练 扩散模型(diffusion model),然后在第二阶段进行蒸馏,以获得最终的一致性模型。
Phase 1: 结构训练
在第一阶段,我们训练 结构生成 任务,同时 保持抗体原始序列信息。具体而言,我们 仅训练结构预测任务,以确保模型首先学习 抗体的三维折叠规律。训练时,我们从数据集中 随机采样抗原-抗体复合体 $x$,并对抗体结构施加不同级别的噪声 $x_t$,然后训练模型 D 以恢复原始抗体结构。目标是确保恢复的结构尽可能接近真实结构。
结构预测的总损失函数如下,最小化 几何损失、框架损失、结构违规损失:
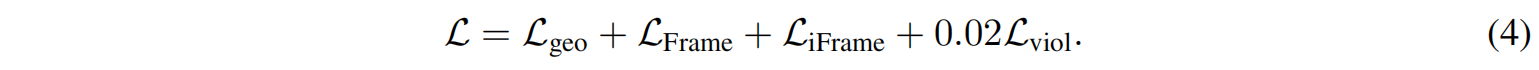
其中:
-
$L_{geo}$ 是几何监督损失(预测氨基酸之间的距离和角度)。
-
$L_{Frame}$ 约束抗体整体结构,$L_{iFrame}$ 进一步扩展到多链抗体的建模。
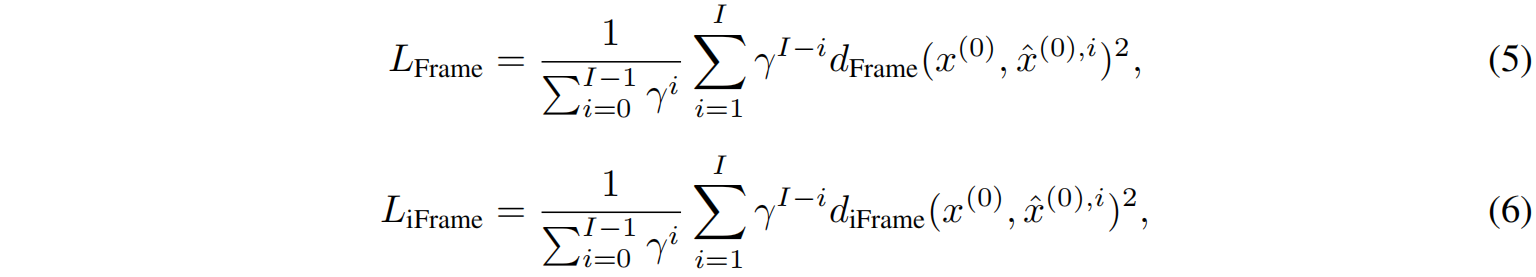
-
$L_{viol}$ 是 结构违规损失(用于修正不合理的键长、键角和空间冲突)。
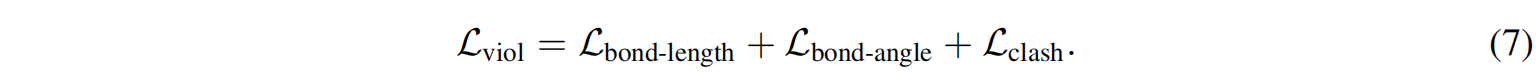
Phase 2: 序列训练
在第二阶段,我们 加入序列设计任务,训练目标变为最小化 序列恢复损失和结构损失:

其中:
- $L_{srcv}$ 是 序列恢复损失,用于训练模型 预测正确的氨基酸类型(采用交叉熵损失)。
- 其他损失项与第一阶段相同。
训练过程中,我们采用 混合策略,赋予不同任务不同的采样概率(例如 CDR H3 设计的权重比其他 CDR 更高,4 :2: 2: 2用于设计CDR H3、CDR H和所有CDR),以提升模型的泛化能力。
Phase 3: 一致性蒸馏模型
完成扩散模型训练后,我们使用 一致性模型蒸馏(Consistency Model Distillation),采用 Song et al. (2023) 提出的损失,减少去噪步骤(从 1000+ 步减少到 1-10 步):
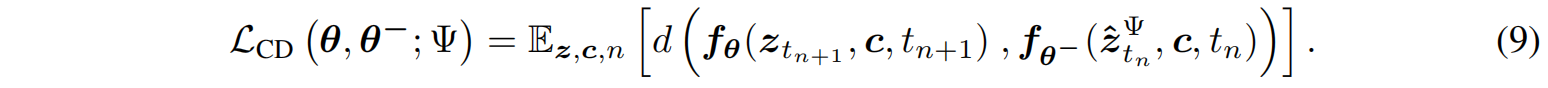
其中,$ẑ_{\Psi t_n}$ 是 从 $t_{n+1}$ 到 $t_n$ 的估计值,用于一致性模型的优化。
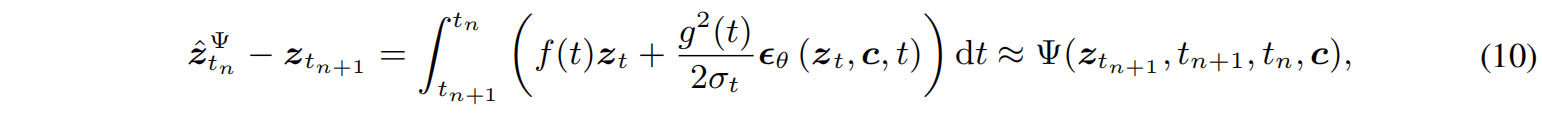
Directly Generate Antibodies

如 Algorithm 1 所示,在设计特定抗原的抗体时,我们首先 随机初始化氨基酸,然后采样 位移坐标(translation coordinates) 和 旋转矩阵(SO(3)),最终通过训练好的 IgGM 生成抗体序列和结构。由于 一致性模型 具有 高效采样能力,IgGM 既可以 单步生成抗体,也可以 采用多步优化以提高稳定性
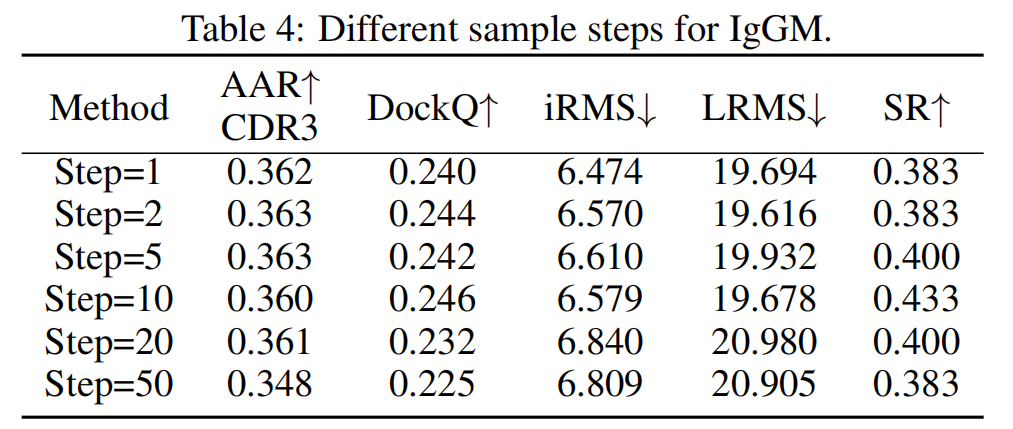
实验结果表明,10 步采样在生成质量与速度之间取得了较好平衡(见 Table 4)。此外,我们可以使用 AlphaFold3 预测的结构作为初始输入,进一步提高 IgGM 的生成质量。
Experiments
从SAbDab数据库中构建了训练、验证和测试集,使用了广泛使用的基于时间划分数据集的方法。我们从2023年下半年删除了与训练集序列相似的抗体,得到了一个包含60个抗体(SAb-23H2-Ab)和27个纳米体测试集(SAb-23H2-Nano)的测试集
由于AlphaFold 3的局限性,为每个示例生成5个样本
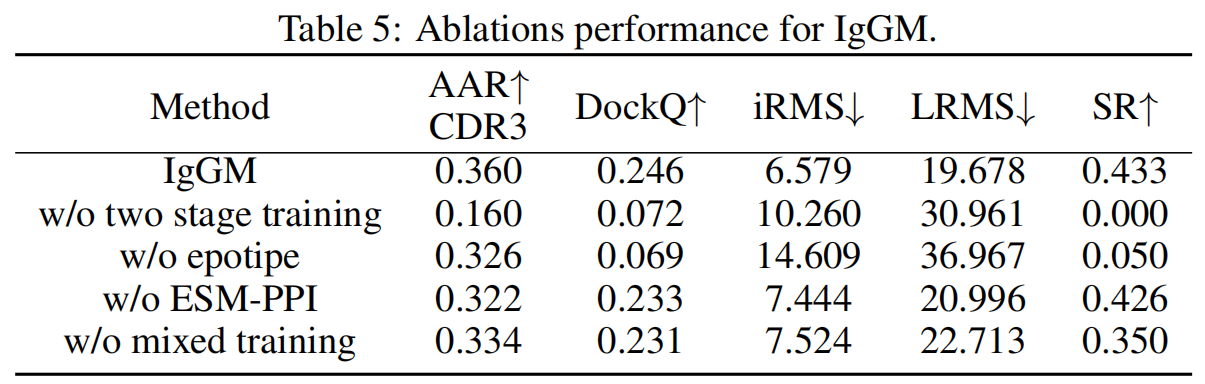
Complex Structure Prediction
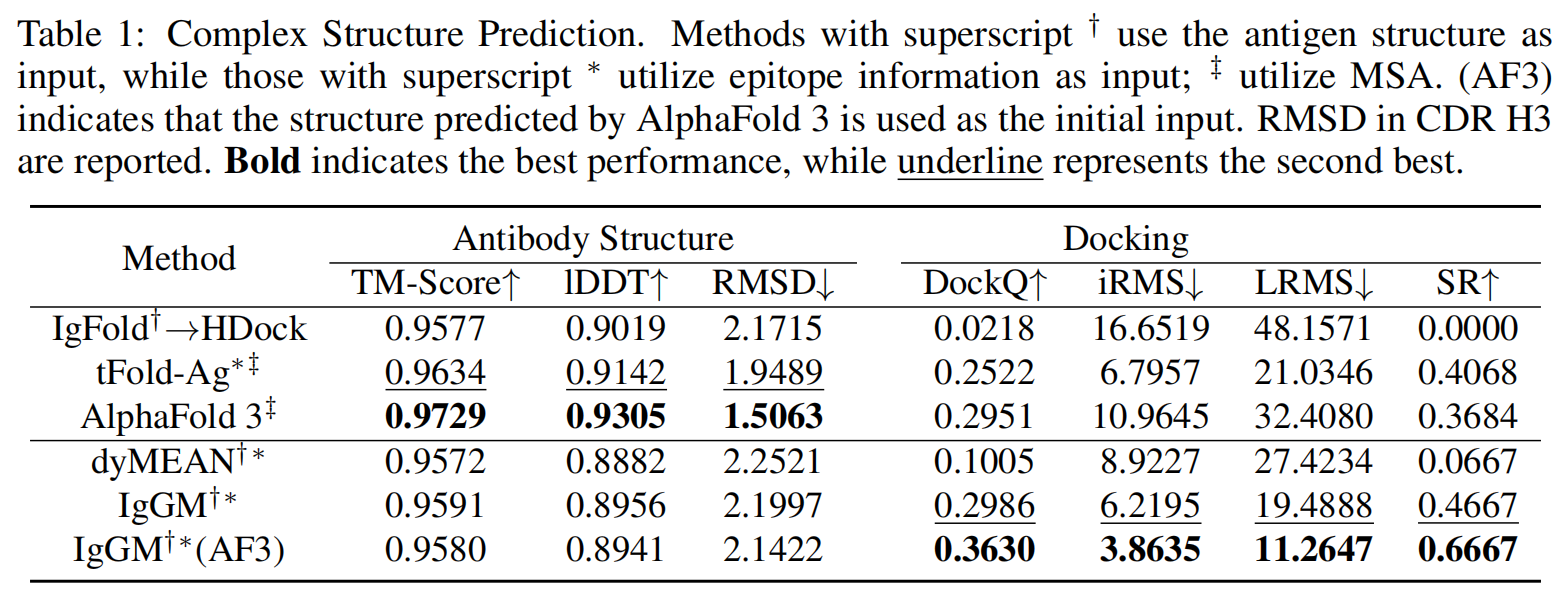
复合物结构预测(Complex Structure Prediction)任务的目标是,在已知抗体序列和抗原的情况下,预测抗体-抗原复合体的三维结构。IgGM 能够在不进行序列设计的前提下完成结构预测。为了与现有方法进行公平比较,我们采用 tFold-Ag 作为基准评测标准,并使用 TM-Score、DockQ、成功率(SR, DockQ > 0.23) 作为评测指标。
在实验中,我们在 SAb23H2 测试集上评估了 IgGM,并与以下四种方法进行对比:
- IgFold+ HDoc):仅支持抗体结构预测,我们用 HDock 对抗体进行对接。
- tFold-Ag:一种基于 AlphaFold 变体的抗体-抗原复合体预测方法。
- AlphaFold3 :最新的通用蛋白质结构预测工具,直接用于复合体预测。
- dyMEAN:一种利用模板初始化的抗体设计方法。
实验结果(见 Table 1)表明,在 抗体结构预测 任务中,IgGM 的性能优于 dyMEAN,尽管相比专门的结构预测方法仍存在一定差距,但整体预测质量接近 SOTA(State-of-the-Art)。在 对接质量(DockQ) 方面,IgGM 超过了所有对比方法,显示其在 抗体-抗原相互作用建模 方面的能力。此外,IgGM 预测的抗体 iRMS 和 LRMS 更低,其 DockQ 成功率达到 46.67%,远超 dyMEAN(6.67%)。如果使用 AlphaFold 3 预测的抗体结构作为初始输入,IgGM 的性能进一步提升,DockQ 成功率提高至 66.67%,表明该策略能够增强结构预测质量。
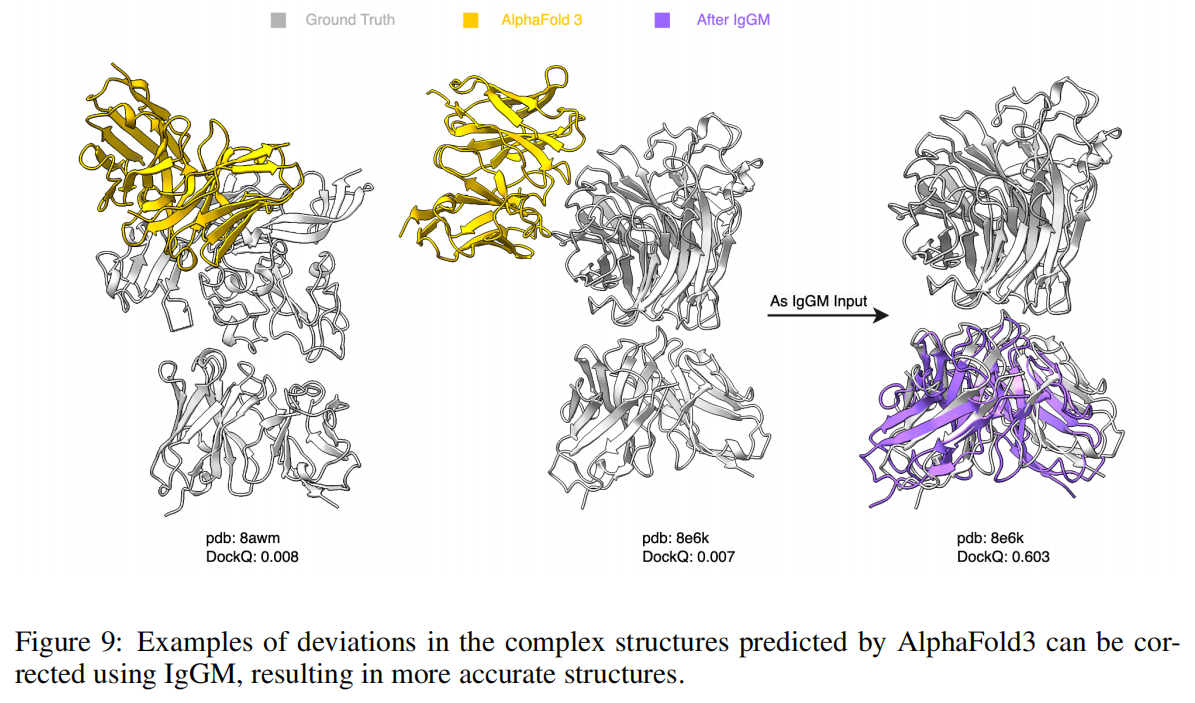
Figure 9 进一步展示了 IgGM 在 修正 AlphaFold3 预测错误的复合物结构 方面的能力,使其生成更符合表位的结构。
De Novo Design of Antibodies for Specific Antigen
在 从头(de novo)设计抗体 任务中,我们评估了 IgGM 在 生成能结合特定抗原的抗体序列和结构 方面的能力。由于现有方法难以实现端到端的抗体设计,我们采用 两种 pipeline 进行对比:
- Pipeline 1(dyMEAN 流程):先用 IgFold 预测抗体结构 → 用 HDock 进行抗原对接 → 采用 CDR 生成方法(MEAN、DiffAb、dyMEAN) 设计 CDR → 进行侧链优化。
- Pipeline 2(AlphaFold 3 流程):使用 AlphaFold 3 直接预测抗体-抗原复合体结构 → 进行抗原结构对齐 → 采用 CDR 生成方法 进行设计。
我们在 SAb-2023H2-Ab 测试集上对比了 MEAN、DiffAb、dyMEAN 和 IgGM。实验结果(见 Table 2)显示:
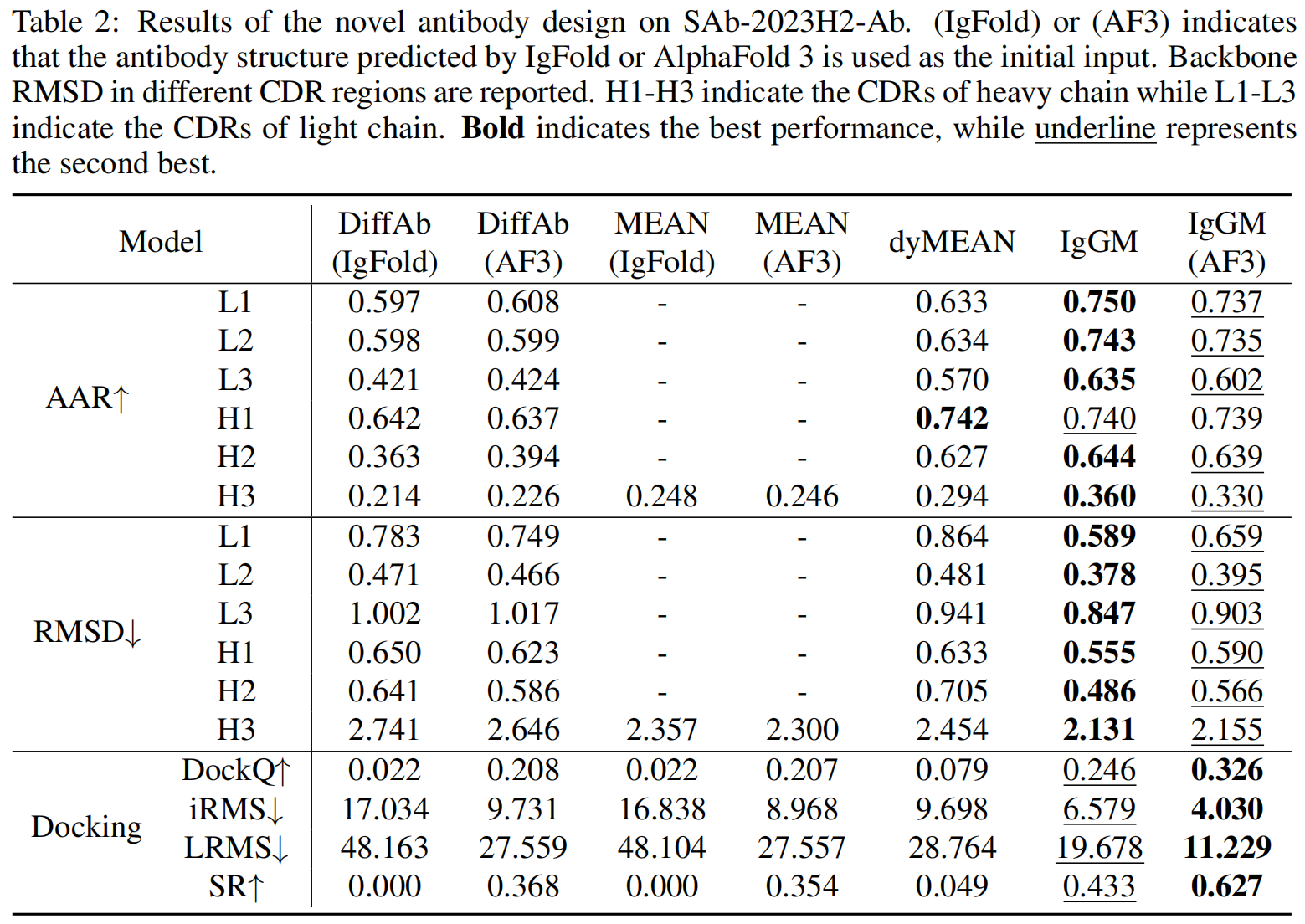
- IgGM 在几乎所有评测指标上均优于其他方法,包括 DockQ、iRMS、LRMS、CDR 结构准确性和序列恢复率(AAR)。
- 仅支持 CDR H3 设计 的 MEAN 表现较弱,而 dyMEAN 和 DiffAb 由于不支持整体结构设计,其生成的抗体结构质量较低。
- IgGM 是唯一在 iRMS < 8、LRMS < 20 的方法,同时成功率(SR)高达 43.3%,表明其 对抗体整体结构和抗原结合界面的优化能力。
- IgGM 的 CDR-H3 序列恢复率(AAR)达到 36%,比 dyMEAN 提高 22.4%,进一步验证其 抗体序列生成的可靠性。
- 使用 AlphaFold 3 作为初始结构输入,IgGM 在 DockQ 相关指标上进一步提升 近 20%,表明强结构预测工具的结合能增强生成效果。
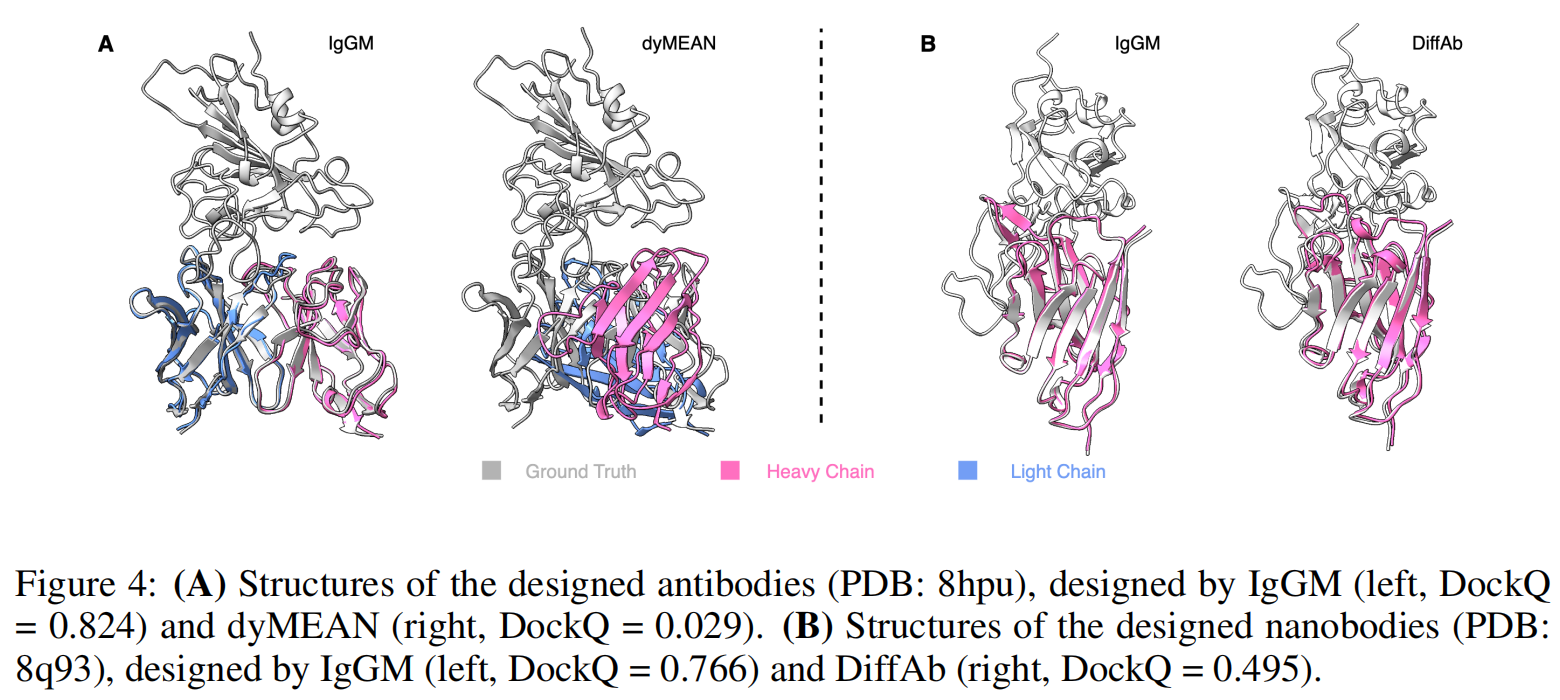
Figure 4(A) 展示了 IgGM 生成的抗体相比 dyMEAN 更贴合抗原表位,DockQ 分数更高。
Structure Prediction and De Novo Design of Nanobodies
纳米抗体(nanobody, VHH) 是 单链抗体,相比传统抗体,CDR H3 更长,且结合模式更加灵活,因此在结构预测和生成任务上更具挑战性。我们使用 SAb-2023H2-Nano 数据集,比较 IgGM 和 DiffAb(AF3 结构初始化) 在 纳米抗体结构预测和 de novo 设计 任务上的性能。
实验结果(见 Table 3)表明:
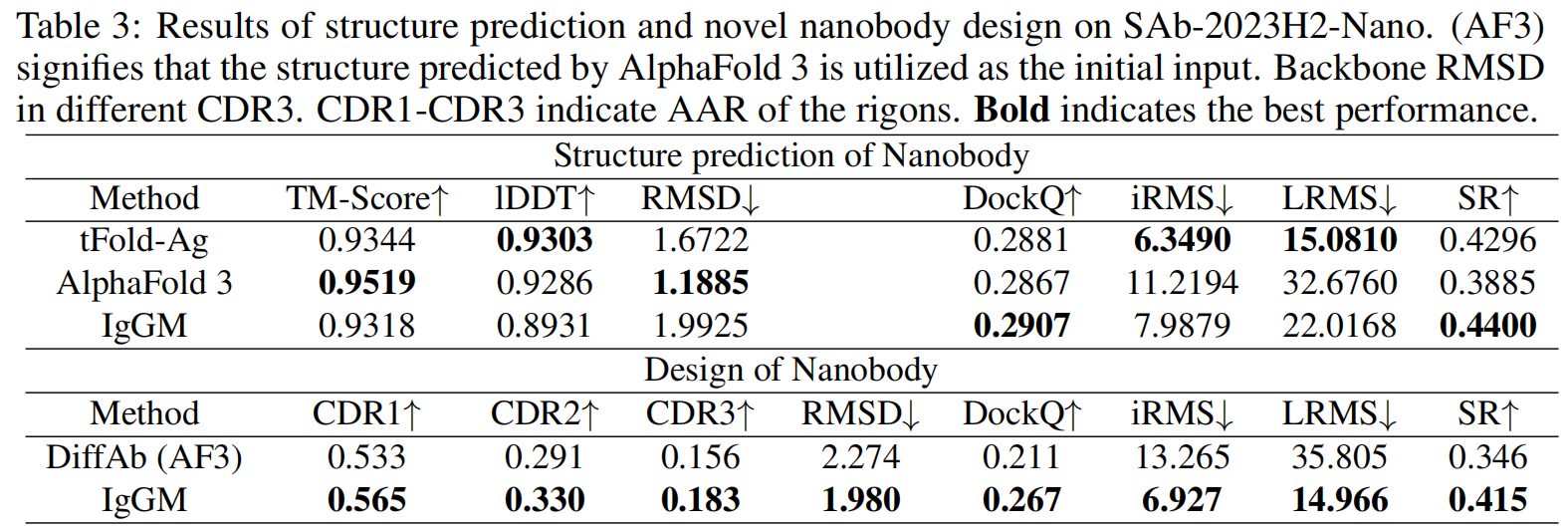
- 纳米抗体的结构预测任务 由于其较为简单的单链特性,整体结果比传统抗体更好。
- 然而,在对接相关指标上,纳米抗体的 DockQ 和成功率低于传统抗体,表明其更复杂的结合模式对预测提出了更高要求。
- IgGM 在 DockQ 和成功率(SR = 44%)上均优于 DiffAb,证明其在 纳米抗体-抗原复合体建模 方面的能力。
- 在 de novo 设计任务 上,IgGM 仍然优于 DiffAb,虽然 CDR 序列恢复率有所下降(由于纳米抗体的 CDR 变异性较大),但生成的结构更加精确,最终 DockQ 成功率提升至 41.5%。
Figure 4(B) 展示了 IgGM 生成的纳米抗体相比 DiffAb 具有更高的 DockQ 分数,更贴合抗原表位。


