论文地址:Pre-training Antibody Language Models for Antigen-Specific Computational Antibody Design
ABGNN:预训练AbBERT+图网络RefineGNN编码抗体抗原
Abstract
抗体是一种能够通过结合病原体来有效保护人体的蛋白质。近年来,基于深度学习的计算抗体设计因其能够自动从数据中挖掘抗体模式,以补充人类经验而受到广泛关注。然而,计算方法严重依赖高质量的抗体结构数据,而这类数据非常有限。此外,决定抗体特异性和结合亲和力的关键部分——互补决定区(CDR)高度可变且难以预测,有限的高质量抗体结构数据的可用性进一步加剧了CDR生成的困难。幸运的是,已有大量抗体序列数据可用于辅助CDR建模并减少对结构数据的依赖。受预训练模型在蛋白质建模方面成功应用的启发,本文开发了一种抗体预训练语言模型,并将其系统性地融入抗原特异性的抗体设计模型中。具体而言,首先基于序列数据预训练了一个新型抗体语言模型,然后提出了一种CDR序列与结构的一次性生成方法,以减少自回归方法的高计算成本和误差传播问题,最后通过精心设计的模块利用预训练抗体模型来增强抗原特异性的抗体生成模型。实验结果表明,我们的方法在序列与结构生成、针对抗原结合的CDR-H3设计以及抗体优化等任务上优于现有基线模型。
1 Introduction
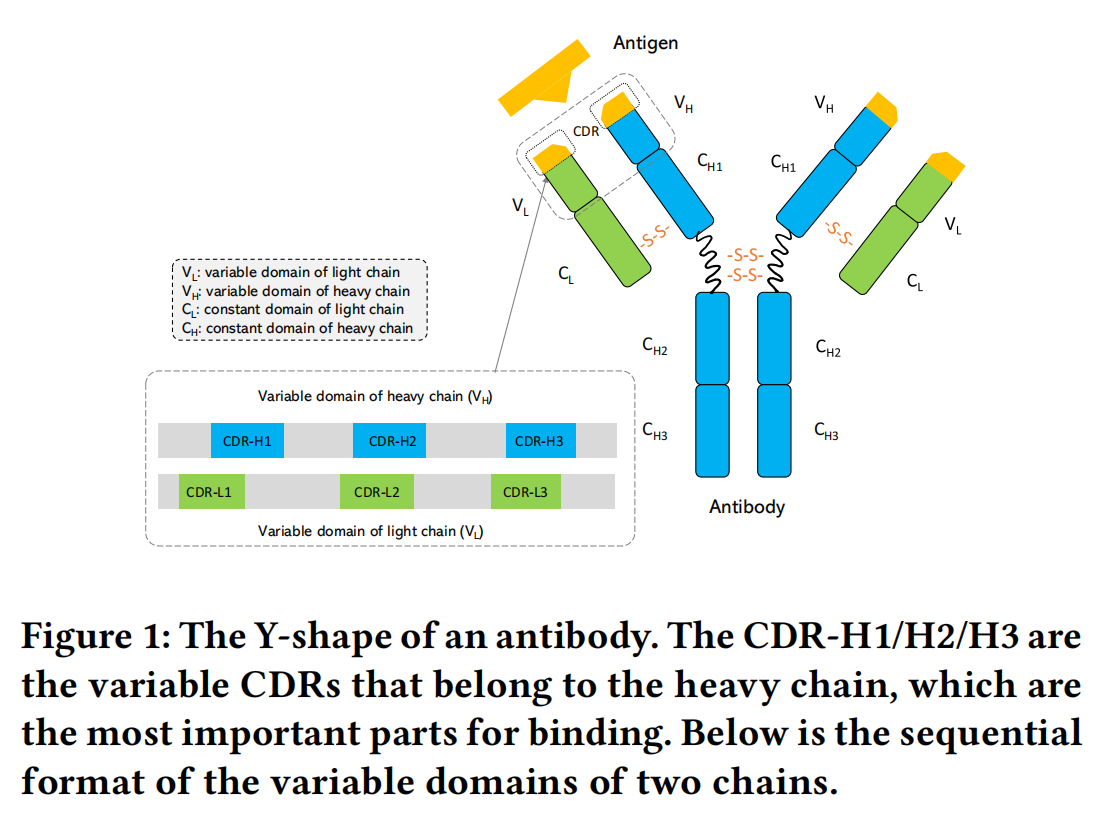
抗体是Y形蛋白质,在人类免疫系统中发挥关键作用,可用于治疗各种病原体感染、癌症等疾病。抗体具有高度特异性,大多数抗体是单克隆抗体,每种抗体通常仅与特定的抗原结合。抗体的结合特异性主要由互补决定区(CDR)决定,尤其是重链上的CDR-H3,它是最关键且变异性最高的区域。因此,抗体设计的主要挑战在于识别和设计能够有效且稳定结合特定抗原的新CDR。
近年来,计算方法被探索用于自动生成具有期望特性的CDR序列,例如高结合亲和力。深度学习在抗体设计中展现了巨大潜力,如基于深度生成模型和图神经网络的方法。然而,早期研究主要集中于生成CDR序列,而近年来,序列与三维结构的联合设计成为更具前景的趋势。例如,一些研究使用深度图神经网络同时建模抗体的序列和结构。然而,目前的方法仍然面临多个挑战。首先,现有数据集中抗体的三维结构数量有限,例如最广泛使用的抗体结构数据库 SAbDab 仅包含数千个结构,而抗原-抗体复合物的数据更为稀少,这给深度学习模型的训练带来了挑战,尤其是对于高度可变的CDR结构而言。其次,当前的抗体设计方法大多采用自回归方式逐步生成氨基酸类型及其对应的结构坐标,这种方法存在两个缺点:一是生成效率低下,需要多步迭代;二是存在误差累积问题,早期生成的错误会影响后续步骤的准确性,最终影响整体生成质量。
为了解决上述挑战,本文提出了一些新的策略:
- 尽管结构数据有限,但抗体序列数据规模庞大,而已有研究表明蛋白质的序列决定其结构,因此,特别设计了一种抗体序列预训练方法,以充分利用大量序列数据,提升抗体表示能力并缓解结构数据的稀缺问题。
- 提出了一种 一次性(one-shot) 生成CDR序列和结构的方法,相比于自回归方法,该方法能够同时生成所有CDR氨基酸,并一次性更新其结构,从而避免了自回归方法的误差传播问题。同时,我们还引入多次迭代精炼步骤(类似于AlphaFold2),以进一步优化生成结果。
- 我在抗体设计模型中系统性地融入了预训练抗体语言模型,而不仅仅是将其作为初始化。具体而言,我们使用 prompt tuning 方法来优化预训练模型,使其不仅保留原有能力,还能为下游抗体设计任务提供可迁移的知识。此外,我们将预训练模型的中间表示与抗体设计模型进行融合,以增强CDR序列和结构的预测能力。
通过上述策略,我们在多个任务上验证了所提方法的有效性,包括 序列与结构生成、抗原结合的CDR-H3设计,以及抗体优化任务。实验结果表明,相比于现有方法,我们的方法在多个评测指标上达到了 最优性能,同时提升了生成的效率和准确性。本文的主要贡献总结如下:
- 提出了一种 专门针对抗体的预训练方法,并将其成功应用于抗体序列-结构的联合设计。
- 采用了一种 一次性(one-shot) 生成策略,以提高抗体生成的效率并避免误差累积。
- 通过大量实验验证了所提出方法的有效性,达到了当前最优的抗体设计性能。
3 Methods
3.1 Background and Overview
抗体由两个对称部分组成,每个部分包含重链和轻链(VH 和 VL),其中可变区(Variable Region)包含 框架区(Framework) 和 三条互补决定区(CDR-H1/H2/H3)。CDR 是决定抗体与抗原结合能力的关键区域,其中 CDR-H3 的变异性最高,因此其准确预测是抗体设计的核心挑战。本文的方法基于 深度图神经网络(GNN),并结合 抗体-抗原复合物(Antigen-Antibody Complex) 进行抗体设计。与之前的方法不同,本文 仅考虑抗原的表位(epitope)区域,而不是整个抗原,并将 抗体框架区 作为上下文信息,以提高设计的准确性。
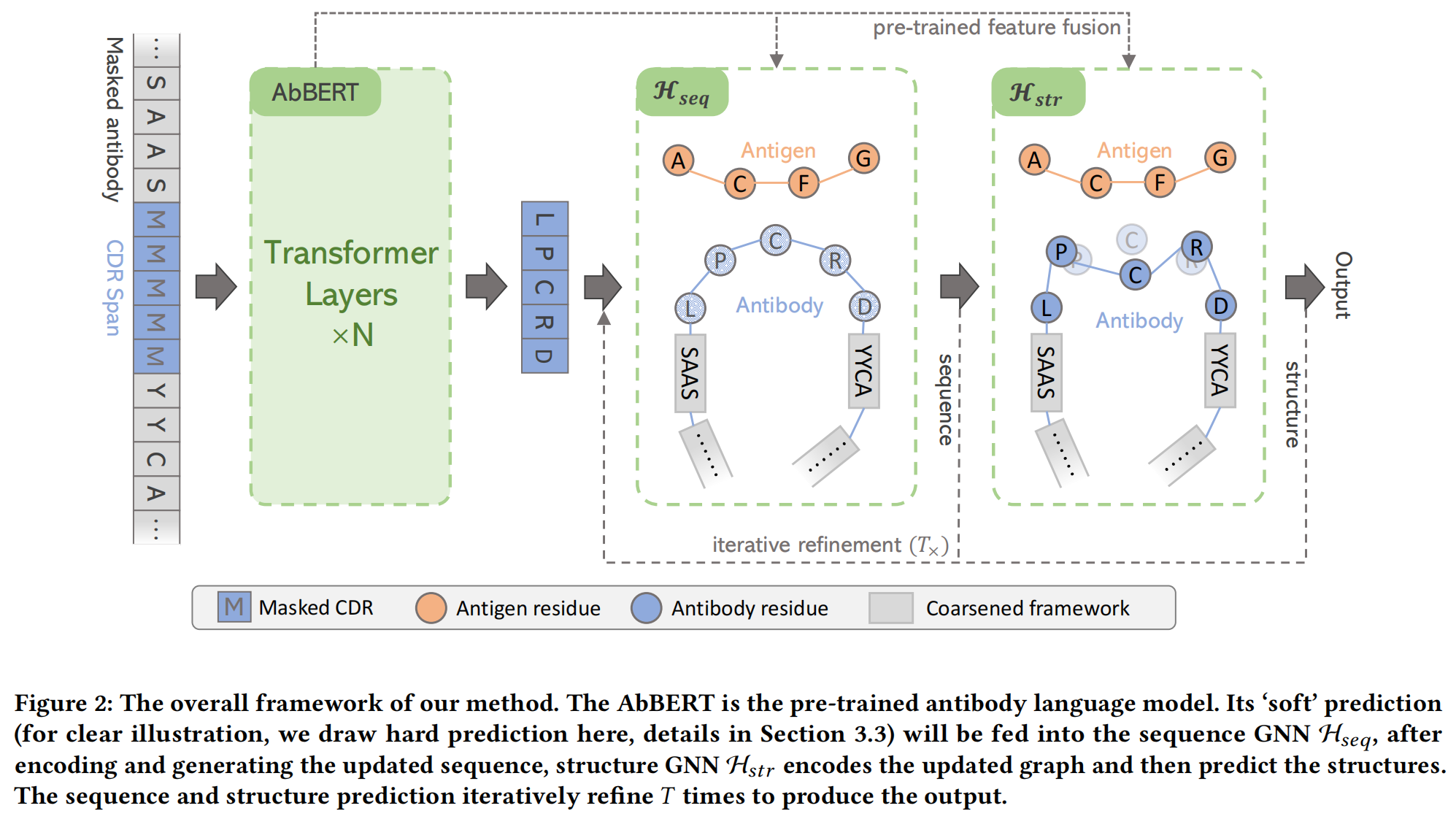
本文的方法采用了 AbBERT 预训练模型 提供的抗体序列表示,并结合 序列 GNN(H_seq)和结构 GNN(H_str) 进行 抗体序列-结构联合生成,最后通过多轮迭代优化最终的生成结果。该方法的整体框架如 Figure 2 所示。
3.2 AbBERT: Antibody Pre-training
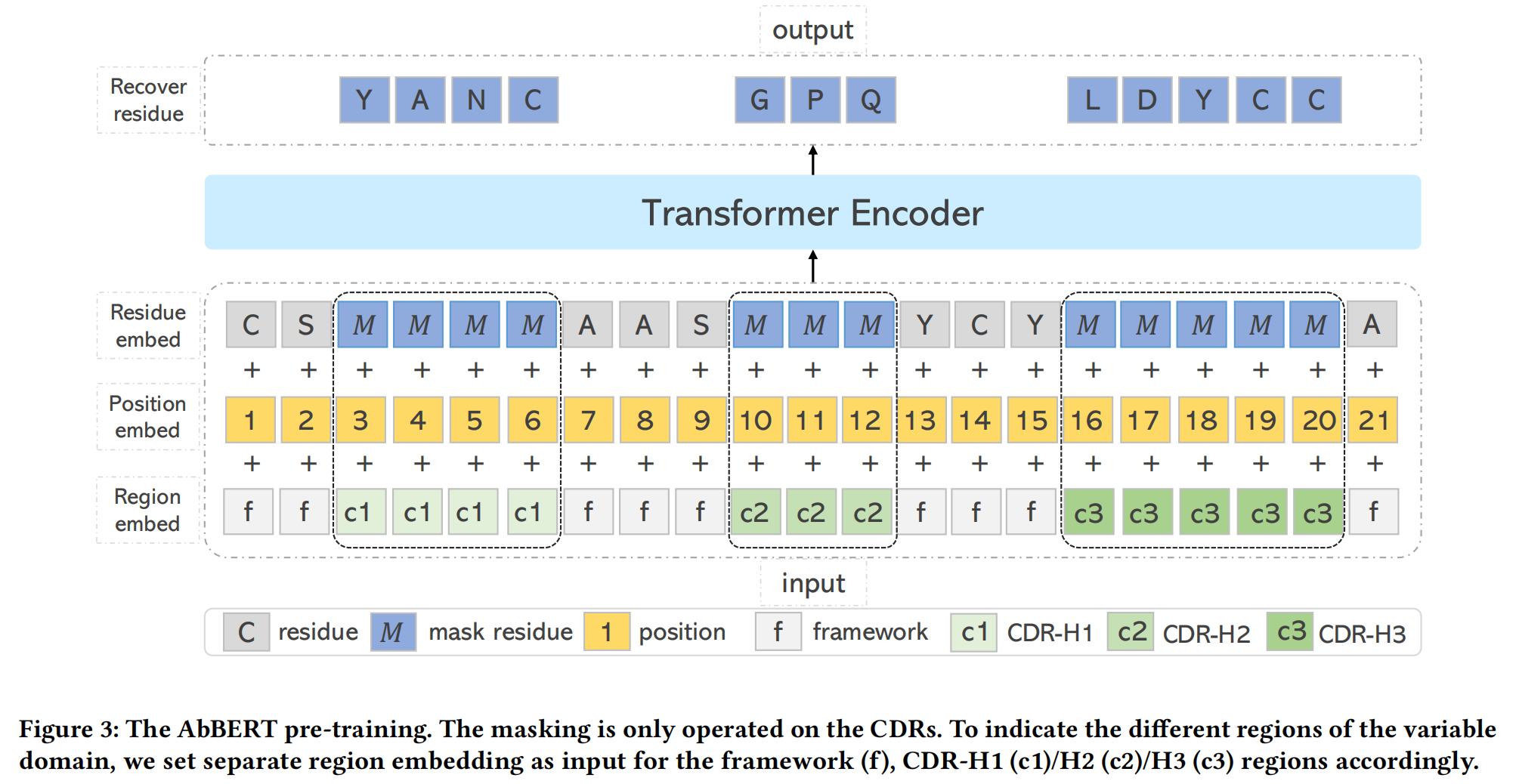
由于高质量的抗体结构数据稀缺,而抗体序列数据量较大,因此本文借鉴蛋白质预训练的成功经验,设计了一种 针对抗体的预训练语言模型 AbBERT,以增强抗体表示能力并提升抗体设计的性能。
AbBERT 预训练采用了类似 BERT 的掩码语言模型(MLM),但针对抗体序列的特殊性进行了优化:
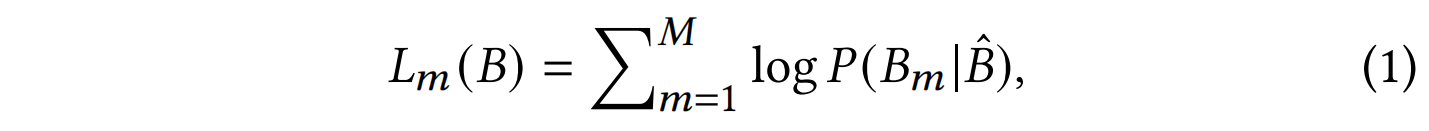
- 仅对 重链(Heavy Chain, VH) 进行训练,因为重链对于抗体功能至关重要。
- 仅对 可变区(Variable Region, VH) 进行预训练,而不包括恒定区(Constant Region)。
- 掩码策略(Masking Strategy):不同于普通 BERT 预训练,AbBERT 仅对 CDR 区域进行掩码,而框架区(Framework)则用于提供上下文信息。这种设计有助于提高 CDR 预测的准确性。
- 区域嵌入(Region Embedding):在输入中为不同的 CDR-H1/H2/H3 及框架区 分别引入独立的嵌入,以帮助模型区分不同区域的功能。
- 掩码比例(Masking Ratio):由于 CDR 区域仅占 VH 序列的 15%,因此使用较高的掩码比例(50%、80% 和 100%)进行实验,以确保模型能够有效学习 CDR 的信息。
训练完成后,AbBERT 可以同时预测所有 CDR 残基(one-shot 预测),提高生成效率,并为后续抗体设计任务提供强大的初始化信息。
3.3 Antibody Sequence-structure Co-design
本文将抗体设计建模为 3D 图补全任务(3D Graph Completion Task),目标是预测 抗原结合区(paratope,即 CDR) 的结构,并生成相应的序列。为此,我们采用 层次化图神经网络(Hierarchical Graph Neural Network, GNN) 进行编码和解码,分别用于序列预测(H_seq)和结构预测(H_str)。所有图的构建均基于 K 近邻(KNN) 计算,节点可以是原子级别或残基级别的。
在本文的方法中,与 Jin et al. [18] 的工作不同,我们不仅考虑了 抗原表位(epitope)和抗体结合区(paratope),还额外加入了 抗体框架区(framework) 作为条件信息,以提高抗体设计的准确性。此外,我们采用 AbBERT 预训练模型 进行 初始化和特征融合,以进一步优化抗体设计的效果。
3.3.1 Encoding
编码部分由 H_seq 和 H_str 共同完成,采用 分层处理(Hierarchical Processing),包括 原子级别(atom-level) 和 残基级别(residue-level) 的特征提取。
- 原子级编码(Atom-level Encoding)
- 主要用于提取抗体和抗原之间的细粒度交互信息。
- 包括 骨架(backbone)原子 和 侧链(side chain)原子 的特征。
- 采用 径向基函数(RBF) 计算原子间的距离,作为 GNN 计算的输入。
- 残基级编码(Residue-level Encoding)
- 主要用于提取 全局结构信息,确保模型能够学习到抗体-抗原的结合模式。
- 每个残基的特征包括:氨基酸类型特征,二面角(Dihedral Angles),极性(Polarity),疏水性(Hydropathy)
- 采用 消息传递网络(MPN) 进行残基特征的传播和学习。
Framework Encoding
框架区在抗体结构中较长,因此采用 粗粒度(Coarse-grained) 编码方式,以减少计算开销:
-
将框架区划分为 残基块(Residue Blocks)。
-
每个残基块的嵌入表示 取其所有残基的平均值。
-
计算公式如下:
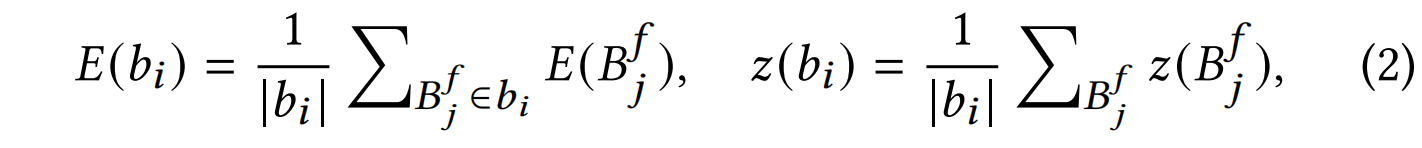
其中,$E(B_f^j)$ 和 $z(B_f^j)$ 分别表示框架区 第 j 个残基的特征和坐标。
Initialization
在抗体设计任务中,由于 CDR 的序列和结构未知,因此必须进行合理的初始化。我们采用 两种不同的初始化策略:
-
序列初始化(Sequence Initialization)
-
以往的方法采用 随机猜测 作为初始序列分布,而我们使用 AbBERT 预训练模型 提供 soft 预测分布 作为初始化信息。
-
具体而言,CDR 的初始状态采用 AbBERT 的 soft 预测分布:
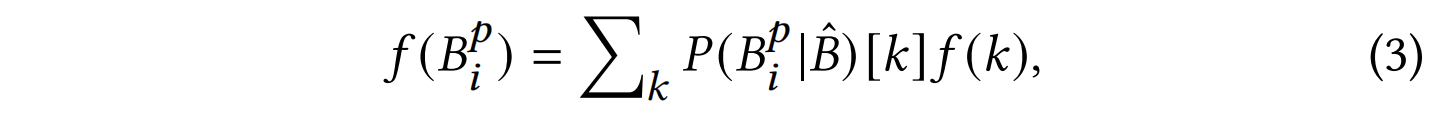
其中,$P(B_p^i | \hat{B})$ 是 AbBERT 预测的氨基酸概率分布,$f(k)$ 是 氨基酸 k 的特征表示。
-
-
结构初始化(Structure Initialization)
- 以往的方法采用 基于距离的复杂初始化方法,而我们的方法 更简单高效:在框架区前后残基之间进行线性插值
Atom-level Encoding
节点特征(Node Features):
- 在 原子图(Atom Graph) 中,每个原子作为一个节点。
- 其特征采用 独热编码(One-hot Encoding),表示该原子的类型。
边特征(Edge Features):
- 原子之间的边特征通过 径向基函数(Radial Basis Function, RBF) 计算距离,公式如下:$f(B_p^i, G_e^j) = \text{RBF}(| z(B_p^i, k) - z(G_e^j, l) |)$
- 其中:$B_p^i, G_e^j$ 分别表示抗体和抗原的原子。$z(B_p^i, k)$ 和 $z(G_e^j, l)$ 分别表示原子的 3D 坐标。RBF 计算两个原子之间的距离
- 使用 MPN 计算 原子级特征向量
Residue-level Encoding
节点特征(Node Features):
-
在 残基图(Residue Graph) 中,每个残基作为一个节点。
-
残基的坐标采用 Cα 原子的 3D 坐标 作为位置信息。
-
每个残基的氨基酸特征 $f(B_p^i)$
由以下信息预定义:二面角(Dihedral Angles),极性(Polarity),疏水性(Hydropathy)
层次化特征融合(Hierarchical Feature Fusion):
-
采用 原子级特征求和 的方式,将原子级信息汇总到残基级别,计算如下:

-
其中:
- ⊕ 表示 拼接(concatenation) 操作。
- $f(B_p^i)$ 和 $f(G_e^j)$ 分别是抗体和抗原的 初始残基特征。
- $\sum_k h(B_p^i, k)$ 是 所有原子级特征的求和。
边特征(Edge Features):
- 残基图的边特征 $f(B_p^i, B_p^j)$ 包含:残基之间的欧几里得距离,方向信息,旋转方向
- 例如,边特征 $f(B_p^i, B_p^j)$ 计算 两个抗体残基 $B_p^i, B_p^j$ 之间的几何关系
残基级消息传递(Residue-level Message Passing):
- 在获得残基的 节点特征 和 边特征 后,使用 MPN 进行消息传递,学习 抗原表位(epitope)和抗体结合区(paratope) 的残基级特征。
3.2.2 Decoding
在序列和结构的预测过程中,我们采用了一种 一次性(One-shot)生成策略,不同于以往的 自回归(Autoregressive) 方式:
- 传统自回归方法的缺点:
- 生成效率低下:需要逐步预测每个氨基酸,计算量大。
- 误差累积问题:早期预测错误会 传播到后续步骤,导致最终结果偏差较大。
- 本文采用一次性生成策略:所有 CDR 氨基酸同时生成,减少计算开销。结构预测同时进行,避免误差传播问题。生成后进行 T 轮迭代优化,类似于 AlphaFold2。
序列生成(Sequence Generation)
-
采用 多分类交叉熵(Cross-entropy) 进行氨基酸预测:
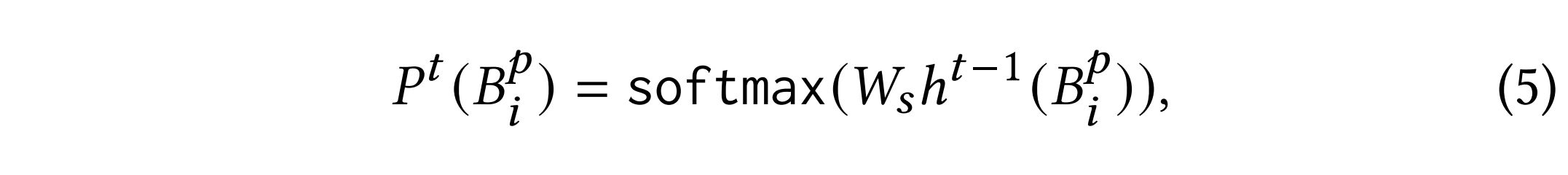
-
在前 T-1 轮优化中,采用 soft 预测分布,最后一轮进行 hard 采样,决定最终氨基酸类型。
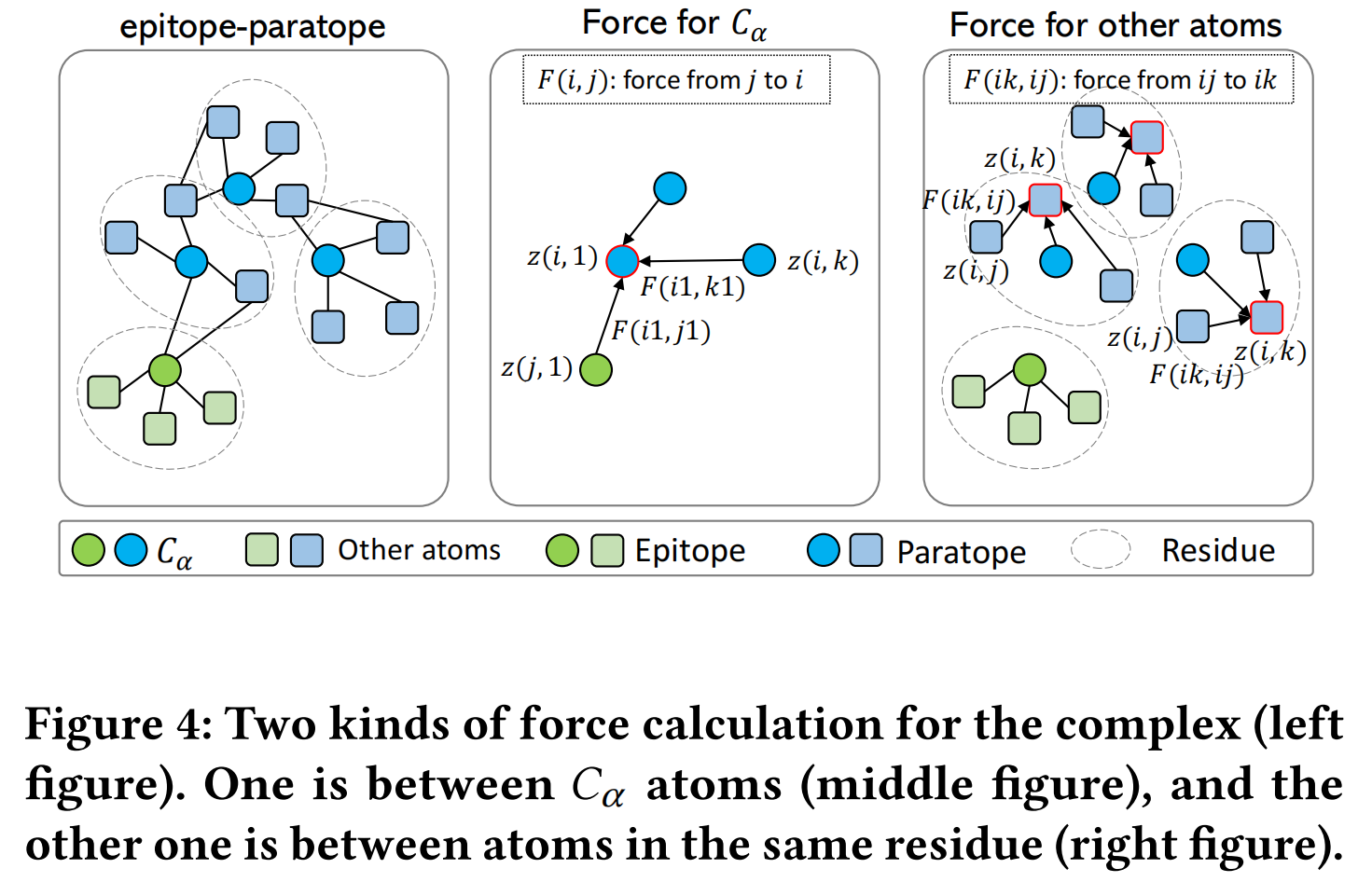
结构预测(Structure Decoding)
-
采用 力驱动(Force-based) 方法,而非直接坐标预测,以保持 旋转和平移等变性(Equivariance)。
-
计算 Cα 原子之间的相互作用力,更新主链坐标:
$F(i, j) = g(h(B_p^i), h(B_p^j)) \cdot (z(B_p^i) - z(B_p^j))$
其中,$g$ 为 前馈神经网络(FFN),$z(B_p^i)$ 为 第 i 个 CDR 残基的坐标。
-
更新规则:
$$
z_t(B_p^i) = z_{t-1}(B_p^i) + \frac{1}{n} \sum_{j \neq i} F_t(i, j) + \frac{1}{m} \sum_k F_t(i, k)
$$
其中,n 和 m 分别表示 相邻 Cα 原子和其他原子的数量。
3.4 Incorporating Pre-training
在抗体设计任务中,CDR 片段的序列预测需要良好的初始化,而随机初始化往往会导致不稳定的生成。因此,利用 AbBERT 预训练模型 提供的 软概率分布(soft prediction distribution) 来初始化 CDR 残基的特征。
除了初始化,我们进一步 在整个抗体设计过程中 充分利用 AbBERT 的能力,而不仅仅是在最初的输入阶段。为此,我们采用 多层特征融合策略,将 AbBERT 的表征融入抗体 GNN 设计模型:
-
融合方式:
-
在 H_seq(序列编码 GNN) 和 H_str(结构编码 GNN) 计算过程中,我们引入 AbBERT 的中间表示,以增强对 CDR 序列特征的建模能力。
-
具体而言,我们将 AbBERT 的上下文表征 作为额外输入:
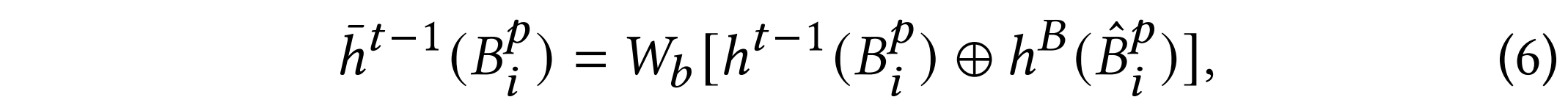
-
前缀微调(Prefix-tuning):
- 传统的微调方法会导致 预训练模型遗忘原始知识,为避免这种情况,我们使用 前缀微调(Prefix-tuning):
- 在 不改变 AbBERT 参数 的情况下,我们仅训练 一个额外的前缀向量,用于 适配抗体设计任务。
- 这样可以 保留 AbBERT 的知识,同时让其适应新的抗体序列生成任务。
3.5 Summary and Algorithm

- 输入数据:抗原表位(Epitope),抗体框架区(Framework Region),初始 CDR 片段(CDR Region)
- 初始化(Initialization):采用 AbBERT 生成 soft 预测分布 初始化 CDR 序列特征。采用 线性插值方法 初始化 CDR 结构坐标。
- 序列-结构联合优化(Sequence-Structure Co-design):
- 使用 H_seq GNN 预测 CDR 序列(基于 softmax 预测分布)。
- 使用 H_str GNN 预测 CDR 结构(基于力驱动的坐标更新)。
- 进行 T 轮迭代优化,逐步调整 CDR 结构,使其更符合物理稳定性和抗原结合特性。
- 最终输出:最优 CDR 序列,优化后的 CDR 结构
4 Experiments
4.1 Overview Settings
(1) 序列与结构预测(Sequence and Structure Prediction)
(2) 抗原结合 CDR-H3 设计(Antigen-binding CDR-H3 Design)
(3) 针对 SARS-CoV-2 的抗体优化(Antibody Optimization for SARS-CoV-2)
基线模型(Baseline Models)
为了公平比较,我们选择了以下基线方法:
- RAbD(Rosetta Antibody Design):基于物理建模的方法,用于序列生成和能量最小化。
- LSTM-based 方法:只针对序列生成,不考虑结构信息。
- AR-GNN(Autoregressive Graph Neural Network):一种图神经网络方法,仅对抗体进行编码,并以自回归方式逐步预测氨基酸。
- RefineGNN:与 AR-GNN 类似,但在每两个氨基酸生成之间进行结构调整,同时对 CDR 和框架区域采用不同分辨率编码,以更好地关注 CDR。
- HSRN(Hierarchical Structure Refinement Network):采用 层次化等变消息传递神经网络(Hierarchical Equivariant Message Passing Neural Networks) 进行抗原-抗体相互作用建模,同时使用 自回归解码和迭代优化 进行抗体设计。
- MEAN:引入 内部(internal)和外部(external)消息传递机制,用于分别编码上下文和表面信息,同时考虑轻链(Light Chain)、重链(Heavy Chain)和抗原(Antigen)。
模型设置(Model Settings)
- 预训练的 AbBERT(Antibody BERT):12 层 Transformer(BERT-base 结构),隐藏层维度:768,注意力头数:12,前缀微调(Prefix-tuning):添加 5 个软 token
- GNN 结构(H_seq 和 H_str)
- 使用 9 近邻(9-nearest neighbors)构建图边
- 每个消息传递网络(MPN)包含 4 层,隐藏维度为 256
- 框架区域(Framework)序列降维处理,每 8 个残基为 1 个 block
- 迭代优化步数 T=5T = 5T=5
预训练(Pre-training)
- 数据来源:Observed Antibody Space(OAS)数据库,包含 超过 10 亿条抗体序列,涵盖 不同免疫状态、生物体和个体。
- 数据处理
- 仅使用 抗体重链(Heavy Chain)序列 进行预训练。
- 经过 序列过滤、聚类(MMseqs2,最小序列同一性设定为 0.7) 以减少冗余数据。
- 最终 随机抽取 5000 万条抗体序列 用于预训练。
- 训练设置:16 块 32GB V100 GPU,Batch Size:每块 GPU 256,梯度累积步数:4,学习率:0.0003
4.2 Task 1: Sequence and Structure Prediction
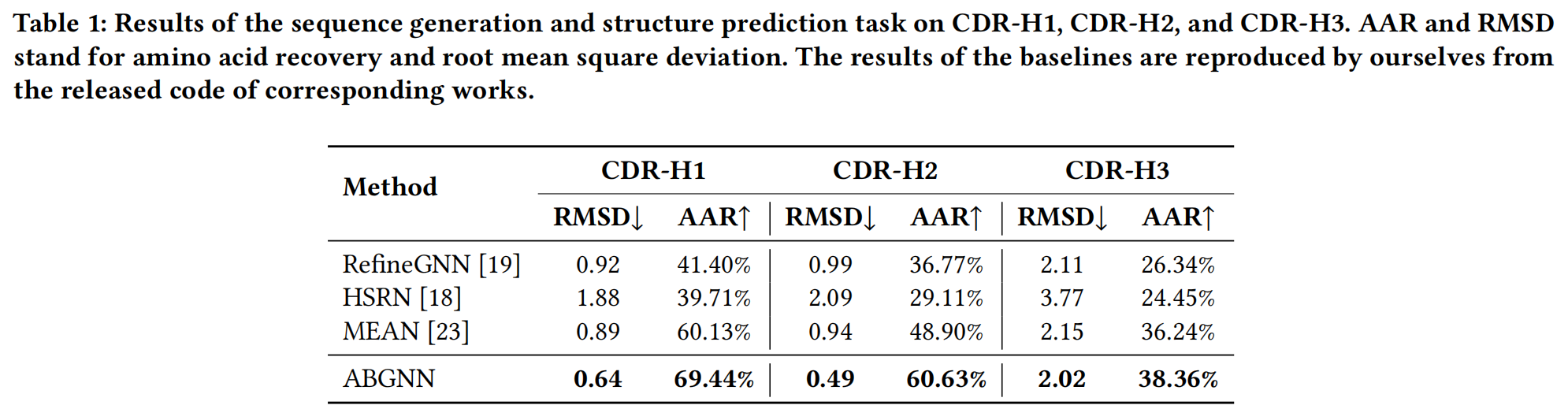
本任务评估模型在 CDR 设计任务 中 同时预测序列和结构 的能力。实验基于 抗体-抗原复合物 数据,要求模型生成 完整的 CDR 片段,包括其 氨基酸序列 和 三维结构。
- 数据集:从 SAbDab 数据库,在CDR区域上使用MMseqs2,两个序列相似度>40%视为同一个cluster,随机划分8:1:1训练验证测试,分别对CDR-H1,CHR-H2和CDR-H3得到765,1093,1659个cluster。
- 测试数据:采用 不同的抗原 进行评估,以测试泛化能力。重点关注 CDR-H3,因其在 抗原结合和抗体功能 中最具变异性和决定性。
4.3 Task 2: Antigen-binding CDR-H3 Design
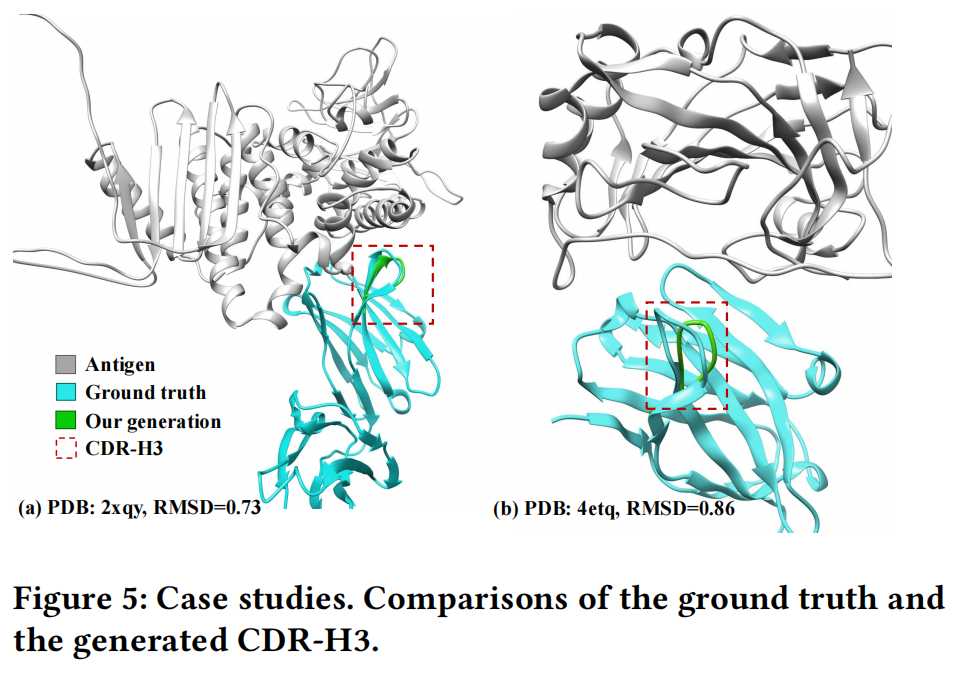
数据(Data)
本任务比一般的 CDR 设计更具挑战性,因为其目标是 生成特定抗原结合的 CDR-H3,因此 抗原被视为条件输入。在此任务中,仅生成 CDR-H3,不涉及其他 CDR 片段。
- 测试数据:来自 Adolf-Bryfogle et al. [1] 的数据集,其中包含 60 种不同抗原类型的抗体-抗原复合物。
- 训练数据:使用 SAbDab 数据库,但 仅使用抗体-抗原复合物数据,不包含单独的抗体结构。
- 数据处理:采用 Jin et al. [18] 处理后的数据集,其中:
- 训练集和验证集共包含 2777 和 169 个抗体-抗原复合物。
实验设置(Inference Procedure)
- 候选 CDR-H3 生成:在推理阶段,本文遵循 Jin et al. [18] 的方法,为 每个抗体生成 10,000 条候选 CDR-H3 序列。
- 筛选策略:从 10,000 个候选序列中,选择 困惑度(Perplexity)最低的前 100 个,用于计算 AAR(Amino Acid Recovery,氨基酸恢复率) 和 RMSD(Root Mean Square Deviation,均方根偏差)。
4.4 Task 3: SARS-CoV-2 Antibody Optimization
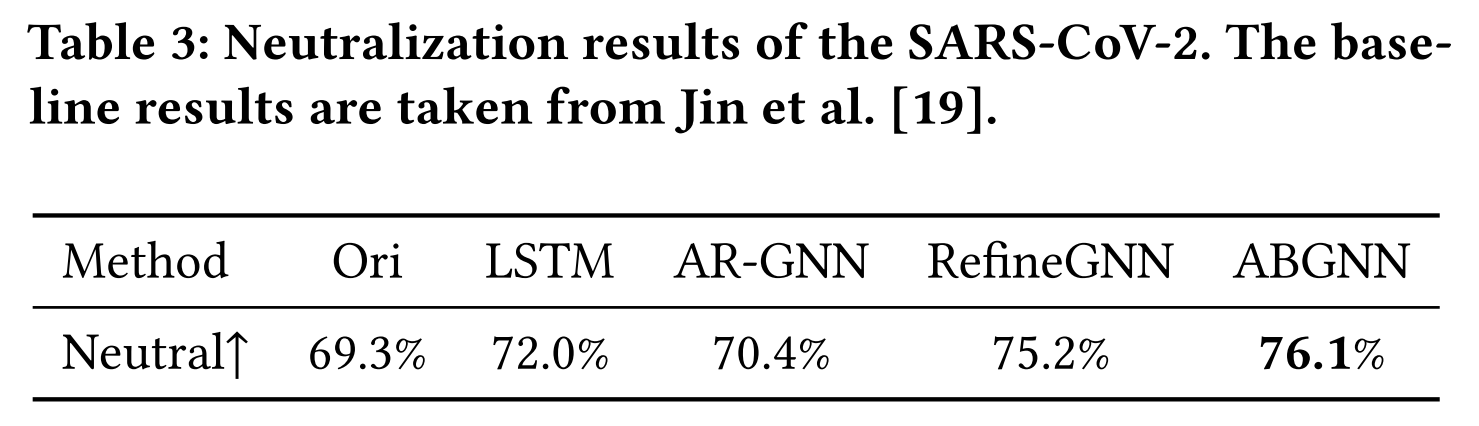
数据(Data)
本任务的目标是 优化 SARS-CoV-2 结合抗体的 CDR-H3。实验数据来源于 Coronavirus Antibody Database [41],其中包含 2411 个与 SARS-CoV-1 或 SARS-CoV-2 结合的抗体。
- 数据集划分:CDR-H3 片段按 8:1:1 的比例划分为 训练集、验证集和测试集。
- 数据预处理:采用 Jin et al. [19] 预处理后的数据集,其中包含 详细的 CDR 片段筛选和序列生成约束(具体处理方式可参考原论文)。
- 优化方法:
- 训练方法遵循 Jin et al. [19],使用 ITA 算法(Iterative Targeted Antibody Optimization, ITA) 对 生成的抗体进行微调(finetuning)。
- CDR 片段的生成方式和约束条件与 Jin et al. [19] 保持一致,以确保对比公平性。
实验结果(Result)
- 由于缺乏湿实验支持,本研究采用 **中和效应预测器 进行评估。
- 评测方式:采用 二分类(binary classification) 任务,使用 Jin et al. [19] 预训练的预测模型 评估生成抗体的 中和能力(neutralization effect)。
- 实验结论:
- 本文方法相比于现有 SOTA 方法,能够优化抗体,使其在预测的中和效应上表现更优。
- 结果显示,模型能够成功重新设计抗体,提高其潜在的中和效果,进一步证明了 AbBERT 在抗体优化任务中的有效性。
5 Study
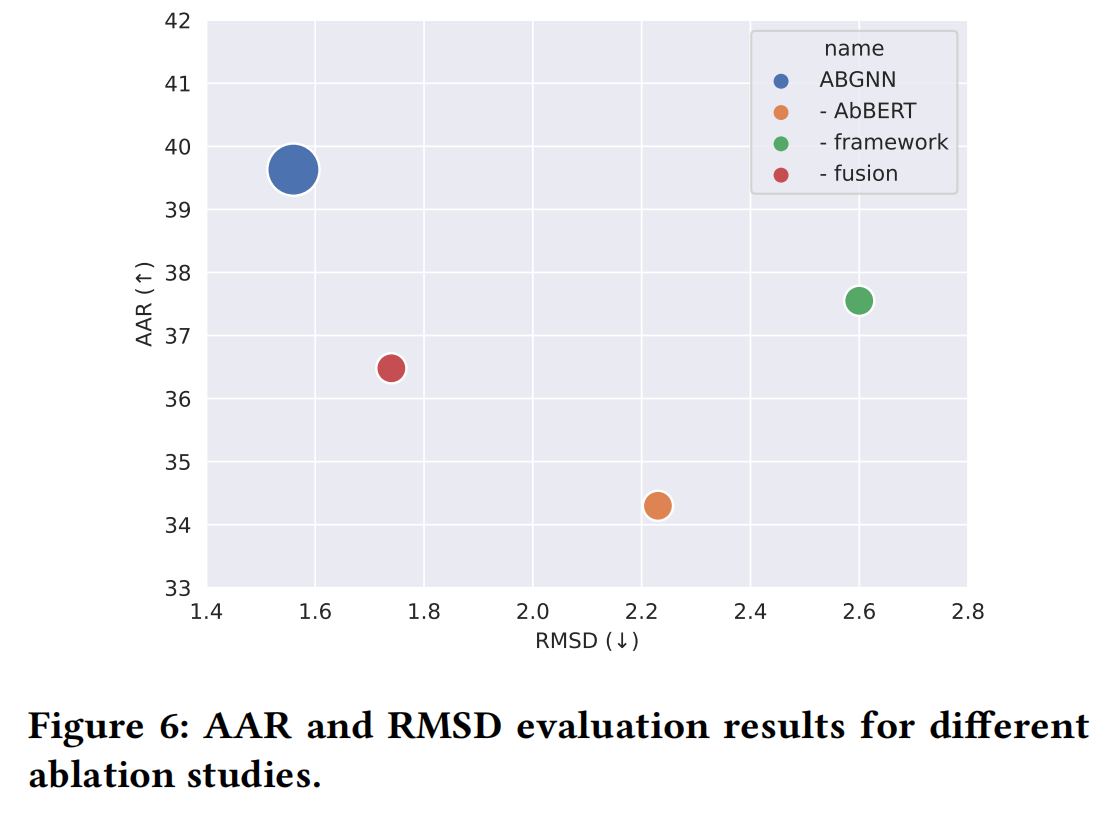
5.1 Ablation Study
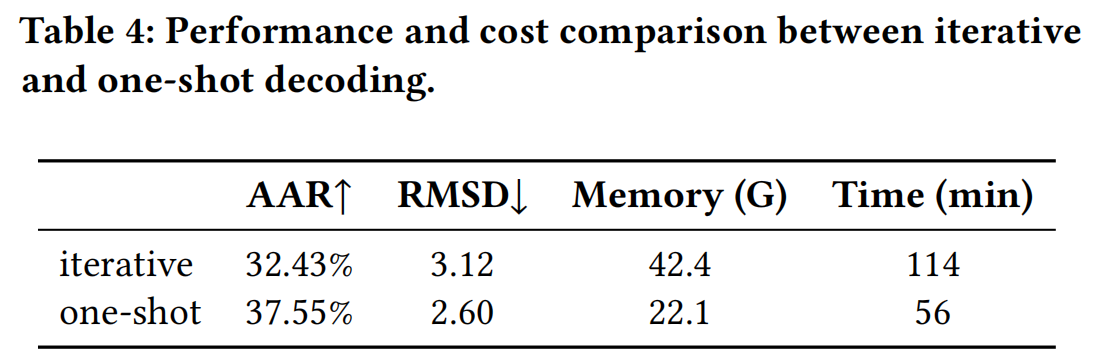
5.2 Effect of Decoding ways

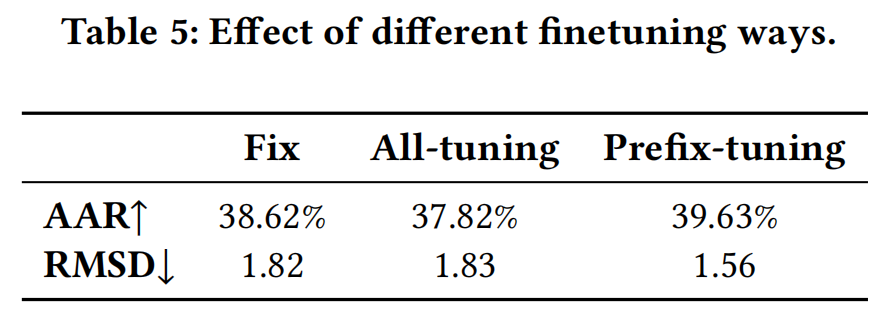
5.3 AbBERT Incorporating Ways
5.4 Effect of Pre-training



